Docosahexaenoic acid (DHA, 22:6n-3) and ecosapentaenoic acid (20:5n-3) have attracted increasing attention since the first epidemiological report on the importance of n-3 essential fatty acids. It is thought that DHA has important functions in brain and retinal tissues. Thraustochytrids, a group of marine protists, are capable of heterotrophic growth, and are potential omega-3 producers for industrial use, especially the members of the
Docosahexaenoic acid (DHA)는 eicosapentaenoic acid (EPA), α-linolenic acid와 함께 오메가-3계 지방산에 속하며, serum triacylglycerides 및 LDL-cholesterol의 함량을 낮추고 HDL-cholesterol 수치를 높임으로써 동맥경화 및 혈전증 등과 같은 심혈관계 질환의 예방에 효과가 있는 것으로 보고되고 있다 (Haglund et al., 1991; Horrocks and Yeo, 1999; Lee and Kim, 2001; Ohr, 2003). 또한 두뇌, 신경계 및 안구조직의 필수지방산으로 특히, 유아의 시력 및 운동능력 개발 등에 중요한 요소이며, 뇌의 구조적 지질에 가장 풍부한 구성요소 중의 하나이다(Kanazawa et al., 1982; Drats and Deose, 1986; Lee et al., 2003).
오메가-3계 고도불포화지방산의 주요 공급원은 어유(Fish oil)이며, 어유에는 7-17%의 DHA가 포함되어있다(Kremer et al., 1987; Stenson et al., 1991). 그러나 식품첨가제로 사용되는 어유는 특유의 비린내와 맛 그리고 산화에 의한 안전성 문제등으로 그 사용이 제한적이다(Tanaka et al.,1992; Carlson, 1996; Gill and Valiverty, 1997). 고도불포화지방산은 해조류나 곰팡이 또는 박테리아가 생합성하여 체내에 에너지원으로 저장하거나 몸체의 구성요소로 사용한다. 해산어류는 오메가-3 고도불포화지방산을 직접 생합성할 수 없기 때문에 체내에서 필요한 고도불포화지방산은 미생물의 섭취를 통하여 충족시키고 있다(Castell et al., 1972; Bremer, 1995; Bremer and Talbot, 1995; Tanaka et al.,1999 ). 따라서 어유를 구성하고 있는 지방산의 양과 조성은 해양미생물 즉, 먹이사슬의 일차생물 오메가-3 고도불포화지방산 생합성 형태에 따라서 결정된다고 할 수 있다(Miller and Jones, 1983; Braden and Carroll, 1986).
오메가-3계 고도불포화지방산은 고등어, 꽁치 등과 같은 등푸른 생선으로부터 추출하여 산업적으로 이용하고 있으나, 어유 특유의 비린내 및 특정 지방산의 비율조절의 어려움 등과 같은 단점을 가지고 있다(Park et al., 1994; Lee et al., 1997; Cho et al., 2004). DHA 같은 고도불포화지방산은 인위적인 화학합성 과정을 통한 생산이 매우 어렵기 때문에 천연물질로부터 획득해야 하는데, 현재 공업적으로는 이들 지방산을 어유에서 추출하여 분리 정제 후 사용하고 있다. 생산되는 어유의 대부분은 수소첨가반응에 의해 마가린과 소프트닝으로 제조되는데, 이 과정에서 이중결합의 위치가 바뀌고 cis형에서 trans형으로 입체 이성화가 일어나는 등 대부분의 오메가-3계 지방산은 파괴 또는 변형된다(Singh and Ward, 1997). 이러한 천연물로부터 추출공정을 통한 고도불포화지방산 생산시 발생하는 여러 한계점을 극복하고자 고도불포화지방산 생산능력이 보고된 미생물을 통하여 경제적으로 유용한 고도불포화지방산을 생산하려는 생물학적 연구가 많이 진행 중이다(Yazawa et al., 1998; Tonon et al., 2002; Owen and Ajay, 2005). 특히, 해양식물성 플랑크톤과
DHA의 주요 공급원으로서 사용되는 어유는 바다로부터 공급되는 양은 한정되어 있고, 수요는 급격히 증가되고 있는 추세이다(Stenson et al., 1991). 이러한 점을 착안하여 이번 연구에서는 DHA를 다량 함유하는 해양미생물(Thraustochytrid 科)을 분리 동정하여 DHA의 새로운 공급원으로서 이용하고자 한다.
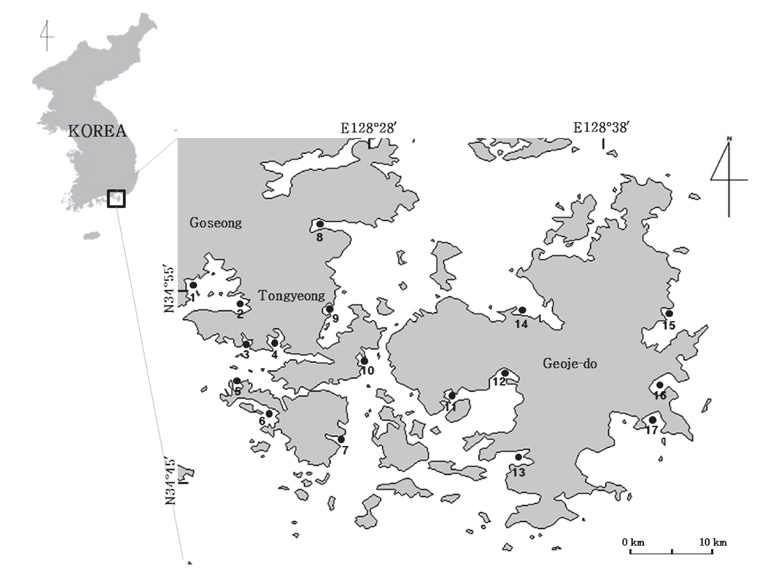
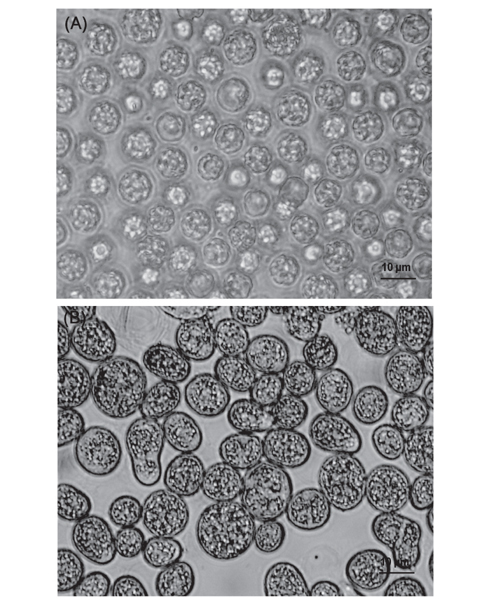
DHA를 다량 함유하는 해양미생물의 분리를 위한 시료는 2006년 4월부터 10월까지 경상남도 통영, 고성 및 거제 일대 해안으로부터 3가지 방법으로 시료를 채취하였다(Fig. 1). 첫째, 부유생물 시료는 3 μm 플랑크톤네트를 이용하여 해수를 여과시킨 여과잔여물을 이용하였고, 둘째, 저질층 시료는 유기퇴적물이 비교적 풍부한 곳을 선정하여 직접 채니하였다. 셋째, 조류(algae)는 해안가 연안에 부유성 조류를 시료로 채집하였다. 채취한 각각의 시료는 10℃ 로 유지되는 Ice Box에 넣어서 실험실로 운반하여 사용하였다.
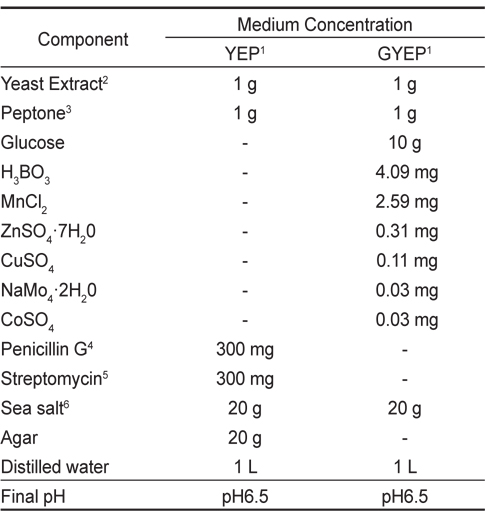
유용미생물의 분리는 Table 1에 나타낸 바와 같이 yeast extract (Becton, Dickinson and Company sperks, MD, USA) 1.0 g/L, peptone (Acumedia Manufacturers, Inc. Lansing, MI, USA) 1.0 g/L를 주요 성분으로 하는YEP 배지를 사용하였으며(Wong et al., 2005), 이 때 세균의 증식억제를 위해 penicillin-G (P3032, Sigma, USA)와 streptomycin sulfate (S-9137, Sigma, USA)를 각각 300 mg씩 별도로 멸균 처리된 증류수에 용해시켜 0.22 μm 여과지(Millipore)로 여과하여 분리용 평판배지에 첨가하였다. 분리용 YEP 배지를 이용하여 채취된 각 시료들을 도말하여 25℃ 세포배양기(BOD Incubator DS-11B, Dasol Scientific Co., Ltd.)에서 72시간 배양하였다. 유용미생물은 평판배지에 형성된 집락(colony)을 현미경을 이용하여 Thraustochytrid 과(科)와 유사형태를 가진 해양미생물을 선발하였다. 선발된 균체의 배양은 glucose 10 g/L, yeast extract 1.0 g/L 그리고 peptone 1.0 g/L 를 주요 성분으로 하는 GYEP 배지를 부분적으로 보완하여(Table 1) pH 6.5로 조정된 평판배지에서 25℃에서 72시간 배양한 후, 이들 균주를 동일한 액상배지로 25℃, 120 rpm의 진탕배양기(KSI-200L, KOENCON)에서 72시간 배양하였다. 균체의 지방산 분석 시료는 균체 배양액을 원심분리기(HA-1000-3, Hanil science Industrial Co., Ltd.)로 5,000 rpm에서 10분간 원심분리한 후, 배양액은 버리고 침전된 세포를 회수하여 동결 건조하였다.
[Table 1.] Composition of YEP1 and GYEP1 medium
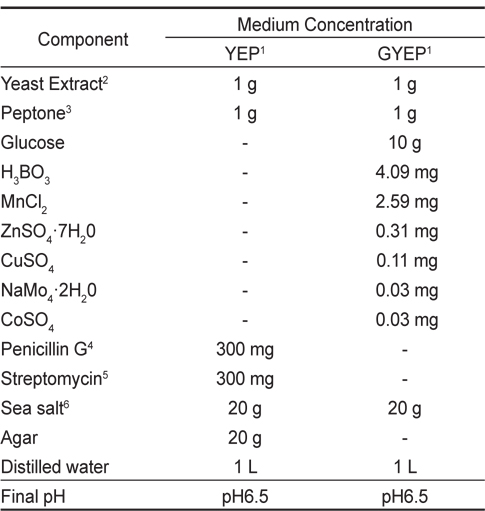
Composition of YEP1 and GYEP1 medium
총 지질 추출은 Bligh and Dyer (1959) 방법에 준하였다. 균체 중의 지질 추출 시료를 비커에 5 g 취하여 세포분쇄기(Homogenizer AM-12, Nihonseiki Kaisha Ltd. Tokyo, Japan)에서 15,000 rpm으로 5분간 분쇄한 후, chloroform : methanol (2:1, v/v) 혼합 용매를 시료의 2 배량 넣어 하루 동안 방치하였다. 다음날 chloroform 층만을 분리하기 위하여 둥근 플라스크 위에 깔때기를 놓고, 여과지를 깔아 그 위에 Na2SO4를 넣어 서서히 chloroform층만 흘러내리게 하였다. 분리된 chloroform 층은 진공회전농축기(Rotavapor R-114, BUCHI)를 사용하여 40℃ 이하에서 용매를 완전히 증발시킨 후, 추출된 총 지질의 무게를 측정하였다. 모든 작업은 질소 기류 하에서 행하였다.
추출된 지질 50 mg 을 cap tube에 취하고, 0.5 N NaOH-methanol 용액 1.5 mL 가하여 질소를 충진한 다음, 100℃에서 8분간 가열하여 검화하였다. 방냉 후 12% BF3-methanol 2 mL 가한 후, tube 뚜껑을 닫고, 100℃에서 11분간 가열하여 methyl화 하였다. 약 30℃로 냉각한 후, iso-octane 1 mL 첨가하고 30초간 vortex mixer로 혼합하였다. 즉시 3 mL의 포화식염수를 가한 다음 흔들어 방치하여 iso-octane층이 분리되도록 하였다. iso-octane층을 시료 병에 옮긴 후, 다시 iso-octane 1 mL 첨가한 다음 흔들어 재추출하여 시료 병에 모으고 이를 지방산 methylester 시료로 하였다.
지방산 분석에 사용하는 GLC는 OmegawaxTM-320 fusedsilica capillary column (30 m×0.32 mm×0.25 μm, i.d., Supelco Co., Bellefonte, PA, USA)를 장착한 Clarus 600 (Perkin Elmer Co. Ltd., USA)을 이용하였다. 분석은 column 조건의 경우 185℃에서 8분간 유지하고 3℃/min씩 230℃까지 상승시킨 후, 10분간 유지하였다. 이 때 주입기는 250℃, 검출기는 270℃ 그리고 carrier gas는 헬륨(He)을 사용하였다. 지방산의 분석은 동일조건에서 분석한 표준품의 ECL과 비교하여 동정하였고, 지방산 표준품은 14:0, 16:0, 18:1, 18:2, 18:3, 20:0, 22:1, 24:0 (Sigma Chemical Co., St. Louis, MO, USA)과 GC-MS로 동정된 menhaden oil을 사용하였다.
분리된 해양미생물의 18S ribosomal DNA sequencing은 Wizard genomic DNA purification kit (Promega, USA)를 이용해 chromosomal DNA를 분리한 후, 18S rRNA sequencing에 사용하는 universal primer인 NS1 (5'-GTAGTCATATGCTTGTCTC-3')와 NS8 (5'-TCCGCAGGTTCACCTACGGA-3') primer를 사용하여 PCR 증폭하였다. 증폭된 PCR 산물은 전기영동장치(Wizard SV Gel)와 PCR clean-up system (Promega, USA)을 이용하여 정제하였다. 정제된 PCR 산물은 ABI PRISM 3700 DNA Analyzer (Applied Biosystem, Foster, CA, USA)를 이용하여 염기서열을 분석하였다. 그 결과는 NCBI의 BLASTN 프로그램을 이용하여 GENEBANK의 ribosomal DNA sequence와 비교하였으며, sequence의 상동성은 Clustal X와 Mega 2 program을 이용하여 비교 분석하였다(Thompson et al., 1994).
모든 자료는 SPSS (12.0) 프로그램을 이용하여 분산분석 (one-way ANOVA)을 실시하여 Duncan's multiple range test (Duncan, 1955)로 평균간의 유의성(
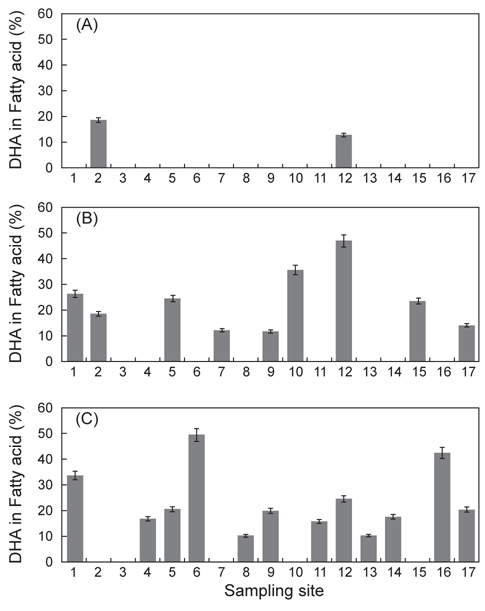
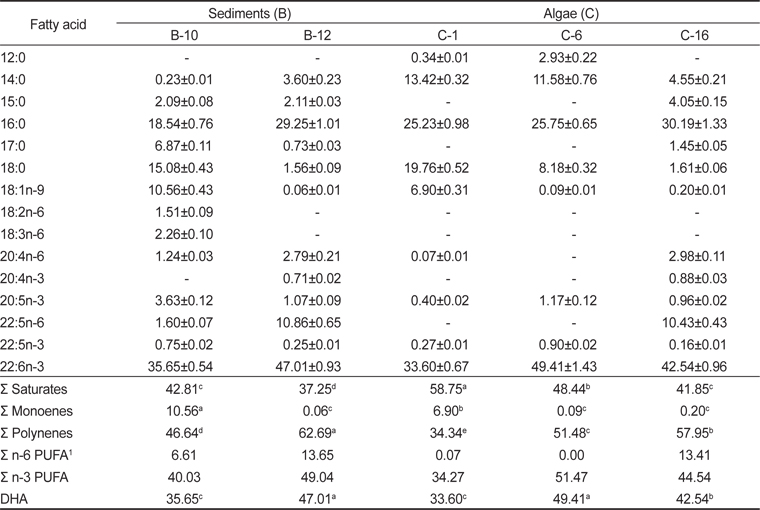
Thraustochytrid 科로 추정되는 해양미생물은 Fig. 2에서 보는 바와 같이 여과해수 잔여물에서 2종, 유기퇴적물에서 9종 그리고 조류에서 12종으로 총 23종을 분리하였다. 여과해수 잔여물에서는 A-2, A-12 지점에서 2개의 균주를 분리하였으며 DHA는 각각 18.64% 와 12.75%로 나타났다. 유기퇴적물에서 채취한 시료는 B-1, B-2, B-5, B-7, B-9, B-10, B-12, B-15 그리고 B-17지점에서 9개의 균주를 분리하였으며 DHA는 각각 26.40, 15.75, 24.59, 12.26, 11.85, 35.65, 47.01, 23.53 그리고 14.14% 로 B-12지점에서 분리한 균주의 DHA가 47.01%로 가장 높게 나타났으며(
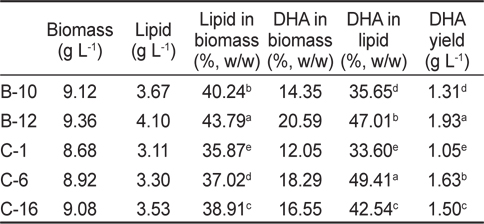
[Table 2.] Biomass, lipid and DHA production of isolated species culture
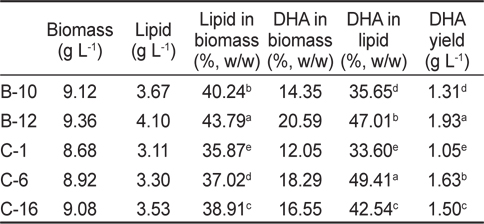
Biomass, lipid and DHA production of isolated species culture
분리 선발된 균주 5종의 총 지질 함량에 관한 결과는 Table 2에 나타낸 바와 같이 B-10, B-12, C-1, C-6 그리고 C-16지점에서 분리된 균주에 따른 biomass는 각각 9.12, 9.36, 8.68, 8.92 그리고 9.08 g/L로 B-12지점에서 분리된 균주가 9.36 g/L로 가장 높게 나타났으며(
[Table 3.] Fatty acid compositions of isolated species cultured for 3 days (% of total fatty acids)
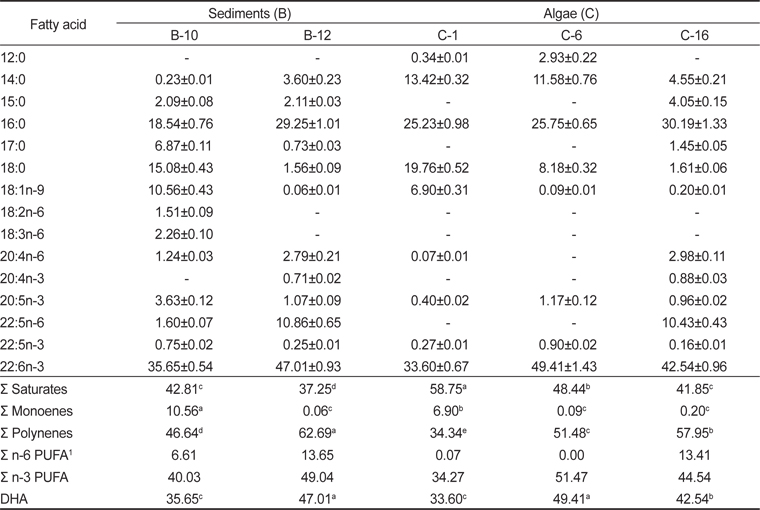
Fatty acid compositions of isolated species cultured for 3 days (% of total fatty acids)
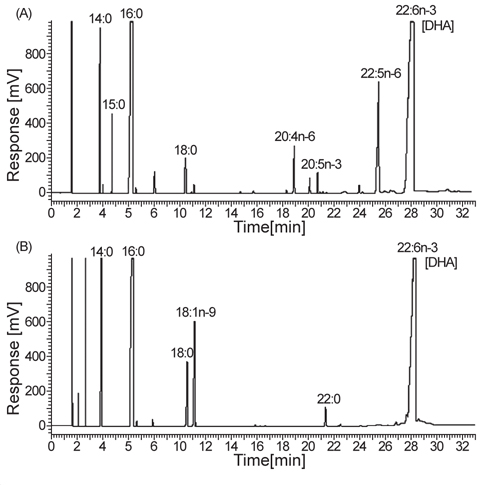
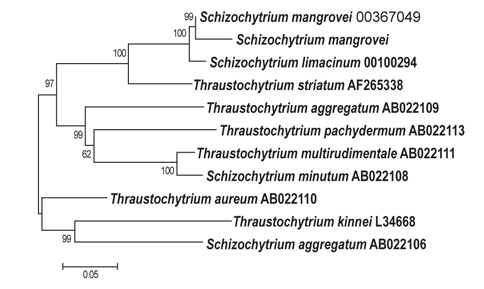
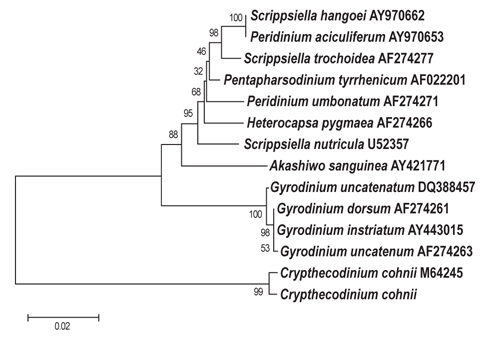
B-12그리고 C-6 지점에서 분리 선발된 2종에 대하여 18S rDNA sequencing 분석을 실시한 결과는 Figs. 3, 4에 나타난 바와 같다. B-12지점에서 분리된 균주는
오메가-3계 고도불포화지방산을 생산하는 미생물에 대한 관심은 EPA나 DHA의 생합성 유전자들이 분석되고 동정됨으로서 이루어졌다(Yazawa, 1996; Tanaka et al., 1999). 고도불포화지방산 생합성에 관여하는 유전자에 대한 연구는 EPA를 생산하는
최근 미생물에 의한 지질 생산에 관하여 다시 관심을 갖게 되었는데 이는 실질적인 기술에 의하여 미생물에 의한 지질 생산이 가능한 것으로 보고하고 있다(Wu et al., 2005; Zhu et al., 2007). 인지질과 sterol 지방산 함량과 조성은 여러 가지 환경조건 즉, 온도, pH, 배지의 조성의 영향을 받는데 식물 조직세포에서 고도불포화지방산은 12℃에서 80%가 형성되나 20℃와 30℃에서는 각각 51%와 30%의 불포화지방산이 합성된다고 보고하고 있다(Browse and Slack, 1993). 온도가 낮으면 불포화지방산의 양이 많아지는 반면 배양 온도가 높으면 포화지방산의 양이 많아진다고 보고되고 있다(Knivett and Cullen, 1965). 이전에 보고된 Thraustochytrids의 종에 따른 적정 배양 온도는 다양하게 나타났는데
이러한 미생물에 의한 지질 생산의 장점은 태양에너지가 필요하지 않고, 날씨에 관계없이 연중 생산이 가능하며, 배양조건에 따라 생산되는 지질의 조성을 조절 할 수 있다는 것이다(Hulan et al., 1989; Ward, 1995; Abril et al., 2003). 유지(油脂)미생물의 지질축척은 배지의 영양소, 대개는 질소가 고갈되었을 때 시작되며, DHA 생산에 미치는 glucose 영향을 검토한 결과 glucose 농도가 낮으면 지방산과 DHA 생성이 촉진된다고 한다(Honda et al., 1998). 또한 지질의 농도는 대수증식기에는 감소되었지만, 배지성분이 제한되어 생육이 지연되면 지질의 생산은 증가되고, DHA 농도는 감소된다는 보고도 있다(Yazawa, 1996; Honda et al., 1998). 따라서 해양미생물의 종에 따른 고도불포화지방산 생성 능력이 우수한 종을 찾아내는 것도 중요하지만, 배양 환경에 따른 물리적인 조건과 영양성분에 따른 생리적인 조건을 적절하게 조절하는 것이 무엇보다 중요하며 이러한 점을 고려하여 더 많은 연구가 이루어진다면 더욱 우수한 양질의 지질과 보다 많은 양의 DHA를 생산할 수 있을 것으로 사료된다.