대용량 문서에서 정보를 추출하는 작업은 정보검색 분야 뿐 아니라 질의응답과 요약분야에서 매우 유용하다. 정보추출은 비정형 데이터로부터 정형화된 정보를 자동으로 추출하는 작업으로써, 개체명 인식, 전문용어 인식, 대용어 참조해소, 관계 추출 작업 등으로 구성된다. 이들 각각의 기술들은 지금까지 독립적으로 연구되어왔기 때문에, 구조적으로 상이한 입출력 방식을 가지며, 하부모듈인 언어처리 엔진들은 특성에 따라 개발 환경이 매우 다양하여 통합 활용이 어렵다. 과학기술문헌의 경우 개체명과 전문용어가 혼재되어 있는 형태로 구성된 문서가 많으므로, 기존의 연구결과를 이용하여 접근한다면 결과물 통합과정의 불편함과 처리속도에 많은 제약이 따른다. 본 연구에서는 생의학 분야 과학기술 문헌을 분석하여 전문용어 및 개체명 등을 통합 추출할 수 있는 기반 프레임워크를 개발한다. 이를 위하여, 문장자동분리, 품사태깅, 기저구인식 등과 같은 기반 언어 분석 모듈은 물론 이를 활용한 개체명 인식기, 전문용어 인식기를 개발하고 이들을 하나의 플랫폼으로 통합한 과학기술 핵심개체 인식 체계를 제안한다. 전체 플랫폼의 성능을 체계적으로 평가하기 위해서, KEEC 2009를 비롯한 다양한 말뭉치를 기반으로 세부 요수 모듈에 대한 성능 평가를 수행하였으며, 비교적 높은 수준의 성능을 확보 하였다. 본 논문에서 개발된 핵심개체자동인식 플랫폼은 정보검색, 질의응답, 문서색인, 사전구축 등 다양한 정보서비스 분야에 활용될 수 있다.
인터넷의 발달과 더불어 대용량 데이터를 실시간으로 처리하여 필요한 지식을 발견하기 위한 정보추출 기술들이 핵심적인 분야로 인식되고 있다. 정보추출은 크게 (1)개체명 인식 (named-entity recognition), (2)대용어 참조해소(coreference resolution), (3)관계 추출(relation extraction)의 세 가지 요소기술로 세분화 된다. 본 연구에서는 이 중 개체명 인식과 대용어 참조해소에 대한 부분을 다룬다.
개체명 인식은 문서내의 원소를 찾아서 기정의된 범주로 분류하는 작업으로, 특정 도메인에 따라 개체명 인식기(Named Entity Re-cognition System)가 개발된다. 개체명 인식은 인명, 지명, 기관명 같은 일반적인 범주와 더불어, 생의학 분야에서는 유전자와, 유전자와 관련된 산출물 등과 같이 분야특화된 범주가 사용된다. BBN1)은 29개의 범주와 64개의 하위범주를 제안하였고, Sekine은 200개의 하위범주를 갖는 확장계층도2)를 제안하였다.
본 연구에서는 일반적인 범주인 인명, 지명, 기관명에 대한 부분만 개체명으로 정의하고, 생의학 분야에 특화된 개체명은 기술용어 및 분야특화명으로 구분되는 전문용어로 분류한다.
대용어 참조해소는 한 명사구와 명사구를 참조하는 개체로 해소하는 작업으로 자연어의 하이퍼링크라 볼 수 있으며, 일반적으로 한번 출현한 단어의 반복을 피하기 위해 사용된다. 개체명과 전문용어가 인식된 후 이들 간의 관계 추출 작업을 수 행할 때 더 많은 자원을 확보하기 위해 대용어 참조해소는 중요한 작업이다. 본 연구에서는 대명사를 참조해소의 대상으로 한다.
현재까지 개체명 인식 분야는 신문기사 및 방송기사 등을 중심으로 연구되었고, 전문용어 인식 분야는 생의학 분야에서 유전자, 단백질과 관련된 용어들에 대하여 많은 연구가 행해졌다. 개체명 인식 기술과 전문용어 인식 기술이 서로 독립적인 영역에서 연구되었고, 전문용어 분야에서도 그 범위는 매우 제한적이다.
과학기술문헌의 경우 일반적인 개체명과 전문용어가 혼재되어 있는 형태로 구성되므로, 기존의 연구결과물을 이용하여 접근하기 위해서는 2단계로 작업을 수행해야 하기 때문에, 그 결과물을 통합하는 과정의 불편함이 있고, 처리속도에서 많은 제약이 따른다. 본 연구에서는 개체명과 전문용어를 통합하여 핵심개체로 정의하고, 한국과학기술정보연구원이 보유하고 있는 해외학술지 데이터베이스 중에서 생의학 분야 문헌들을 대상으로 개체명과 전문용어를 동시에 인식하고, 이를 참조하는 대용어에 대한 참조해소를 수행하는 통합된 플랫폼을 구현한다.
본 연구에서는 과학기술문헌에서 중요하게 인식되는 인명, 지명, 기관명의 일반적인 개체명과 분야에 특화된 전문용어를 통합하여 핵심개체로 지정하고 인식한다. 또한 핵심개체를 가리키는 대용대명사에 대한 참조해소를 수행한다.
현재까지 개체명과 전문용어 인식 그리고 대용어 참조해소에 관한 연구는 독립적으로 수행되어 왔기 때문에 핵심개체의 구성요소인 개체명과 전문용어에 대한 관련연구와 대용대명사 참조해소에 관한 연구를 각각 조사한다.
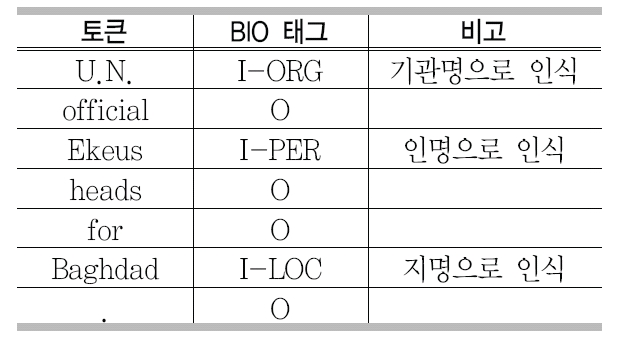
개체명은 고유명사(복합명사 포함)와 시간 등을 나타내는 수식 표현을 말한다 .아래는 개체명의 예이며 PER은 인명을 나타내고 LOC는 지명을, ORG는 기관명을 나타낸다.
[PER Wolff], currently a journalist in [LOC Argentina], played with [PER Del Bosque] in the final years of the seventies in [ORG Real Madrid].
개체명 인식은 정보 추출(Information Ex-traction)의 한 분야로써 문서내에서 개체명을 추출하고 기정의된 분야(인명, 지명, 기관명 등)로 분류하는 작업을 말한다. 개체명 인식에 관한 연구는 MUC-6(Message Understanding Conferences)3)에서 유래되어 최근에는 개체명을 약 200여 개로 나누어 질의응답 시스템의 문장 분석 에 널리 사용되고 있다.4) MUC-6에 참가한 많은 시스템은 특정 언어에 제한된 규칙과 자신만의 입출력 방법을 사용하여 다른 언어나 다른 영역에 쉽게 적용할 수 없었다.
MUC-6 이후 개체명에 대한 연구가 꾸준히 진행되어 CoNLL(Conference on Computational Natural Language Learning) 20025)와 20036)을통해서 많은 발전이 있었다. 이 대회에 참가한 대부분의 시스템은 기계학습 방법을 이용하였으며 영어의 경우에 약89%의 정확률을 보인다. 기계학습 방법에서는 주로 BIO 태그(B: 개체명의 시작, I: 개체명의 중간, O : 관계없음)를 이용하는데 <표 1>은 CoNLL에서 사용한 예이다.
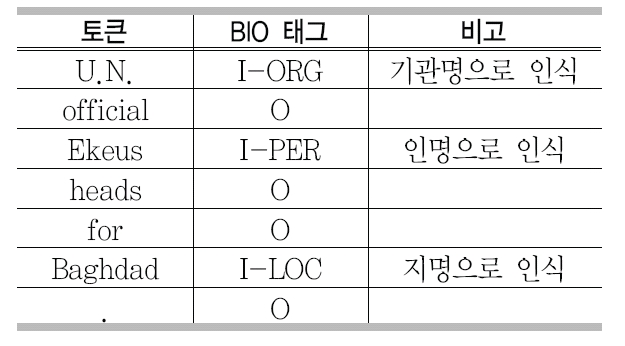
CoNLL에서 사용된 BIO 태그
Black과 Vasilakopolos(2002)는 90년대 초반 Eric Brill이 처음 소개한 변환기반 학습(Transformation-based learning)을 이용하였다. 변환기반 학습은 형태소 부착, 영어 전치사구 접속문제 해결, 기저구 인식, 철자 수정 등 자연어처리의 다양한 분야에 사용된 기계학습기법으로 첫 번째 단계에서 개체명을 인식하는 작업을 수행하고, 두 번째 단계에서 인식된 개체명 후보들에 대한 적합한 분류를 제공하는 방법이다.
Carreras, Marques, Padro(2002)는 CoNLL 2002에서 개체명 인식을 위해 잘 알려진 BIO태그 외에, 개체명의 시작과 종료 경계를 인식하기 위한 Open-Close&I와 Global Open-Close방식을 도입하였고, 개체명 분류를 위해 Ada-Boost 이진 분류 알고리즘을 사용하였다.
Collins(2002)는 개체명 경계인식을 위해 최대 엔트로피(Maximum Entropy, ME)태거를 이용하여 추천된 상위 N개의 후보에 대해 voted perceptron 알고리즘을 이용하여 가중치를 재부여하는 방법을 제안하였다 .학습 및 실험은 웹 데이터를 이용하였고 ME 태거의 85.3%의 F1-척도에 비해 87.9%의 F1-척도를 보여주었다.
Watanabe, Asahara, Matsumoto(2007)는 HTML로 구성된 위키피디아 문서들을 그래프 구조로 표현하였다. HTML에서 하이퍼링크를 개체명으로 취급하고 이를 그래프상의 노드로 표현하였고, CRFs(Conditional Random Fields)를 도입하여 그래프 구조상의 노드들을 분류하였다.
1) http://www.ldc.upenn.edu/Catalog/docs/LDC2005T33/BBN-Types-Subtypes.html 2) http://nlp.cs.nyu.edu/ene 3) http://www.cs.nyu.edu/cs/faculty/grishman/muc6.html 4) Extended Named Entity Hierarchy, http://nlp.cs.nyu.edu/ene/ 5) http://www.cnts.ua.ac.be/conll2002/ner/ 6) http://www.cnts.ua.ac.be/conll2003/ner/
전문용어는 초기에 용어사전을 이용해서 관리되고 보급되어 왔다. 그러나 과학기술의 급속한 발전으로 전문분야와 전문용어도 또한 급속히 생성되고 있어 용어사전만으로는 도저히 전문용어를 파악하여 관리할 수 없는 상황이다(오종훈, 최기선 2006).
과학기술문헌 중 생의학 분야의 전문용어들은 문맥에 따라 상이한 대상을 나타낼 수 있고 (예: ferritin은 단백질 또는 실험을 의미한다), 한 개체가 여러 개의 전문용어로 표현될 수 있고(예: PTEN, MMAC1은 동일한 유전자를 의미한다), 신규용어들이 신속하게 생성 되므로 전문용어 인식이 더욱 어렵다.7)
생의학 분야의 전문용어 자동 인식 연구는 크게 규칙 기반 연구, 통계 기반 연구 그리고 앞의 두 연구방법을 병행하는 혼합형 연구로 구분한다. 규칙 기반 연구는 사전이나 규칙을 사용하는 방법으로 수작업을 통한 규칙의 정확성과 사전의 크기가 인식의 정확률을 결정한다. 통계 기반 연구는 지도 학습과 비지도 학습으로 나눠진다. 지도 학습은 사람의 판단을 통해 만들어진 대량의 말뭉치가 준비되어 있을 때 사용하기 좋은 방법이고, 비지도식 학습은 소량의 말뭉치를 대상으로 초기 규칙을 학습.인식의 과정을 반복해 성능을 향상시키는 방법이다. 일반적으로 비지도 학습보다 지도학습이 좋은 인식 결과를 보인다. 학습에 사용되는 통계 모델은, 은닉 마코프 모델(hidden Markov model), 신경망(neural network), SVM(support vector machine), 최대 엔트로피 모델(maximum entropy model) 등이 있다.
Tanabe, Wilbur(2002)는 규칙기반 접근방법을 이용한 AbGene 시스템을 개발하였다. AbGene은 Brill 품사태거를 확장하여 유전자명과 단백질명에 관한 태그를 추가하였고, 생의학 분야 문헌으로부터 수집된 7,000개의 학습데이터를 이용해 학습하였고, 정확률 85.7%와 재현율 66.7%를 보여준다.
Chang, Schutze, Altman(2004)은 문장내에서 출현하는 전문용어후보들에 후보의 빈도수, 형태소 분석결과, 문맥 등을 고려하여 가중치를 할당하는 GAPSCORE 시스템을 개발하였다. 더 높은 점수를 획득한 후보는 유전자명, 단백질명이 될 확률이 높다. GAPS-CORE 시스템은 Yapex 말뭉치로 학습되었고 74%의 정확률, 81%의 재현율을 보인다 .
Zhou, Zhang, Su(2004)는 은닉마코프 모델(HMM : Hidden Markov Model)을 이용 하였다. 대문 자 시작 정보, 접두사와 접미사 정보, 품사정보, 시작 단어 등을 전문용어후보 추출을 위한 자질로 선택하였고, GENIA 말뭉치 2.1을 사용하여 학습 및 실험을 한 결과, 66.5%의 정확률과 66.6%의 재현율을 보였다.
현재까지 개발된 생의학 분야 전문용어 인식 기술은 전체적으로 F1-척도 75%와 85% 사이를 보여준다.
7) 본 연구에서 대상 데이터를 과학기술 분야 중 생의학 분야로 제한하였기 때문에, 생의학 분야 전문용어 추출기술에 초점을 맞춘다.
대용어는 대용의 기능을 담당하는 어휘로서 선행어보다 간결한 형식을 사용하여 반복되는 성분을 대신하는 것으로 명확한 진술이나 단어의 반복적인 사용을 피하고자할 때 사용하는 말이며, 대명사가 그 대표적인 예이다. 문장의 의미를 정확히 파악하기 위해서는 문장에 사용된 대용어가 이전 문장/대화의 어떤 사물/행위를 가리키는지를 구별하여야 하는데 이러한 과정을 대용어 처리 혹은 참조해소라 한다.
대용어 처리에 대한 연구는 꽤 오랜 역사를 가지고 있으나 실용화된 시스템을 사용하는 경우는 거의 없다. 과거에는 중심화 이론(centering theory)을 바탕으로 하는 규칙기반의 연구가 주로 진행되었으나 최근에 와서는 기계학습을 이용한 대용어 처리에 관한 연구도 활발히 진행되고 있다(Elsner and Charniak 2007). 대부분의 연구들의 성능은 뛰어나지 않지만 관계 추출 분야에서 대용어를 그대로 사용할 수 없고, 대용어가 가리키는 개체를 정확히 파악해서 이 정보를 이용한다면 관계 추출의 성능을 크게 개선할 수 있다.
대용어 참조해소 방법은 규칙기반에 의한 방법(Grosz, Joshi 1995; Carbonell, Brown 1988; Lappin, H. Leass 1994)과 기계학습에 의한 방법(Soon, Ng, Lim 2003; Yang, Su, Tan 2008)이 있다.
규칙기반에 의한 방법은 대용어에 대하여 대용어 후보들을 수집하여 성(gender), 수(number) 등의 제약조건을 만족하는 후보들 중에서 경험적으로 가능성이 높은 후보를 선택한다. 규칙기반 시스템의 경우 여러 유형의 대용어 관계를 규칙화하는 것이 쉽지 않다는 점에서 많은 제약이 있다.
Lapain & Leass(1994)는 RAP(Resolution of Anaphora Procedure)를 제안하였다. RAP는 제약조건에 만족하는 후보들 중 구의 속성이나 위치 문장거리 등에 대해 상이한 가중치를 할당하여 가장 높은 점수를 획득한 후보를 선택하는 시스템이다.
기계학습 방법은 학습 말뭉치로부터 대용어 관계를 결정하는 규칙을 학습하는 접근방법이다. 대용어와 대용어가 태깅된 학습 말뭉치로부터 유용한 자질들을 선택하고 추출하여, 모델을 학습한다. 학습된 모델을 만드는 방법은 크게 두 가지로 구분된다. 첫 번째 방법은 대용어와 선행어 후보가 대용 관계에 속하는지를 결정하는 이진 분류 방법(Soon, Ng, Lim 2003; Pnnzetto, Strube 2006)이다. 두 번째 방법은 선행어 후보들에 대해서 점수를 계산하고 그 점수에 따라 선행어 후보들을 순위화하여 가장 높은 순위에 있는 후보를 선택하는 방법(Lappin, Leass 1994)이다.
Kilicaslan, Guner, Yildrim(2009)은 SVM (Support Vector Machine)을 이용한 터키어의 참조해소 시스템을 제안하였으며 약 73%의 정확률을 보인다. Denis, Baldridge(2009)는 순위화 모델을 제안하였고, 영어에 대하여 72.4%의 정확률을 보인다. Nguyen, Kim, Tsujii(2008)는 GENIA 말뭉치에 대하여 최대 엔트로피 모델을 이용해서 약 71.43%의 정확도를 보인다.
본 연구에서는 기존에 독립적으로 연구되던 개체명인식 분야, 전문용어 인식분야, 대용어 참조해소 분야를 통합하여 과학기술문헌을 대상으로 핵심개체를 탐지하기 위한 플랫폼을 제안한다. 과학기술문헌 중 대상분야를 생의학 분야로 한정하고, 추출할 핵심개체를 정의하고, 핵심개체 추출을 위한 분야별 기계학습 모델을 개발하고 이를 통합한 과학기술 핵심개체 신규탐지 시스템을 개발한다.
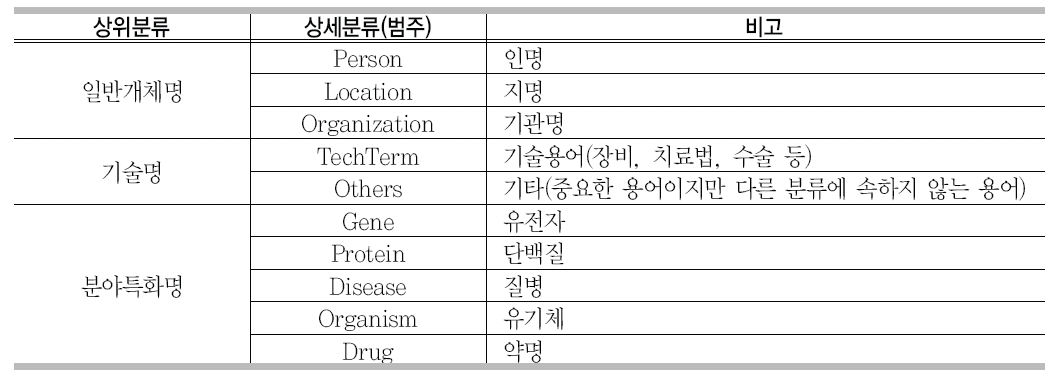
본 연구에서 대상 데이터를 생의학 분야 문헌으로 선정하였고, 생의학 분야 문헌에서 중요한 역할을 담당하는 개체명들과 전문용어들을 통합한 핵심개체들을 <표 2>와 같이 10개의 분류로 정의하였다.
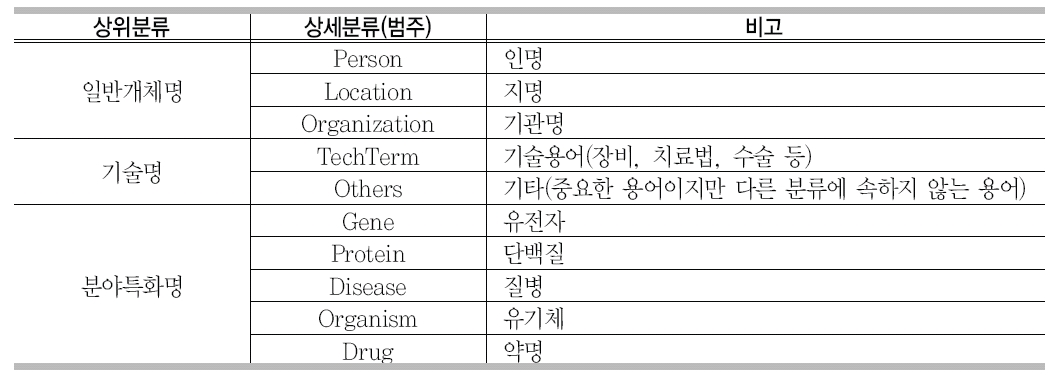
핵심개체 분류표
일반개체명은 개체명 인식에서 주로 사용되는 인명(Person), 지명(Location), 기관명(Organization)으로 분류하였으며, 전문용어는 생의학 분야에 특화된 분야특화명과 장비, 치료법, 수술 등에 포함되는 기술용어와 기타로 분류하였다.
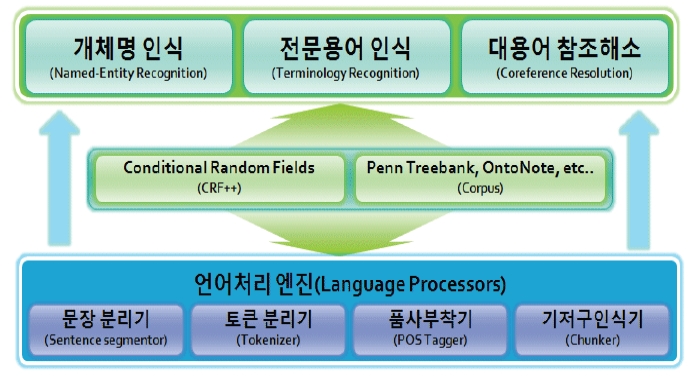
과학기술문헌에서 개체명과 전문용어를 인식하고, 대용어에 대한 참조해소를 수행하는 전체적인 시스템은 <그림 1>과 같다. 하부시스템인 언어처리 엔진(Language Processor)은 문장분리기, 토큰 분리기, 품사부착기, 기저구 인식기의 4가지 모듈로 구성되어 입력된 문서를 가공하고, 필요한 자질들을 추출하는 역할을 담당한다. 개체명 인식기, 전문용어 인식기, 대용어 참조해소기는 하부의 언어처리 과정에서 추출된 각각의 자질들 또는 통합된 자질들을 사용한다.
전체적인 작업은 (1)언어처리 단계, (2)전문 용어 및 개체명 인식 단계, (3)대용어 참조해소 단계로 진행된다.
첫째, 언어처리 단계에서는 입력된 문장을 문서단위로 분리하고, 의미 있는 단위로 토큰을 인식하고, 토큰에 대한 적절한 품사를 부착하고, 기저명사구를 인식한다.
둘째, 개체명 인식기에서 인명, 지명, 기관명을 추출하고, 전문용어 인식기에서 기술명(2개 분야) 및 분야특화명(5개 분야)을 추출한 뒤, 그 결과를 하나의 문서로 통합한다.
셋째, 대용어 참조해소기에서 문서내에 출현하는 대명사들을 대상으로 선행어를 발견하는 작업을수행한다. 선행어가 발견된 대명사에 대해서 선행어에 대한 정보(개체명, 전문용어)를 동일하게 적용하고, 선행어에 대한 참조 정보를 함께 기록한다.
본 연구에서는 개체명과 전문용어에 대한 자동인식 그리고 대용어에 대한 선행어 자동탐지를 위해 기계학습모델을 이용하였다. 지금까지 연구되어온 여러 기계학습모델 중에서 속도, 정확성 등을 고려하여 CRF 모델8)을 선정하였다. 기계학습모델을 구현하기 위해서는 필요한 학습 자질을 추출하기 위해 다양한 언어적인 분석이 필요하고, 이를 위해 문장분리, 토큰분리, 품사부착, 기저구 인식 등의 언어처리 모듈을 개발하였다. 기존에 공개된 모듈들은 서로 다른 컴퓨터 언어들을 사용하고, 입력과 출력의 형식이 서로 상이하여 전체 시스템의 효율적인 통합과 성능을 위해 직접 개발하였다.
Penn Treebank의 구성
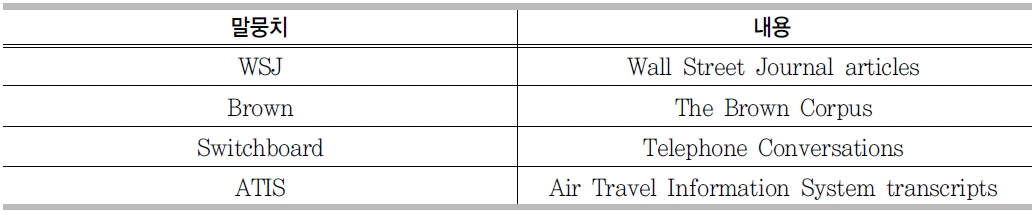
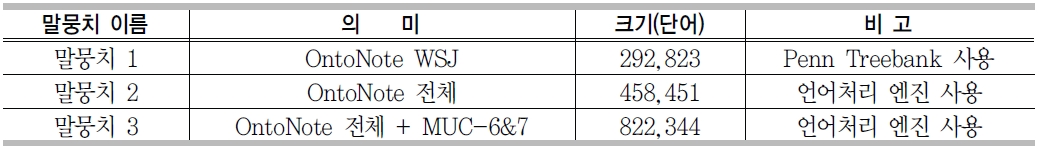
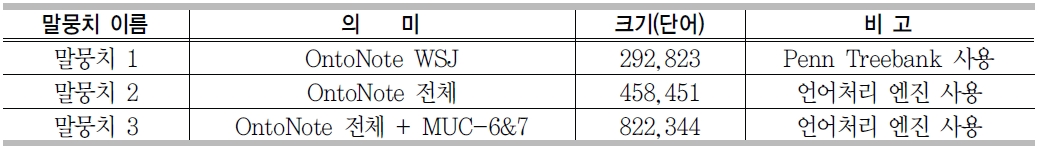
<표 3>은 언어처리엔진의 기계학습모델을 학습 및 테스트하기 위해 사용한 말뭉치이다. Penn Treebank는 자연어에 대한 언어학적 구조를 분석하기 위한 목적을 위해 진행된 프로젝트의 산출물로 문장의 구문구조와 의미론적 정보, 품사정보를 담고 있다.
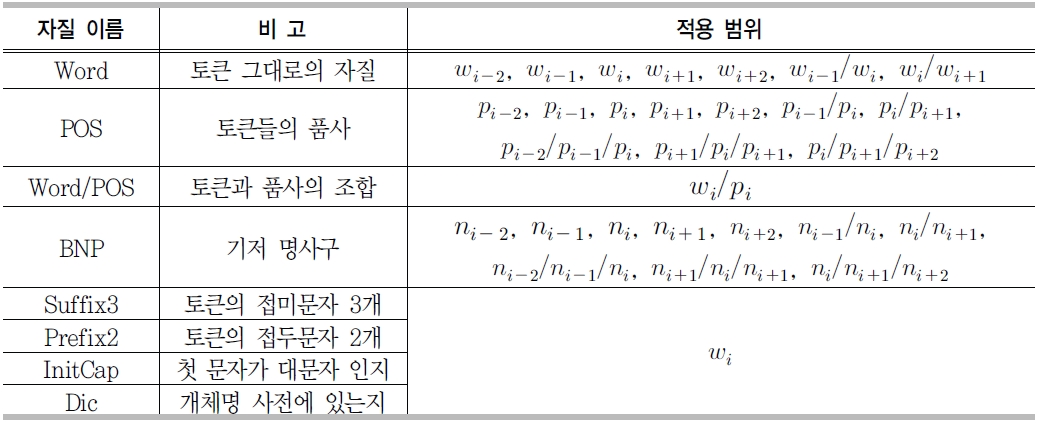
토큰분리기에서는 Char(현재 자신의 문자), Pre1Next1Cap(앞뒤가 대문자), SpecialChar(특수기호), Prev_CCO(자신포함 이전 3문자), Next_OCC(자신포함 이후 3문자) 자질을 사용하고, 현재단어에만 자질들을 적용하였다.
품사부착기는 Word(단어), Suffix3(접미사 3문자), Prefix2(접두사 2문자)를 자질로 취하였고 이전 2개 단어와 현재 그리고 다음 2개 단어에 자질들을 적용하였다.
기저구인식기는 품사부착기에서 사용한 자질 외에 POS(품사정보), InitCap(첫글자가 대문자) 자질을 선택하여 이전 2개 단어, 현재, 다음 2개 단어와 이전 2개 품사, 현재품사, 다음 2개 품사에 자질들을 적용하였다.
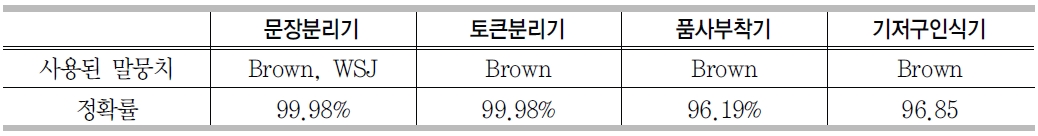
실험을 위해 말뭉치의 1/10을 학습을 위해 사용하였고, 9/10를 테스트를 위해 사용하였다. <표 4>는 본 연구에서 개발한 언어처리엔진에서 사용한 말뭉치 종류와 정확률을 보여준다. 4개의 모듈 모두 높은 정확률을 보여주었다.
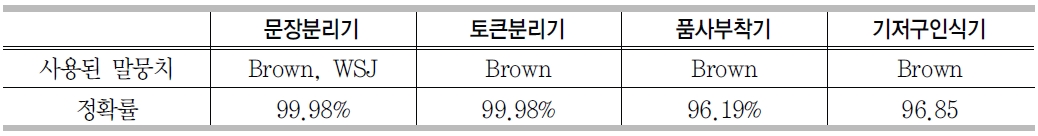
언어처리엔진 정확률
개체명 인식의 자질 집합
8) CRF(Conditional Random Fields)는 일반문서 또는 생물학 서열같은 연속적인 데이터의 태깅이나 파싱을 위해 사용되는 기계학습모델의 한 종류(http://en.wikipedia.org/wiki/Conditional_random_field)
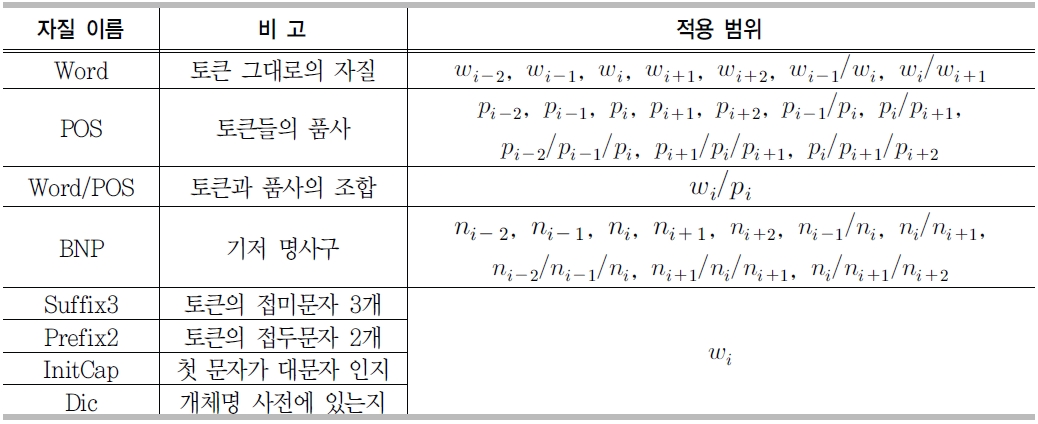
MUC-6와 CoNLL 2002, 2003에서 제안된 개체명 인식 시스템들은 2장에서 언급한 BIO 태그를 이용한 기계학습 방법을 이용하였다. 기계학습 방법의 경우 자질의 수와 정확률이 항상 비례하지 않고, 자질의 조합에 따라 성능이 달라지는 경우가 많으므로 모든 자질들을 동시에 사용하지 않는다. 본 연구에서는 정보이득(information gain)을 이용하여 필요한 자질을 선택한다. <표 5>는 선택된 자질집합을 보여준다.
<표 5>에서 보는 바와 같이 사전(Dic)자질을 제외하고 다른 자질들은 3.1에서 설명한 언어처리엔진에서 사용하는 자질들과 동일하다. 개체명 인식을 위해 사전을 사용하는 경우, 시스템 전체가 기계학습 기반이므로 사전에 존재유무 자체를 하나의 자질로 인식하여 시스템을 구현하여야 한다. 하지만 학습 말뭉치의 경우 형태소 단위로 만들어져 있고, 개체명은 구 단위로 이루어져 있어서 바로 적용할 수 없다. 본 연구에서는 "BIPLO" 템플릿을 제작하여 이를 해결하였다. "BI"는 개체명의 처음과 중간을 나타내고, "PLO"는 인명, 지명, 기관명을 구분하는 태그이다.
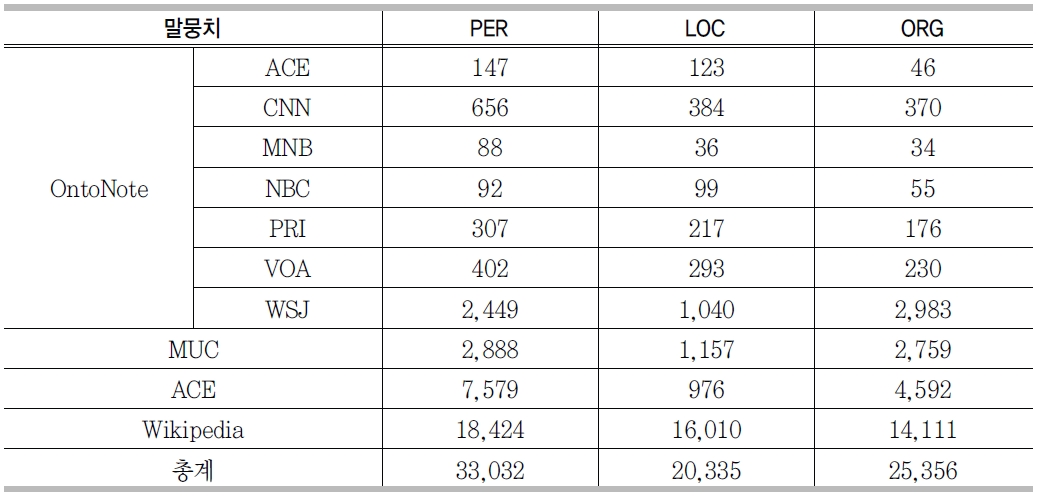
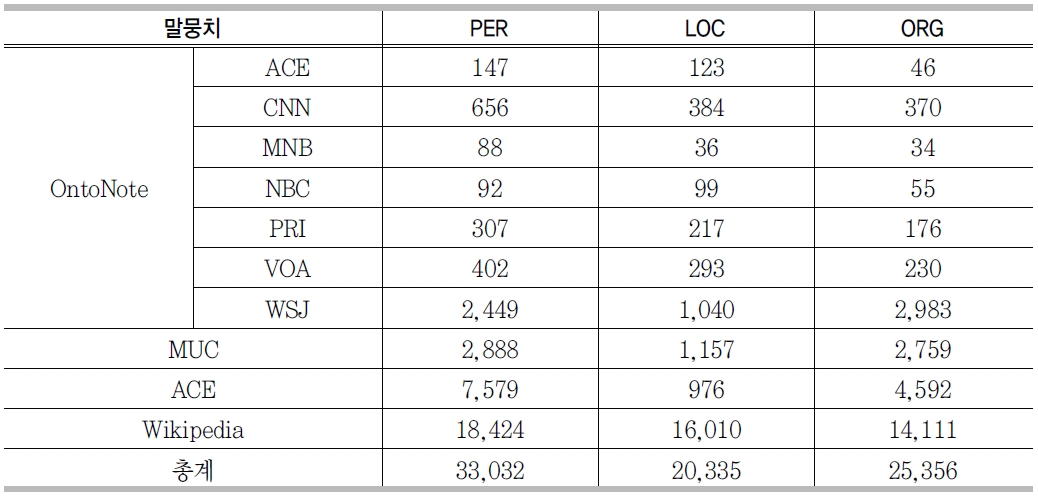
일반적으로 기계학습 기반 엔진들은 학습데이터가 충분할 경우 좋은 성능을 보여준다. 개체명 인식 성능 향상을 위해 <표 6>에서 보는 바와 같이, OntoNote 말뭉치, MUC 말뭉치 등 다양한 언어자원을 사용하여 개체명 사전을 구축하였다.
구축된 개체명 사전의 표제어 수
<표 6>에서 사용한 외부 말뭉치들은 개체명 정보를 포함하지만, 본 연구의 개체명 인식기에서 사용한 자질들을 위한 문장분리 정보, 토큰분리 정보, 품사 정보가 결여되어 있다. 개체명 인식기의 성능을 높이기 위해, 3.3에서 소개한 언어처리모듈을 이용하여 문장분리 정보, 품사 정보 등을 외부 말뭉치에 추가하여 본 연구에서 사용할 수 있도록 확장하였다.
전문용어는 핵심개체 중 분야특화명과 기술명을 의미하며, Gene(유전자), Protein(단백질), Organism(유기체), Disease(질병), Drug(약명), TechTerm(기술용어), Others(기타) 7개의 범주로 분류된다.
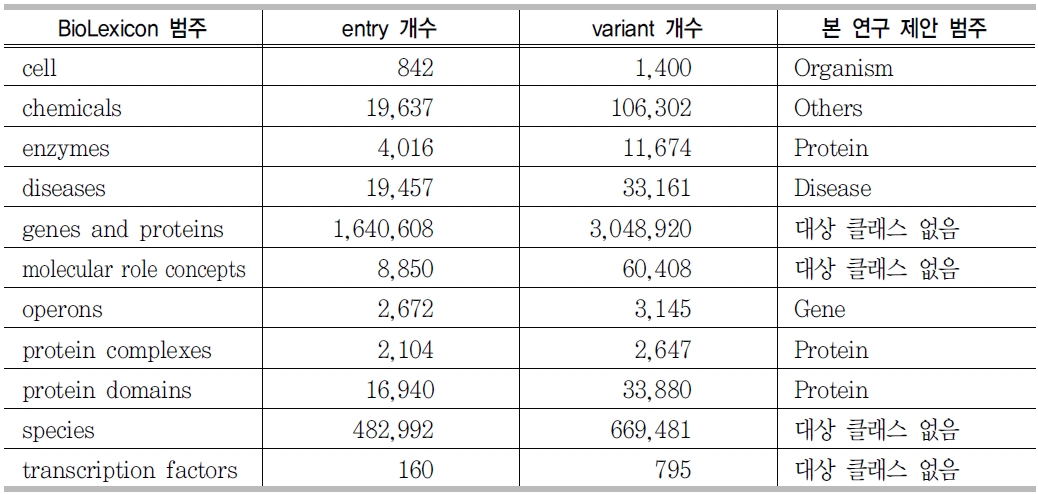
대상 데이터를 생의학 분야로 선정하였기 때문에, 전문용어 인식기의 성능을 높이기 위하여 기 구축되어 있는 생의학 분야 말뭉치인 BioLexicon, Gene Ontology, GENIA를 이용하였다.
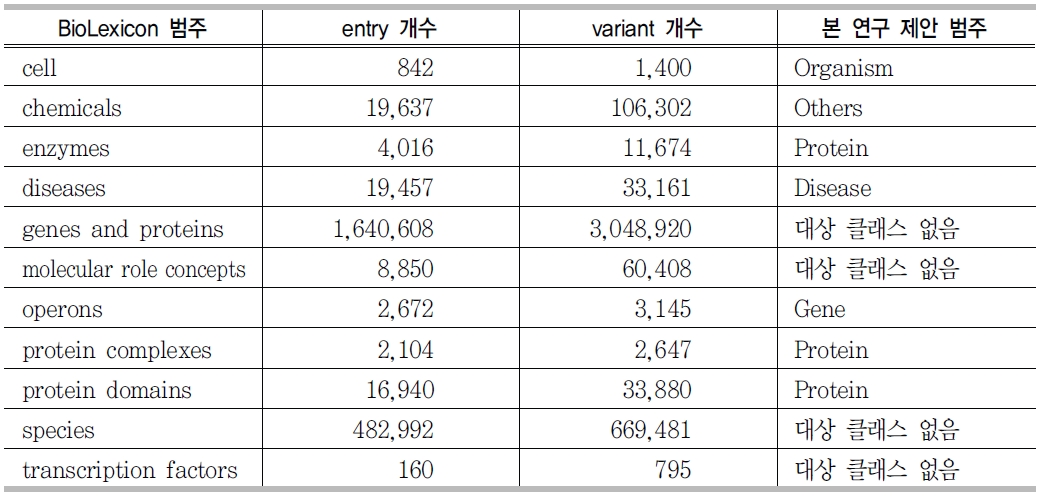
사용된 말뭉치들은 본 연구에서 선정한 7개의 범주와 일치하는 부분도 있지만 세분화된 범주나 전혀 관계없는 범주 또한 존재하기 때문에, 본 연구에서 제안한 핵심개체의 범주로 변환하는 작업을 선행하였다. <표 7>은 BioLexicon 말뭉치 범주와 대응되는 범주를 보여준다.
BioLexicon 범주 및 대응 범주
[<표 8>] 전문용어 인식을 위한 자질 집합과 자질값
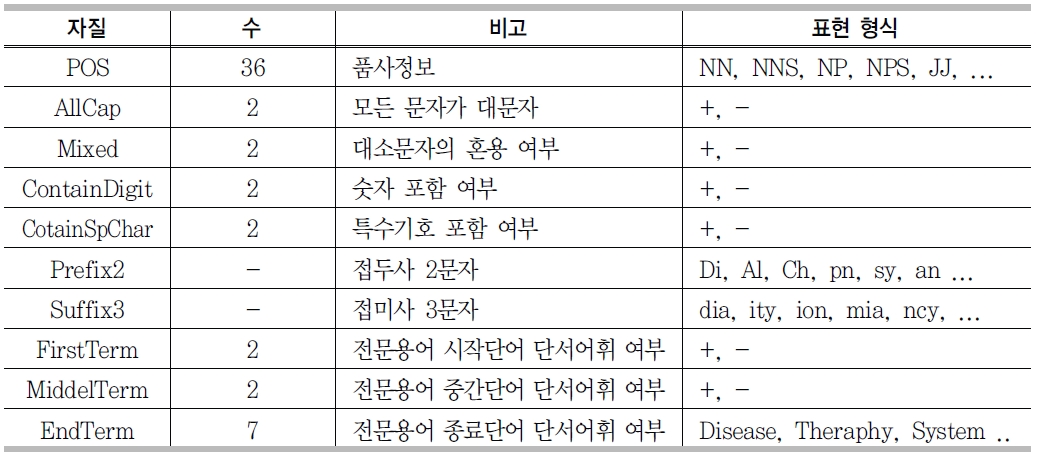
전문용어 인식을 위한 자질 집합과 자질값
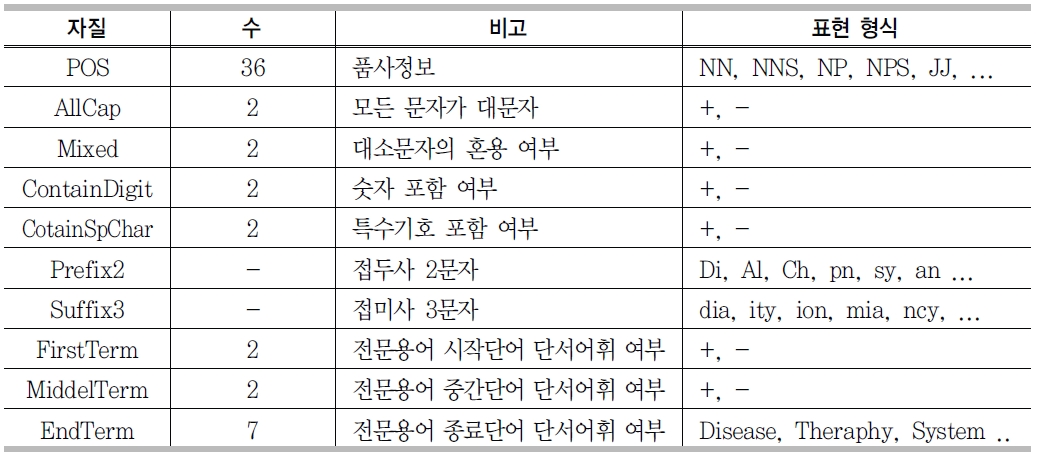
전문용어 인식을 위한 기계학습모델의 자질들은 <표 8>에서 보는 바와 같이 10가지가 선정되었다. 통계적으로 볼 때 생의학 분야 전문 용어 중 소문자로만 구성된 용어가 절반 이상이지만, 특정질병, 단백질, 약명, 유전자의 이름에 해당하는 전문용어는 대문자, 숫자, 특수 기호 등을 포함하는 경우가 많다 (예 : 5HT2a, BHC110/LSD1, CaMKII).
이러한 생의학 분야 전문용어의 특성을 반영하기 위해, 대소문자가 섞여있는지 여부를 묻는 자질(Mixed)과 숫자, 특수기호의 포함 여부를 묻는 자질(ContainDigit, Contain-SpChar)을 사용하였다. 알파벳 20자 이상의 긴 용어들의 경우 약어로 표현되는 경향을 반영하기 위해, 모두 대문자인지 여부를 묻는 자질(AllCap)을 사용하였다 .
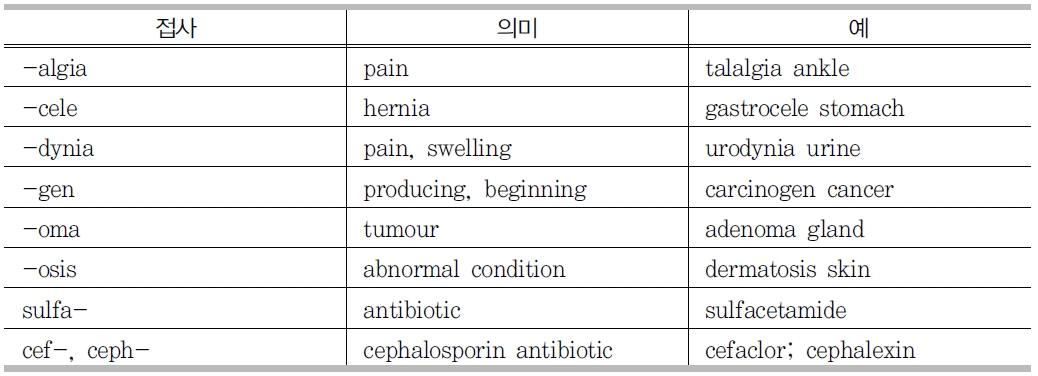
생의학 분야 전문용어의 또 다른 특징은 중 하나는 그리스 라틴어원을 사용한다는 점이다. 이러한 용어들은 접두사, 접미사가 동일한 형태소로 구성되며 특별한 의미를 갖는다. <표 9>는 특정 접두사, 접미사가 가지는 의미와 예를 보여준다.
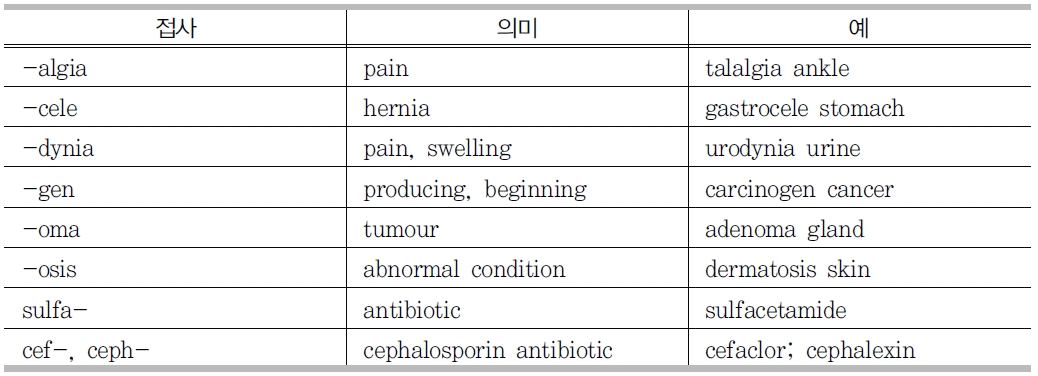
질병명과 약명에 관련된 접두사와 접미사
전문용어 단서어휘
접사의 길이는 유동적이므로, 특징만 뽑아낼 수 있도록 접미사는 3개의 문자, 접두사는 2개의 문자를 자질(Prefix2, Suffix3)로 사용하였다.
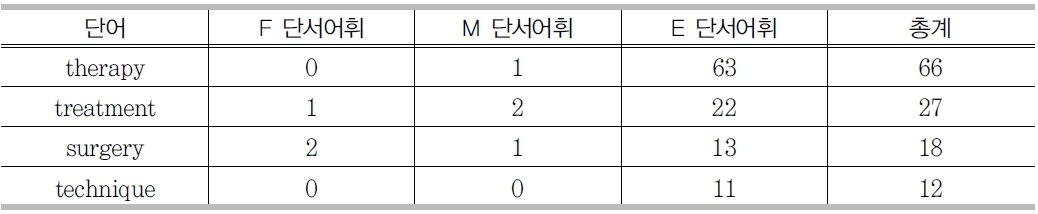
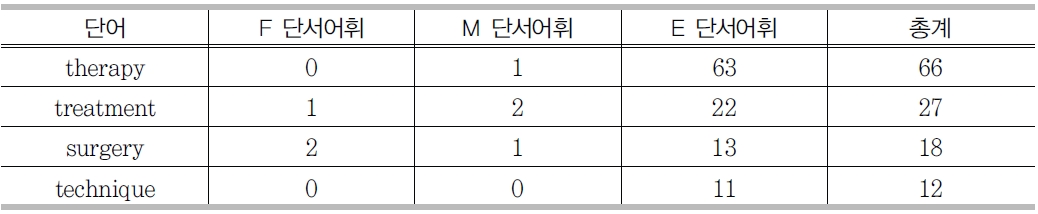
2어절 이상의 복합어로 구성된 전문용어에 대해서 전문용어 사전을 활용하였다. 각 분야의 전문용어 사전마다 어절별로 나누어서 가장 앞에 나오는 어절(F단서어휘), 중간에 나오는 어절(M단서어휘), 마지막에 나오는 어절(E 단서어휘)로 나누어 빈도별로 정리한 것이 단서어휘이다(<표 10> 참조).
단서어휘는 전문용어 인식 시스템에서 중요한 역할을 담당 한다. 특히 복합어인 전문용어 중 마지막 단어와 관련된 단서어휘는 경계인식 외에, 분야 결정을 위한 중요한 자질이 되므로, 단서어휘 사전에 포함되어 있는지에 대한 여부뿐 아니라, 분야정보도 자질 값에 포함하였다.
본 연구에서 제안된 시스템은 크게 두 부분으로 구성된다. 하나는 대용어로서 대명사를 인식하는 시스템이고 다른 하나는 인식된 대명사의 선행어를 찾는 시스템이다. 전자를 대용어 인식 시스템(anaphoric pronoun identifier)이라고 하고 후자를 선행어 결정 시스템(pronoun resolver)이라고 한다. 품사가 대명사(PRP)라고 해서 모두 대용어는 아니다. 즉 문서 내에 어떤 선행어를 가리키지 않는 대명사가 존재한다는 것이다. 예를 들면 문장 "Itis important not to give up"에서 'It'은 선행어를 가지지 않는 대명사이다. 대용어 인식 시스템은 품사가 대명사(PRP)인 단어 중에서 대용어를 찾아내는 시스템이다.
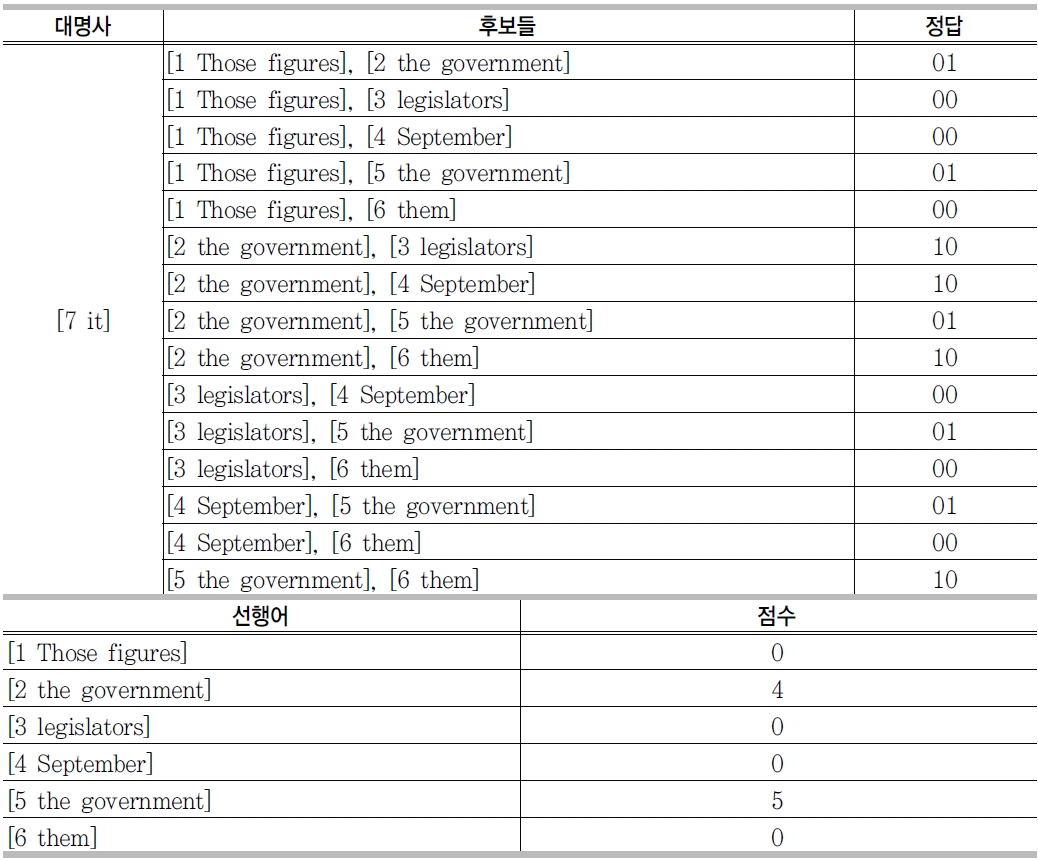
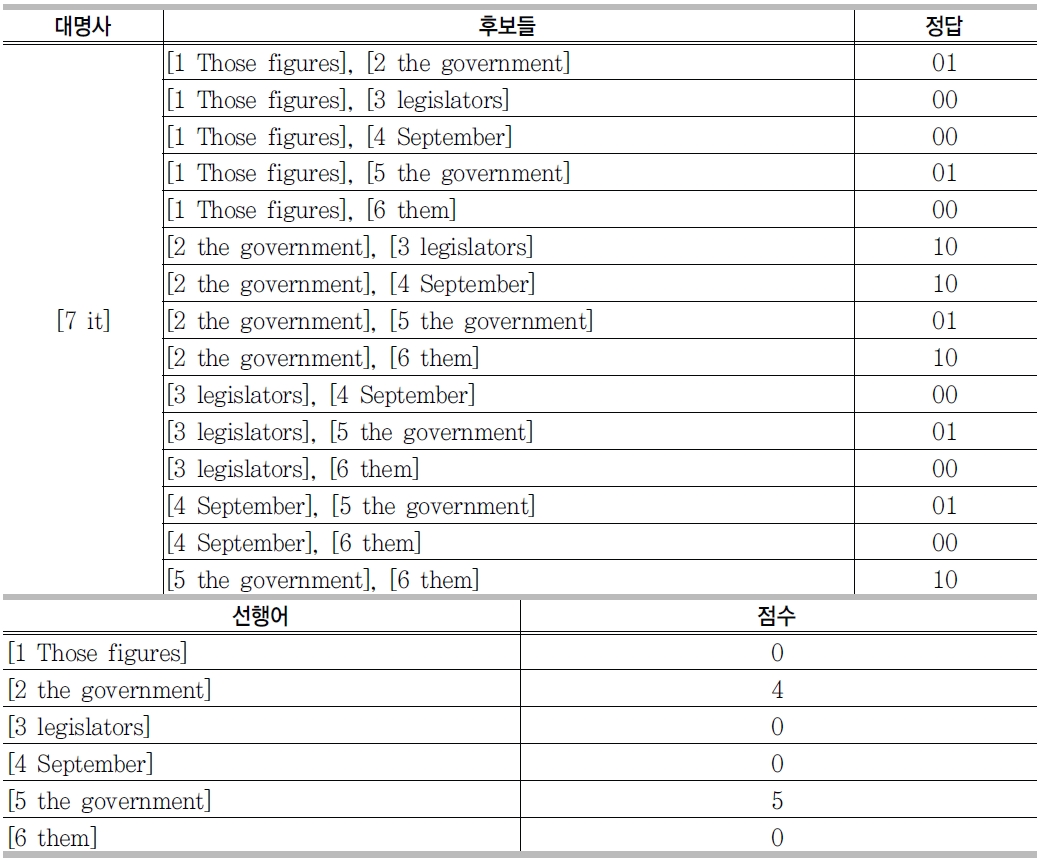
대용어 인식 시스템에 의해서 인식된 대용대명사(anaphoric pronoun)를 대상으로 선행어를 결정하는 시스템이 선행어 결정 시스템이다. 아래의 예문을 보면 2개의 대용대명 사 'them'과 'it'이 있으며 각각의 선행어는 'Those figure'와 'the government'이다.

대용어 인식 시스템의 자질 집합
대용어 참조를 해소를 위한 자질은 대용대명사 자신과 주변의 어휘 정보들과 품사 정보를 사용한다. 여러 번의 실험을통해서 <표 11>과 같은 자질 집합을 선택하였다.
선행어 결정 시스템에 사용되는 자질 집합은 대용 대명사에 대한 자질, 선행어에 대한 자질, 대용 대명사와 선행어 사이의 관계 자질 세 가지로 분류한다. 각각에 대해 품사나 격, 위치 정보 등을 조합하여 자질들을 생성하게 된다. 하지만 자질들의 수가 너무 많고, 기계학습의 학습 속도 문제로 인해 이러한 자질의 모든 조합에 대해 실험하는 것은 사실상 불가능하므로, 기존 연구들을 참고하여 사용할 수 있는 자질들의 목록을 작성하고, 결정 트리에서 사용되는 정보 이득(information gain)을 이용하여 선행어 결정 시스템을 위한 자질 집합을 <표12>와 같이 결정하였다.
본 연구에서 선행어 결정을 위해 TCM(twin-candidate model)을 사용한다. TCM은 대용대명사와 각각의 후보 선행어들 사이의 관계에 의해서 최종 선행어를 결정하는 것이 아니라 두 후보 선행어들끼리 경쟁할 수 있도록 모델링된 것이다. TCM에 의해서 모델이 되면 최종적으로 선행어를 결정하기 위해서 후보들끼리 경쟁을 하게 되는데 경쟁 방법에는 크게 두 가지 방법이 존재한다.
첫 번째는 승자진출전(tournament) 방법으로 모든 후보들 중 가장 먼저 2개의 후보를 선택하고 경쟁을 붙이게 된다. 이 경쟁에서 살아남은 후보와 아직 선택되지 않은 후보 중 1개를 선택하여 다시 경쟁하는 형태이다. 모든 후보들이 선택되어 남아있는 후보가 없어지면 경쟁은 끝이 나고 최후에 살아남아 있는 후보가 정답으로 결정되는 시스템이다.

승자진출전을 통한 선행어 결정
두 번째는 연맹전(league) 방법으로 모든 후보들에 대해 경쟁을 붙여서 가장 승률이 좋은 후보를 선택하는 시스템이다. 경쟁에 대한 정답 태그는 총 3가지로 "00", "10", "01"이 그것이다. "00"은 둘 다 정답이 아닌 경우이고 "10"은 앞의 것이, "01"은 뒤에 것이 경쟁에서 살아남은 것을 표시하는 태그이다. <표 13>은 승사진출전 방식의 실제 경쟁하는 예를, <표 14>는 연맹전 방식의 실제 예를 보여준다.
본 연구는 한국과학기술정보연구원이 보유하고 있는 과학기술데이터 중에서 생의학 분야 데이터에 대한 핵심개체를 인식하는 데 그 목적이 있다. 이를 위해 구축된 테스트컬렉션 KEEC 20099)를 대상으로 본 연구에서 개발한 핵심개체 탐지기술에 관한 성능을 실험하였다. 2장에서 언급한 관련 연구와 동일한 분야와 실험데이터를 사용하지 않아 정확한 실험비교를 수행하지 못하지만 전체적으로 비교적 높은 수준의 성능을 보여주었다.
연맹전을 통한 선행어 결정
실험에 사용된 말뭉치의 종류

개체명 인식의 성능 평가
개체명 시스템은 두 가지 방법으로 구현되었다. 하나는 개체명의 경계를 인식한 후에 개체명의 종류를 분류하는 경계분류 순차 시스템이고 다른 하나는 경계인식과 분류를 동시에 수행하는 경계분류 동시 시스템이다. <표 16>은 3.3에서 소개한 세 종류의 말뭉치를 각 시스템에 적용하여 성능을 평가한 결과이다.
<표 16>에서 보는 바와 같이 말뭉치의 규모가 확장함에 따라, 경계분류 순차시스템과 경계분류 동시 시스템 모두 더 좋은 결과를 나타내고, 경계인식과 개체명분류를 순차적으로 하는 경우보다 동시에 수행하는 경계분류 동시 시스템의 성능이 우월함을 알 수 있다. 본 연구에서는 이 결과를 바탕으로 통합시스템을 위한 개체명 인식기를 경계분류 동시 시스템으로 적용하였다.
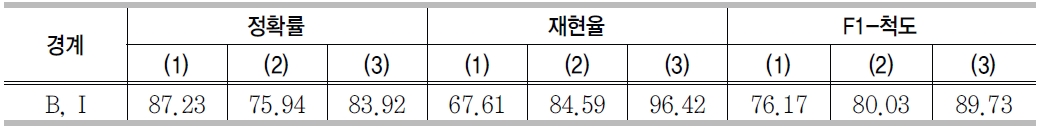
전문용어 경계인식의 성능 평가
9) KEEC 2009(KISTI Entity Extraction Collection 2009) : 한국과학기술정보연구원이 보유하고 있는 해외학술지 중 생의학 관련 분야 문헌을 선정하여 테스트컬렉션을 구축하였으며, 핵심개체 2만1,142를 포함하는 8,303 문장으로 구성되어 있다.
전문용어 인식기도 개체명 인식기와 동일하게 전문용어에 대한 경계인식과 인식된 전문용어에 대하여 분류를 수행하는 두 가지 시스템으로 구현되었다. 성능평가를 위해서 한국과학 기술정보연구원에서 구축한 테스트컬렉션(KEEC 2009)을 이용하였다. 성능평가는 ㉮외부 사전을 이용한 경우와 ㉯단서어휘를 일반화한 경우를 조합하여 (1)㉮, ㉯ 모두 사용하지 않은 경우, (2)㉯만 사용한 경우, (3)㉮, ㉯ 모두 사용한 경우에 대하여 각각 수행하였다.
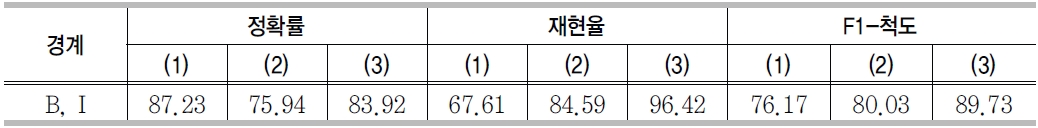
<표 17>은 전문용어 경계인식의 실험결과이다. 정확률은 (1)의 경우가, 재현율과 F1-척도는 (3)의 경우가 가장 좋은 결과를 보였다. (1)의 정확률은 87.23%로 가장 높은 반면 재현율이 다른 경우와 비교하여 현저하게 낮음을 보였다 . (1)의 경우 외부사전과 단서어휘를 사용하지 않았기 때문에 전문용어 인식률은 낮지만, 인식된 전문용어에 대한 경계는 정확히 인식했음을 의미한다. 사전과 단서어휘를 함께 사용한 (3)의 경우 정확률, 재현율 모두에서 전반적으로 높은 성능을 보여준다.
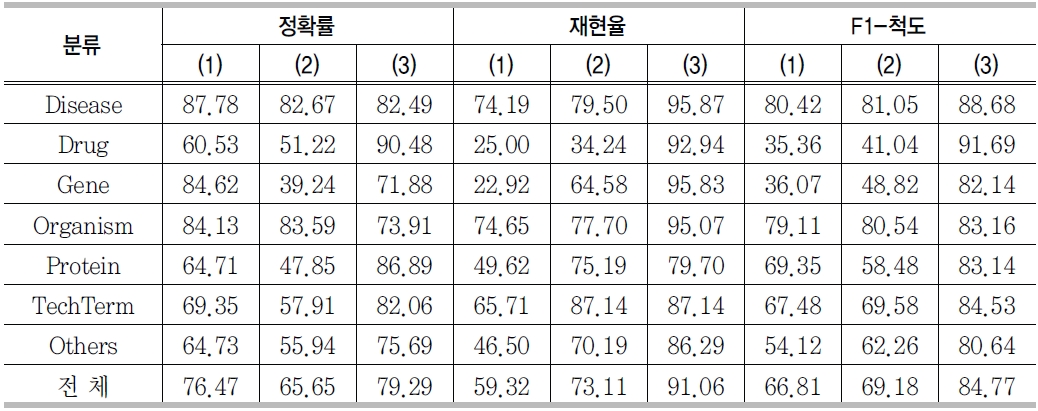
<표 18>은 전문용어 분야인식의 실험결과이다. 경계인식의 경우와 마찬가지로 외부사전과 단서어휘를 사용한 (3)의 경우에 가장 좋은 실험결과를 보여주었다. 사전이나 단서어휘를 사용하지 않은 (1), (2)의 경우에는 실험수치가 상당히 낮은 경우를 볼 수 있다. 정확률 측정 (2)의 Gene은 39.24%, 재현율 측정 (1)의 Gene은 22.92%로 매우 낮았다.
전문용어의 경계인식과 분야인식의 두 경우 모두, 외부 사전과 단서어휘를 이용한 경우에 가장 좋은 F-1 척도를 보여준다. 본 연구의 결과는 2.2에서 소개한 생의학 분야 전문용어 인식에 대한 세계 최고 수준에 근접(F1-척도 75%∼85%사이)하는 실험결과를 보였다.
대용어 참조해소 시스템에서는 대용대명사가 가리키는 선행어를 정확하게 찾는 비율을 성능 척도로 사용하며 이를 선행어 결정 정확률
Aa = nca / na × 100
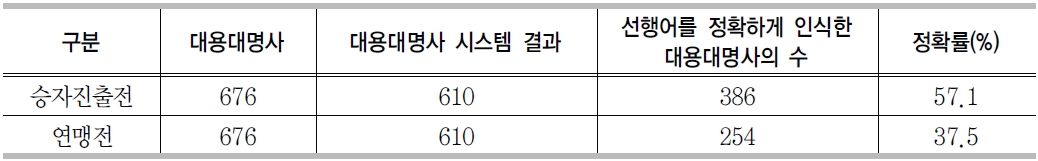
대용어 참조해소 시스템의 성능평가를 위해 전문용어 평가를 위해 사용했던 테스트컬렉션과 동일한 KEEC 2009를 사용하였다. <표 19>는 연맹전과 승자진출전에 대한 성능 평가 결과이다. 모두 대용대명사 결정 시스템을 통해 결정된 대용대명사를 대상으로 선행어 결정 시스템을 적용한 결과로써 대용대명사 결정 시스템의 에러가 그대로 전파된 결과이다.
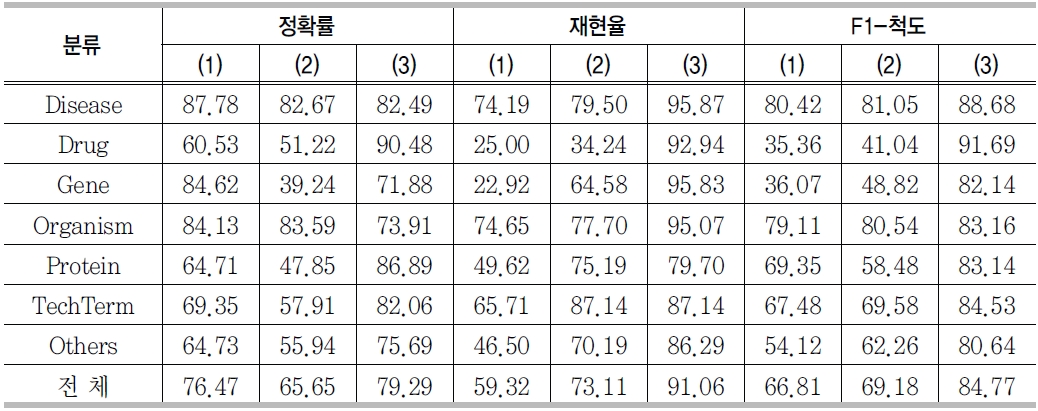
전문용어 분야분류의 성능 평가
<표 19>를 살펴보면 승자진출전에 비해서 연맹전이 상당히 낮은 정확률을 보이는 것을 알 수 있다. 이는 연맹전의 특성상 모든 후보들 간의 경쟁이 일어남에 따라 정답 태그 중 "00"의 비율이 너무 높아져서 학습 결과가 "00"쪽으로 치우치는 현상 때문이다.
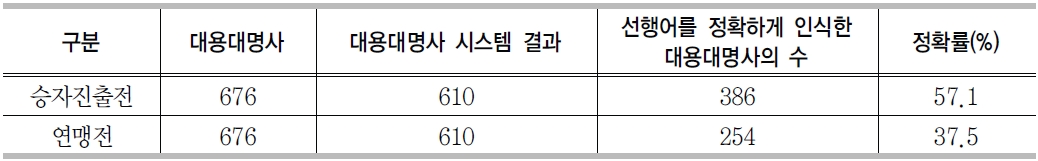
대용어 참조해소 시스템의 정확률

선행어 결정 시스템의 정확률
[<표 21>] 대용대명사 결정 시스템을 거치지 않은 참조해소 시스템 정확률

대용대명사 결정 시스템을 거치지 않은 참조해소 시스템 정확률
본 연구에서는 독립적으로 개발되어오던 개체명 인식 기술과 전문용어 인식 기술을 하나 의 작업으로 통합하여, 과학기술문헌에 포함된 핵심개체인 개체명(named entity)과 전문 용어(terminology)를 자동으로 인식하고 이들을 가리 키는 대용어를 결정하는 시스템을 개발하였다.
대상 데이터는 한국과학기술정보연구원이 보유하고 있는 과학기술데이터 중 생의학 분야로 선정하였고, 일반적인 개체명과 분야 종속적인 전문용어들에 대한 분류를 핵심개체로 정의하였다. 일반 개체명은 인명(Person), 지명(Location), 기관명(Organization) 3개 분류로 지정하고, 전문용어는 질병명(disease), 병명(drug), 유전자명(gene), 생물조직명(organism), 단백질명(protein), 기술명(techterm), 기타(others) 7개 분류로 지정하였다.
개체명 인식, 전문용어인식, 대용어 참조해소에 대한 세계적인 연구방향과 수준을 파악하고, 이 기술들에 공통적으로 적용할 수 방법으로 기계학습방법 중 Conditional Random Field(CRF)를 선정하였다. 문장분리기, 토큰 분리기, 품사부착기, 기저구 인식기 등의 하부 언어처리 엔진을 개발하여 통합플랫폼에 공통으로 사용하였다.
개체명 인식기는 개체명에 대한 경계인식과 인식된 개체명에 대한 분류를 순차적으로 수행하는 경계분류 순차 시스템과 이 작업을 동시에 수행하는 경계분류 동시 시스템을 구현하여, 성능을 비교하였다. 두 시스템 모두 다양한 외부 말뭉치를 이용하여 학습 및 실험하였고 더 좋은 성능을 나타낸 경계분류 동시 시스템을 최종시스템으로 선정하였다. 전문용어 인식기는 생의학 분야의 전문용어 특성들을 조사하여 기계학습모델의 자질로서 선정하였고, 성능 향상을 위해 외부 말뭉치들을 가공하고, 단서어휘를 추출하여 인식시스템에 적용하였다. 대용어 참조해소기는 경쟁에서 살아남은 후보와 신규후보와 경쟁하는 승자진출전 방법, 모든 후보들에 대해 경쟁을 하는 연맹전방법 두 가지로 구현하여 성능을 실험하였다. 더 높은 성능을 보여준 승자진출전 방식이 최종시스템으로 결정되었다.
과학기술핵심개체 자동인식 통합 플랫폼은 전체적으로 비교적 높은 성능을보여주었고, 생의학 분야 데이터에 적용하여 핵심개체를 추출하고, 관계추출 시스템의 기반데이터로 활용하였고, 앞으로 과학기술 신규용어 탐지, 과학기술사전 자동구축, 의미 기반 정보검색, 관계 추출 기반 질의응답 등 한국과학기술정보연구원에서 제공하는 다양한 정보서비스 분야에 활용될 것으로 기대된다.