This study screened the biological activity of an acidified gill extract of the Manila clam Ruditapes philippinarum including antimicrobial, hemolytic, membrane permeabilization, and DNA-binding activity, and purified the antimicrobial material. The acidified gill extract showed potent antimicrobial activity against Bacillus subtilis and Escherichia coli without significant hemolytic activity, but showed no membrane permeabilization or DNA-binding ability. An antimicrobial material was purified from the acidified gill extract using C18 reversed-phase and cation-exchange high-performance liquid chromatography (HPLC). Treatment of the purified material with trypsin completely abolished all of the antibacterial activity against Bacillus subtilis, suggesting that the purified material is a proteinaceous antibiotic. The molecular weight of the purified material was 2571.9 Da, but no primary structural information was obtained due to N-terminal blocking. A future study should confirm the primary structure. Our results suggest that the Manila clam gill contains proteinaceous antibiotics that have a role in first-line defense. This information could be used to better understand the Manila clam innate immune system.
대부분의 척추동물 및 무척추동물들은 세균, 바이러스, 곰팡이, 또는 기생충 등과 같은 외부 미생물의 침입으로부터 자신을 방어하기 위한 효과적인 면역 체계를 각각 포함하고 있다(Hancock and Diamond, 2000). 선천면역과 적응면역이 각각 잘 발달되어 있는 척추동물과는 달리 무척추동물은 선천면역 만을 포함하고 있으므로 외부 환경에 대한 방어 작용은 오로지 선천면역에만 의존하고 있다. 일반적으로 선천면역은 외부 침입물질에 대한 초기방어 및 적응 면역의 유도를 담당하므로 그 기능과 역할에 대한 관심이 최근 들어서 증대되고 있는 실정이다. 선천면역을 담당하는 대표적인 물질로는 lectin, prophenoloxidase activating factor, phagocytosis, antimicrobial peptide 등이 알려져 있으며, 이들은 외부로부터 생체 내로의 이물질 침입에 대한 1차 방어 역할을 담당 한다고 알려져 있다(Relf et al., 1999; Tincu and Taylor, 2004). 최근 들어서 여러 가지의 선천 면역 요소들 중에서 비 특이적이고 빠른 반응성을 가지는 자연 친화적인 항균 펩타이드에 대한 관심이 증가되고 있는 실정이다(Zasloff, 2002).
항균 펩타이드는 유전자에 의해 암호화되어 있는 양친매성의 α-helix나 β-sheet 구조를 취하는 positive net charge를 가지는 12-100개의 아미노산으로 구성된 선천면역에 관여하는 물질로서, 미생물뿐만 아니라 무척추동물에서 척추동물에 이르는 넓은 범위에서 분포하고 있는 단백질성 항균물질이다(van’t Hof et al., 2001; Stark et al., 2002; Izadpanah and Gallo, 2005). 또한 항균 펩타이드는 막을 주요 target site로 삼고 있으므로 내성균 유발의 가능성이 거의 없는 특징을 가지고 있어서 차세대 항생제 개발을 위한 주요 후보물질로서 각광을 받고 있다(Hancock REW and Diamond G, 2000; Zasloff M, 2002).
항균 펩타이드는 구조에 따라서 크게 4가지 종류로 나눌 수 있다(Andreu and Rivas, 1998): i) Cys을 포함하지 않는 linear α-helical peptide, ii) Cys을 포함하는 looped peptide, iii) 특이 구조를 취하지 않는 random extended peptide, iv) 특이 아미노산을 많이 포함한 peptide 등이다. 항균 펩타이드는 이온 결합과 소수성 상호작용을 이용한 barrel-stave model, toroidal model 또는 carpet model을 통해서 membrane pore를 형성하거나 세포내부로 들어가 세포 내 기관이나 대사과정의 저해를 통해서 항균 효과를 나타낸다고 알려져 있다(Shai, 2002; Li, 2009). 현재까지 해양무척추동물 유래 항균 peptide는 갯지렁이(Ovchinnikova et al., 2004), 군소(Iijima et al., 2003), 투구게(Miyata et al., 1989; Saito et al., 1995), 꽃게(Khoo et al., 1999), 흰 다리 새우(Destoumieux et al., 1997), 홍합(Charlet et al., 1996; Mitta et al., 1999; Mitta et al., 2000), 굴(Seo et al., 2005; 2012; 2013; Nam et al., 2015; Gueguen et al., 2006), 전복(De Zoysa et al., 2009) 등이 보고되어 있다.
바지락은 우리나라의 패류산업에서 큰 비중을 차지하는 패류 양식 자원중의 하나로서 중요성이 크게 증대되고 있으나, 최근 들어서 다양한 병원성 미생물 감염원에 의한 대량 폐사가 일어나고 있어서 경제적, 환경적으로 큰 문제가 되고 있다(Park et al., 2006). 따라서 이러한 문제점을 해결하기 위한 기초자료로서 바지락의 host defense mechanism을 이해하고, 질병 감염원의 종류 또는 분포와 바지락의 선천면역 요소들 간의 상호연관성 연구가 절실히 요구되고 있는 실정이다. 특히, 다양한 해양무척추동물에 있어서 선천면역요소 중의 하나인 항균 펩타이드의 질병감염에 대한 효과적인 방어기능이 보고되면서 질병감염에 대한 연구가 활발히 진행되고 있는 반면, 바지락과 연관된 항균 펩타이드에 대한 연구는 극히 제한적으로 진행되고 있는 실정이다. 현재까지 보고된 바지락 유래 항균 펩타이드는 defensin(Zhang et al., 2015), big defensin (Zhao et al., 2010) 또는 mussel defensin MGD-1 유사 peptide (Kang et al., 2006) 등과 같은 defensin 류에 한정되어 있으므로 선천면역을 담당하는 다양한 항균 펩타이드에 대한 연구가 필요한 실정이다.
따라서 본 연구는 바지락(Manila clam,
본 실험에 사용한 바지락(Manila clam,
항균활성 측정을 위한 tryptic soy broth (TSB)와 agarose type I (Low EEO Agar)은 Merck사(Merck, Darmstadt, Germany)와 Sigma사(St. Louis, MO, USA)에서 각각 구입하여 사용하였다. 정제 과정에서 사용된 HPLC용 water와 acetonitrile (CH3CN)은 Tedia사(Ohio, USA)로부터 구입하였고, 그 이외의 모든 시약들은 특급을 사용하였다.
바지락의 패각을 흐르는 수돗물에 씻어서 패각 오염물을 제거한 후, 아가미와 외투막 조직을 취하여 5 mL volume이 될 때까지 얼음 속에서 모았다. 모아진 아가미와 외투막 조직에 끓고 있는 4 배량의 1% acetic acid(v/v)를 첨가하여 100℃에서 5분 동안 끓인 후, 얼음에 보관하여 충분히 냉각 시켰다. 충분히 냉각된 조직은 homogenizer (T10 basic ULTRA-TURRAX, IKA, USA)를 사용하여 완전히 파쇄 시켰다. 조직 파쇄액은 4℃에서 20분 동안 8,000
항균활성 측정에는 그람 양성균인
추출물 또는 정제된 항균물질의 단백질성 확인을 위해서 trypsin 처리 전·후의 항균활성 변화를 확인하였다. 바지락 추출물 또는 정제된 물질 4 μL에 trypsin (1,000 μg/mL) 1 μL를 첨가하고 37℃에서 60분간 반응시킨 뒤, URDA법으로
추출물에 존재하는 독성물질의 유무를 확인하기 위해서 인간의 적혈구에 대한 용혈활성을 측정하였다(Seo et al., 2014). 사람의 혈액(Blood type: B)에 동량의 phosphate buffered saline (PBS, 50 mM sodium phosphate, 150 mM NaCl, pH 7.4)을 넣고 혼합한 후 800 rpm에서 15분간 원심 분리하여 상등액은 버리고 침전물을 얻었다. 이러한 세척과정을 3회 반복한 후 얻어진 적혈구를 1% 혼합용액이 되도록 PBS를 첨가한 후 사용하였다. 얻어진 적혈구 90 μL를 96-well microplate에 도입시키고 바지락 추출물 또는 정제물질 10 μL를 첨가한 다음 37℃에서 60분간 반응시켰다. 이후 반응액을 원심분리(1,500 rpm, 10분, 4℃) 하여 얻어진 상등액 80 μL를 새로운 microtiter plate로 옮긴 후 헤모글로빈 유출 정도를 확인하기 위해서 490 nm에서 흡광도를 측정하였다. 적혈구의 100% 용혈을 위한 비교물질로는 triton X-100 (0.1%)을 사용하였다.
바지락 추출물 내의 양이온성 단백질/펩타이드의 조성분포와 활성 band를 확인하기 위해서 acid-urea PAGE와 bug-blot을
AU-PAGE를 위한 gel은 urea 3.6 g, D.W 3.4 mL, 30% acrylamide/1.6% bis-acrylamide solution 5 mL, glacial HAc 0.65 mL를 비커에 넣고 stirring (10min, RT)과 degassing (20 min, RT, desiccator)을 한 후 TEMED 0.06 mL, 10% APS 0.275 mL를 첨가한 후 stirring (1 min, RT)하고, gel액을 붓고 유출 유무를 확인한 뒤 comb를 꽂은 후 충분히 실온에서 굳혔다. 이후 장치에 5% HAc를 붓고 pre-run (150 v, 60 min, RT)을 하였다. Well에 도입할 각 시료는 20 µL의 2×sample buffer와 1:1(v/v)로 혼합해서 사용하였고, standard는 histone H1 (∼21 kDa), lysozyme (∼11 kDa), aprotinin (∼6.5 kDa) 및 piscidin 1 (∼2.5 kDa)을 포함하는 20 µL의 0.01% HAc와 2×Sample buffer를 1:1 (v/v)로 혼합해서 사용하였다. Pre-run이 끝난 후 running buffer (5% HAC)를 교체하고 well에 시료를 도입시킨 후 전기영동을 실시하였다(150 v, 45 min). 전기영동 후, 1개의 gel은 CBB R-250으로 염색시켰고, 나머지 1개의 gel을 rinse buffer (10 mM PB buffer, pH 7.4)로 2회 세척을 한 후에
>
추출물들의 cytoplasmic membrane permeabilizatin assay
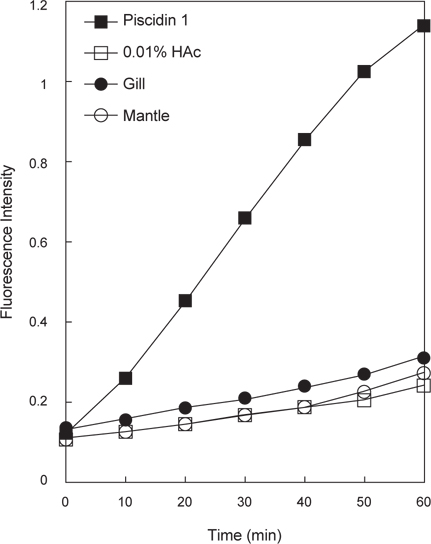
바지락 추출물의 세균 내막 투과성 측정을 위해서 nonmembrane-permeative chromogenic substrate인 o-nitrophenyl-β-D-galactopyranoside (ONPG)에 대한 추출물 도입 후의
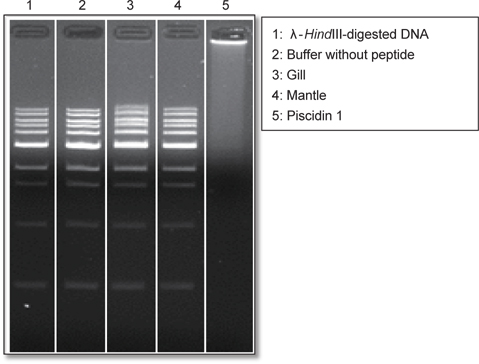
바지락 추출물에 존재하는 염기성의 DNA 결합성 물질의 존재유무를 확인하기 위해서 DNA binding에 의한 DNA band들의 agarose gel-electrophoresis에서의 이동의 저해정도를 확인하는 DNA-binding assay를 수행하였다(Hsu et al., 2003; Alfred et al., 2013). 이를 위해서 1 kb DNA marker (λ-
아가미 추출물에 존재하는 항균물질의 분리 및 정제과정은 HPLC (YL9100 HPLC system, Young Lin Instrument Co., Korea)를 이용하여 수행하였다. 추출물에 존재하는 항균물질의 정제과정은 역상 및 이온 교환성을 갖는 2가지 특성의 HPLC column을 사용하여 수행되었고, 정제과정 중의 분획들에 대한 항균활성은
최종 정제된 물질의 분자량은 Ultraflex III matrix-assisted laser desorption ionisation time-of-flight (MALDI-TOF) mass spectrometer (Bruker Daltonik GmbH, Bremen, Germany)를 사용하여 linear mode로 측정하였다. 정제물질을 0.1% TFA/50% CH3CN (1:1, v/v)에 녹인 후 α-cyano-4- hydroxycinnamic acid (CHCA) matrix solution (10 mg/mL CHCA in 0.1% TFA/50% CH3CN, 1:1, v/v)과 혼합하여 MALDI plate에 도입한 후에 분자량을 측정하였다. Standard로는 Calmix 2를 사용하였다.
정제물의 N-terminal amino acid sequence는 pulse liquid automatic sequencer (model 473A; Applied Biosystems, Foster City, CA, USA)를 사용하여 분석하였다.
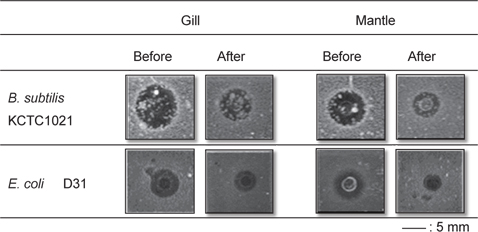
바지락 추출물의 항균활성
바지락(Manila clam,
Acid-urea PAGE와 bug-blot
바지락 추출물내의 염기성 단백질/펩타이드의 구성분포와 그들의 항균활성 유무를 확인하기 위해서 AU-PAGE와 bug-blot를 수행하였다(Fig. 2). 그 결과, 바지락 추출물들은 저분자 물질보다는 상대적으로 고분자 물질의 농도가 높게 나타났으며, 외투막 추출물보다는 아가미 추출물에서 구성물질들의 다양성이 더 높게 나타났다. 또한, 추출물의 항균활성은 slow migration zone (SMZ) (~11 kDa)과 rapid migration zone (RMZ) (~2.5 kDa)을 포함하는 두 위치에서 나타났으며, RMZ의 항균활성이 SMZ보다는 강하게 나타났다. 이러한 결과는 바지락 추출물에는 다양한 분자량의 항균물질이 존재하고 있으며, 아마도 저분자의 항균물질이 보다 중추적인 역할을 담당하는 것으로 판단된다.
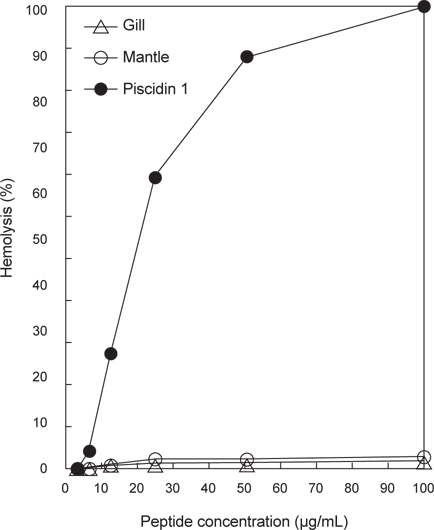
바지락 추출물의 hemolysis
바지락 추출물들에 포함된 독성물질의 유무를 확인하기 위해서 추출물을 인간 적혈구(혈액형: B형)에 대한 용혈활성(hemolytic activity)을 측정하였다(Fig. 3). 비교물질로는 piscidin 1을 사용하였으며 100% hemolysis의 표준물질로는 0.1% TritonX 100을 사용하였다. 측정결과 대조군으로 사용된 항균 펩타이드인 piscidin 1은 낮은 농도(12.5 μg/mL)에서도 강한 용혈활성을 나타낸 반면, 바지락 추출물들은 용혈활성을 거의 나타내지 않았다. 이러한 결과는 바지락 추출물들은 세포독성을 나타내는 물질을 거의 포함하고 있지 않다는 것을 의미하는 것이다.
바지락 추출물에 포함된 항균물질의 작용부위 확인 실험
바지락 추출물에 포함된 항균물질의 target site를 예측하기 위해서 세균 내막 투과성과 핵내 구성물인 DNA와의 상호작용 유무를 관찰하였다. 우선, 항균물질의 target site가 핵 내부의 DNA인지를 확인하기 위해서 DNA-peptide interaction에 의한 DNA migration 저해현상을 관찰할 수 있는 DNA-binding assay를 수행하였다(Fig. 4). 그 결과, 양성 대조군으로 사용한 piscidin 1은 강한 DNA-binding 활성을 나타낸 반면 바지락 추출물들은 DNA migration에 전혀 영향을 주지 않는 것으로 나타났다. 이러한 결과는 바지락 추출물에 존재하는 항균물질은 핵 내의 DNA와의 상호작용에 의해 항균작용을 나타내는 것은 아님을 의미하는 것이다. 두 번째로, 추출물에 포함된 항균물질의 target site가 세포막 자체인지를 확인하기 위해서 membrane permeabilization assay를 수행하였다(Fig. 5). 실험결과 양성 대조군으로 사용한 piscidin 1은 강한 내막 투과성을 나타낸 반면 음성 대조군으로 사용한 1% HAc와 측정에 사용된 바지락 추출물들은 막 투과활성을 거의 나타내지 않았다. 이것은 실험에 사용된 1% HAc는 막 투과활성에 거의 영향이 없음을 나타내는 것이므로 바지락 추출물들에는 막 투과 활성물질이 거의 포함하고 있지 않다는 것을 의미하는 것이다. 이러한 결과는 추출물내의 항균물질의 예상 작용 target site은 막 자체는 아니라는 것을 의미하는 것이다. 두 가지의 실험 결과 바지락 추출물에 포함된 항균물질의 작용기작은 세포막을 직접 공격하거나 핵 내부의 DNA와의 직접적인 상호작용에 의한 것보다는 세포표면 물질들과의 상호작용 또는 세포질에 존재하는 세포 내 구성 성분에 작용할 가능성이 있다는 것을 의미하는 것이다.
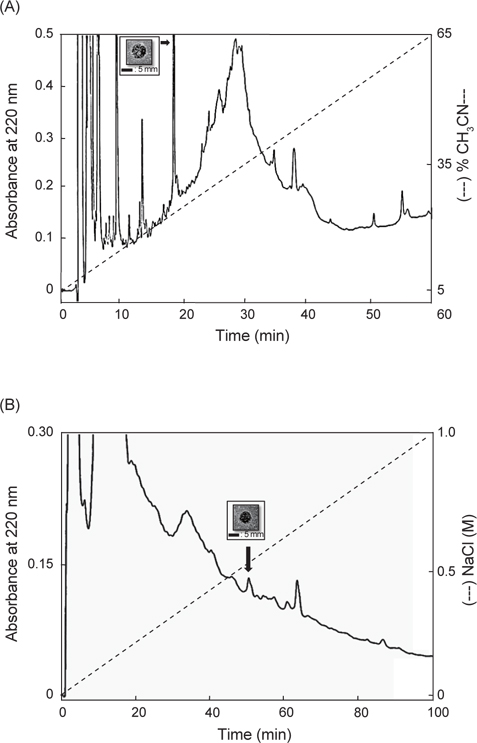
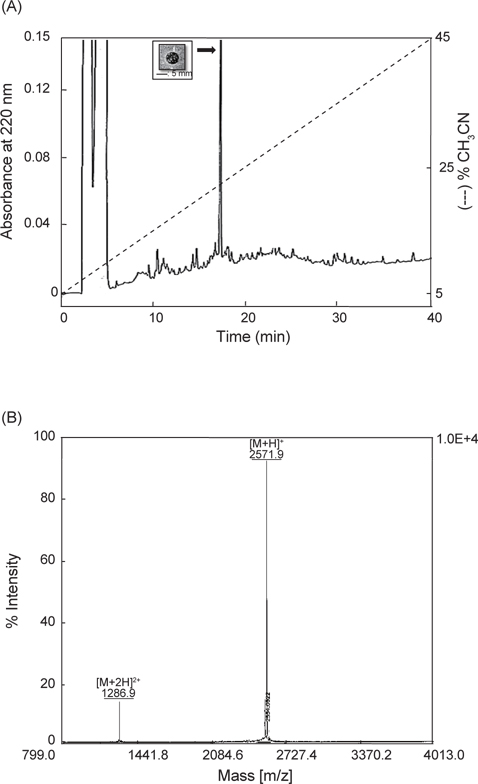
바지락 아가미 추출물로부터 항균물질을 정제하기 위해서 역상 HPLC column 및 이온교환 column을 사용한 정제과정을 수행함으로써 순수한 항균물질을 정제하였다. 첫 번째 정제과정으로 아가미 추출물을 CapCell-Pak C18 column (4.6 mm×250 mm, Shiseido, Japan)에 도입하였으며, 분리된 각각의 분획들은
최종 정제된 항균물질의 분자량은 Ultraflex III matrix-assisted laser desorption ionisation time-of-flight (MALDI-TOF) mass spectrometer (Bruker Daltonik GmbH, Bremen, Germany)를 이용하여 linear mode로서 측정하였다(Fig. 7B). 분자량 측정을 위한 matrix로는 CHCA (ɑ-cyano-4-hydroxycinamic acid)를 사용하였으며, standard로는 calmix 2를 사용하였다. 분자량을 측정한 결과, 정제된 물질의 singly charged species는 2571.9 [M+H]+와doubly charged species는 1,286.9 [M+2H]2+의 mass data가 얻어졌으며, 이를 바탕으로 정제 물질의 분자량은 2,571.9 Da으로 확인되었다. 이러한 결과를 바탕으로 정제 물질의 일차구조를 분석하기 위해서 edman degradation을 통한 N-말단 서열분석을 수행하였으나 구성 아미노산에 대한 분석 결과를 얻지 못하였다. 이것은 아마도 정제된 항균물질의 N-말단이 번역 후 변형(post-translational modification) 과정에 의한 specific functional group으로 blocking된 것에 기인한 것으로 판단된다. 일반적으로 척추 또는 무척추동물 유래의 많은 항균 펩타이드들에서 분해효소에 대한 저항성 강화 등의 목적으로 번역 후 변형과정을 통해서 N-말단이 blocking되어 있는 경우가 자주 보고되고 있다(Andreu and Rivas, 1998). 본 연구에서 정제된 단백질성 항균물질의 N-terminal blocked group의 확인과 완전한 일차구조의 규명을 위한 추가적인 연구과정이 수행되어야 할 것으로 판단된다.
본 연구의 결과 바지락의 아가미와 외투막에는 다양한 단백질성 항균물질들이 포함되어 있는 것으로 확인되었으며 특히, 아가미 추출물에서 분자량이 2571.9 Da의 단백질성 항균물질의 존재를 확인할 수 있었다. 이러한 결과는 바지락의 선천면역에 대한 이해를 증대시킬 수 있는 기초자료로 활용될 수 있을 것으로 판단된다.