조피볼락(
분자생물학적 종 분류 및 동정을 위한 유전자 마커로는 ribosomal RNA (rRNA), mitochondrial DNA (mtDNA) 및 microsatellite DNA등이 일반적으로 사용된다. 진핵생물의 단백질 합성에 있어 중추역할을 하는 ribosomal DNA는 유전자의 계통발생학적 분석에 사용되는 rRNA를 code하는 분자 마커로써 18S, 5.8S 및 28S 유전자와 internal 및 external transcribed spacers (ITS and ETS)로 구성되어 있다. 이중 18S rRNA는 유전자의 변이가 심하지 않기에 다양한 생물군의 분류에 사용되고 있다(Nyaku et al., 2013). mtDNA는 고변이성 유전자로서 종의 진화적 유연관계를 나타내고 있으며, 그 중 cytochrome c oxidase subunit I (COI)는 다양한 해양 무척추동물들의 유전학적 연구에 사용되는 유전자 마커이다(Edmands et al., 1996). Cytochrome 은 생체세포 내에 존재하는 헴단백질 색소 중 하나로서 고등동식물의 세포, 세균, 곰팡이 및 효모 등의 미토콘드리아에 존재한다. Cytochrome은 철 포르피린의 구조, 단백질과의 결합 상태에 따라 a, b, c 및 d의 4군으로 구별되는데, 이 중에서 cytochrome c는 산화환원반응에 관여하는 효소로서 미토콘드리아의 외막에 존재하며 호흡과 순환에 의해 세포내에 운반된 산소를 활성화하여 세포호흡을 원활하게 하는 데 필요하다. Cytochrome c oxidase는 cytochrome c 에서 전자를 직접 분자상 산소로 넘겨주는 효소활성을 하는 호흡효소를 뜻하는데 동물군은 미토콘드리아의 COI를 표준바코드 유전자로 이용하여 종 동정에 사용하기도 한다. Herbert (2003)는 DNA barcoding 기법이 유전자 서열을 이용하여 종을 동정하는 방법으로서 적당하며 동물의 동정으로 미토콘드리아의 COI 유전자 서열을 사용할 것을 제안하기도 하였으며, 최근의 다른 연구에서는 미토콘드리아 COI 유전자 염기서열의 다형성을 바탕으로 어류간 계통 유연관계를 확인하기도 하였다(Han et al., 2014).
따라서 본 연구에서는 선충의 18S rRNA 및 COI 유전자를 증폭시킬 수 있는 primer를 사용하여 선충 유전자 분석을 실시하였으며, 획득한 염기서열을 바탕으로 조피볼락에서 분리된 선충의 계통발생학적 분석을 실시하였다. 또한 어류 조직 내에 위치한 선충 자충의 분석 및 중간숙주의 탐색에 사용하기 위하여 18S rRNA 유전자를 증폭시킬 수 있는 primer를 제작하고, 선충을 분자생물학적으로 검출할 수 있는 PCR법을 개발하였다.
2014년도 1월, 서해안 천수만 일대에 양식중인 조피볼락에서 선충을 분리 후 100% ET-OH에 고정하여 보관한 시료를 DNA 추출을 위해 사용하였다. 50 mg의 선충을 E-tube에 넣고 PBS를 사용하여 마쇄하였고, 원심분리(8,000 rpm, 10 min) 후 상청액을 회수하였다. DNA 추출은 High pure template preparation kit(Roche, Germany)을 사용하였으며, 제조사의 매뉴얼에 따라 수행하였다.
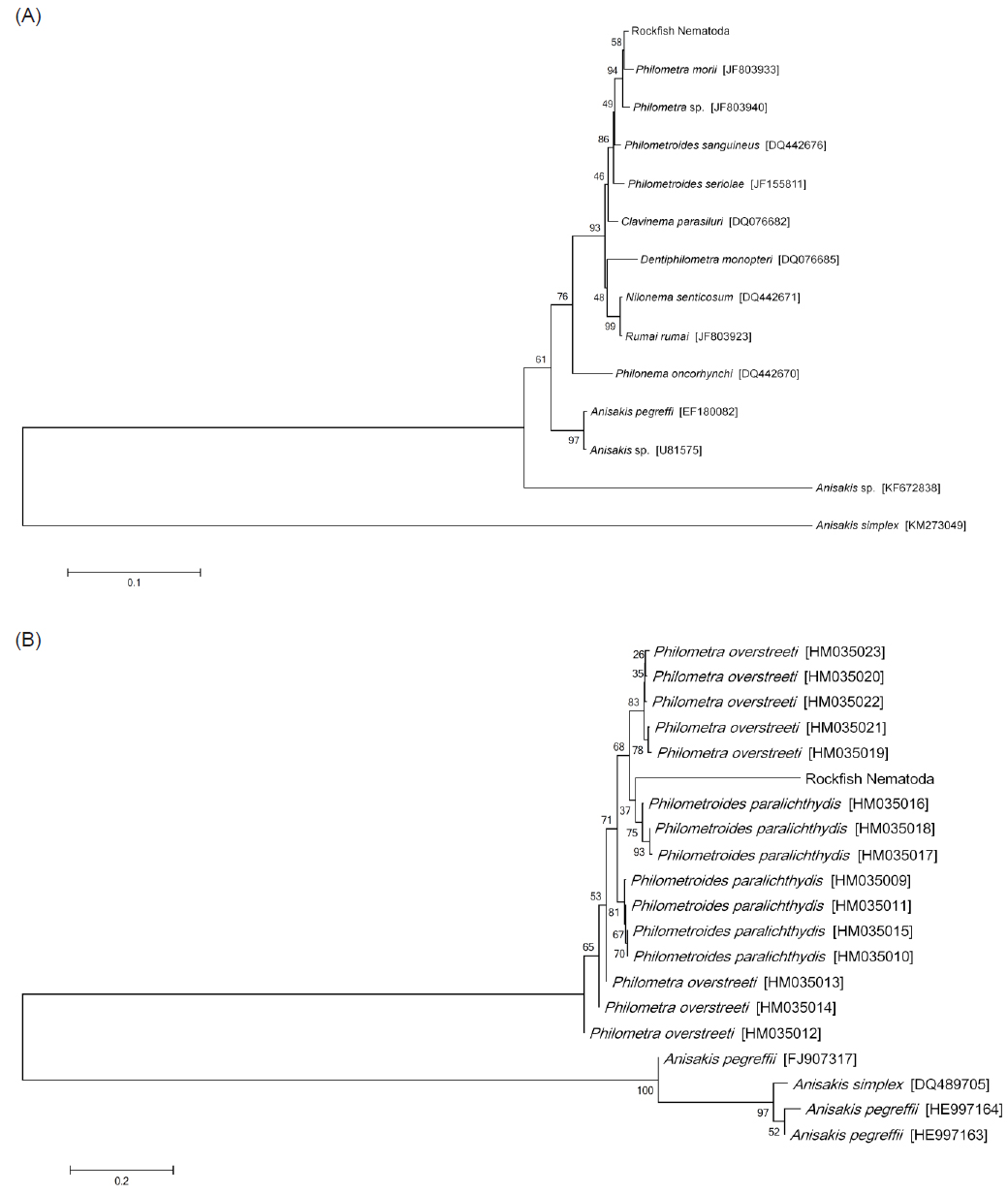
선충의 종 동정을 위하여 18S rRNA와 COI 유전자를 분석하였다. PCR법은 Emerald Taq (Takara, Japan)을 사용하여 제조사의 매뉴얼에 따라 수행하였으며, 각 primer sequence와 PCR 조건은 Table 1에 표기하였다. PCR product는 1.5% agarose gel (Bioneer, Korea) 상에서 전기영동을 실시하여 band를 확인하였으며, PCR 증폭이 확인된 시료는 sequencing을 실시하여 염기서열을 확인하였다. 염기서열의 분석에는 BioEdit Ver. 7.2.3, MEGA Ver. 6를 사용하였으며, National Center for Biotechnology Information (NCBI)에서 제공되는 Basic Local Alignment Search Tool (BLAST)을 이용하여 기존에 보고된 선충 유전자와의 상동성 분석을 하였다. 또한 얻어진 염기서열을 바탕으로 실시한 유전자 계통분석은 BioEdit Ver. 7.2.3 program을 사용한 염기서열 정렬과 MEGA Ver. 6 program을 사용한 근린결합분석(NJ; neighbor-joining analysis, 1000 rounds of bootstrap)을 통하여 각 염기서열간의 유전적 거리와 계통도(phylogenetic tree)를 획득하였다. 본 연구에서 비교한 18S rRNA (1,695 bp) 염기서열은GenBank에 등록되어있는 Philometridae에 속하는 8종의 선충 18S rRNA 유전자(
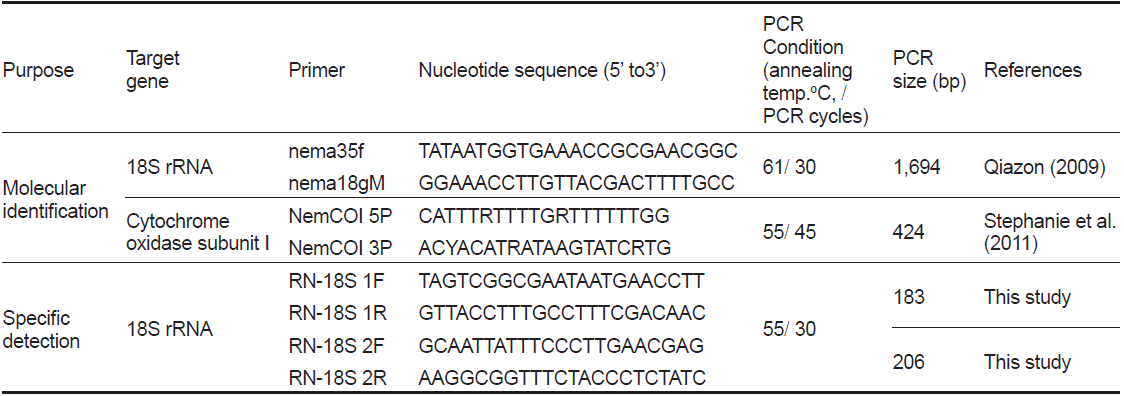
[Table 1.] Primer information and PCR condition for this study
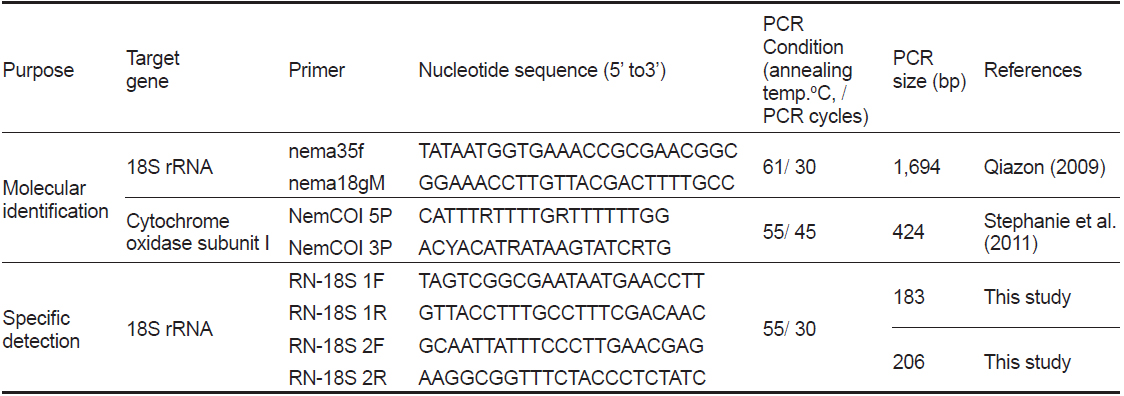
Primer information and PCR condition for this study
위 연구에서 얻어진 조피볼락 선충의 18S rRNA 염기서열 중 다른 Philometridae 의 18S rRNA와 염기서열의 차이가 보이는 특이적인 영역에서 2개의 primer set를 제작하여 선충 검출을 위한 PCR법 개발에 사용하였다. PCR조건은 Table 1에 표기하였으며, PCR annealing 온도에 따른 검출률도 확인 하였다. 또한 조피볼락의 선충 검출에 사용된 primer의 특이성을 검증하기 위하여 선충을 포함한 다른 기생충 및 세균의 DNA를 추출한 뒤 PCR을 실시하였다. 실험에 사용된 기생충은 조피볼락에서 분리된 선충 이외에 어류에서 분리된 스쿠티카, 쿠도아 및 아니사키스 충을 사용하였으며, 세균은 어류에서 분리된
넙치에서 유래된 스쿠티카충은 보유중인 CHSE-214 어류주화세포에서 배양중인 신선한 충을 사용하였으며 쿠도아충은 넙치의 근육에서 분리된 것을 사용하였다. 아니사키스충은 조피볼락과 넙치의 내장에서 각각 분리된 후 100% ET-OH에 고정된 것을 사용하였으며, 각각의 기생충은 상법과 동일하게 DNA를 추출한 뒤 PCR에 사용하였다. 또한 각 세균은 1.5% NaCl이 포함된 Brain Heart Infusion (BHI, Gibco, USA) broth 배지에서 25℃에서 12시간 동안 배양된 신선한 균을 사용하여 상법과 동일하게 DNA를 추출한 뒤 PCR에 사용하였다. PCR product는 1.5% agarose gel (Bioneer, Korea) 상에서 전기영동을 실시하여 band를 확인하였다.
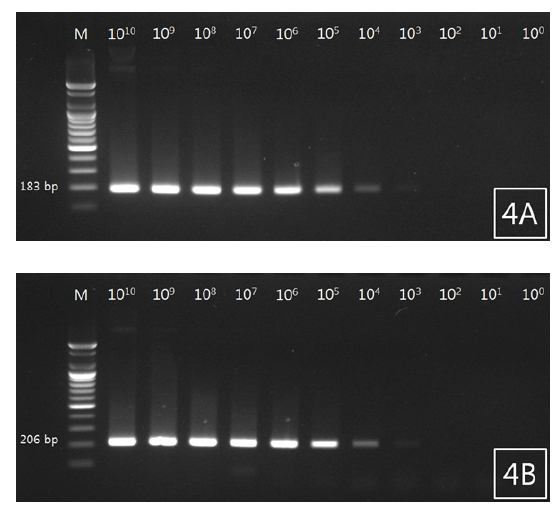
선충 검출 PCR primer의 감도 확인실험은 다음과 같은 방법으로 실시하였다. 조피볼락에서 분리된 선충의 PCR product는 1.5% agarose gel 상에서 전기영동을 실시한 뒤, PCR product purification kit (Roche, Germany)를 사용하여 Gel Purification을 실시하였다. 정제된 PCR product는 pGEM TEasy vector (Promega, USA)을 사용하여 cloning을 실시하였고, Plasmid Mini Extraction kit (Bioneer, Korea)를 사용하여 plasmid DNA를 획득하였다. 선충의 plasmid는 copy수를 계산한 뒤 Nuclease-free water (Bioneer, Korea)를 사용하여 1×1010copy부터 1×100copy까지 10배씩 단계희석 하였고 이를 template로 하여 PCR을 실시하였다. PCR product는 1.5% agarose gel 상에서 전기영동을 실시하여 band를 확인하였다.
본 연구에서는 조피볼락에서 분리된 선충의 분자생물학적 동정을 실시하기 위하여 선충류의 18S rRNA 및 COI 유전자를 증폭할 수 있는 universal primer를 각각 사용하여 PCR과 유전자 분석을 실시하였다. 18S rRNA의 유전자를 증폭하기 위하여 Quiazon (2009)의 논문에 인용된 PCR조건을 사용하였으나, PCR 반응이 일어나지 않아PCR 조건을 수정하여 설정하였다(Table 1). 조피볼락 선충의 genomic DNA를 template로 사용하여18S rRNA의 유전자를 증폭한 결과 1,695 bp의 염기서열을 획득하였으며(GenBank accession no. LC071530), 해당 염기서열에 대한 NCBI BLAST 결과
하지만 조피볼락 선충 COI 유전자(PCR product size 389bp; GenBank accession no. LC073304)의 경우 NCBI BLAST 결과 소나무에서 분리되는 선충류인
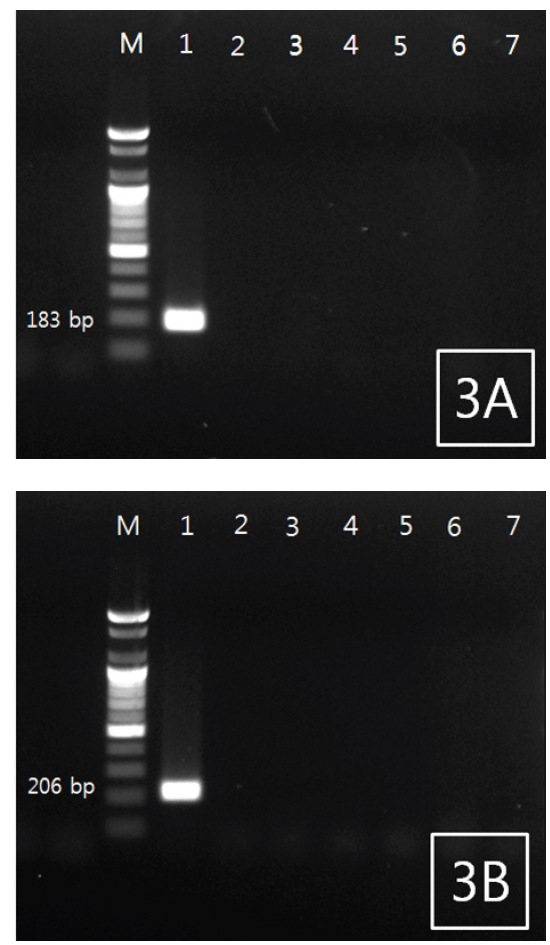
조피볼락 선충을 특이적으로 검출하기 위한 PCR법을 위하여 18S rRNA 염기서열 중에서 2쌍의 primer set를 새로 디자인하였다(Fig. 1). 조피볼락 선충 검출 Primer 제작을 위해서, 조피볼락 선충의 18S rRNA와 상동성이 비교적 높은 다른 어종에서 분리되는 Philometrodae 기생충의 18S rRNA유전자 염기서열을 비교하여 조피볼락 선충 특이 primer 제작에 활용하였다. 선충의 DNA를 추출한 뒤 PCR을 실시한 결과 2종의 primer set 모두에서 양성을 나타내었으며, agarose gel 상에서 183 및 206 bp의 band를 각각 나타내었다. 추가적으로 선충 유전자의 검출을 위한 PCR 조건의 최적화를 위하여 BIO-RAD 회사의 Peltier Thermal Cycler (PTC-0220)을 사용하여 gradient PCR을 실시하였다. 조절된 annealing temp는 42-62℃이었으며 2종의 primer 모두 55℃에서 최적의 조건을 나타내었다(data not shown).
조피볼락 선충 특이적 primer의 검증을 위하여 5종의 기생충(조피볼락 유래 선충 및 아니사키스충, 넙치 유래 스쿠티카충, 아니사키스충 및 쿠도아충)과 2종의 세균(
조피볼락 선충 검출용 primer를 사용하여 선충 유전자가 증폭된 plasmid DNA를 제작하였고, 검출감도를 확인하였다. 그 결과 18S rRNA plasmid DNA는 1×1010copy부터 1×103copy까지 band가 확인되었으며 두 primer에서 비슷한 검출감도를 확인할 수 있었다(Fig. 4A, 4B).
Philometrid nematodes의 생활사에서 자충은 중간숙주에 의해 먹히게 되고, 중간숙주를 어류가 먹어서 종숙주에 감염이 된다. 중간숙주는 대개 요각류(copepodes)로서 유영생활을 하며 어류의 먹이가 된다(Wang, 2002). 체내에 새끼를 가지고 있는 암컷 선충의 경우 숙주의 근육을 통해 피부를 관통하여 구멍을 생성한다. 그 상처를 통하여 자충을 수계로 배출하며, 그 자충을 중간숙주가 섭취하며 최종적으로 어류에 감염이 된다고 한다(Gonzalez et al., 2007). Seo et al. (2014)은 2013년 8월, 조피볼락의 피부에서 선충이 감염된 후 어체 외부로 빠져나가면서 생긴 다수의 상처를 발견하였다. 또한 2013년 10월, 선충의 감염이 확인되지 않았기에 선충은 1년 주기의 생활사를 가지며, 고수온기 어체의 피부에 상처를 내어 자충을 방출한다고 생각된다. 선행연구와 마찬가지로 성충의 경우에 육안으로 검사가 가능한 선충은 자충의 경우 육안으로 확인이 불가능하며 이는 중간숙주도 마찬가지이다. Seo et al. (2014)에 의하면 선충류의 특성상 숙주로부터 선충을 감염이후에 치료는 매우 어려우므로, 생활사 중 선충의 감염을 차단하여야 하며, 선충의 중간숙주를 탐색하여 감염경로를 제거하는 방법에 대한 연구가 필요하다고 하였다. 따라서 본 연구에서 개발한 선충의 유전자 검출을 위한 PCR법을 사용한다면 어류 내 또는 생사료 등에 존재 가능한 자충을 포함한 중간숙주의 탐색이 가능하며 생활사의 규명에 도움이 될 것이라 생각한다.
본 연구에서는 양식 조피볼락에서 분리된 선충의18S rRNA 유전자 분석결과