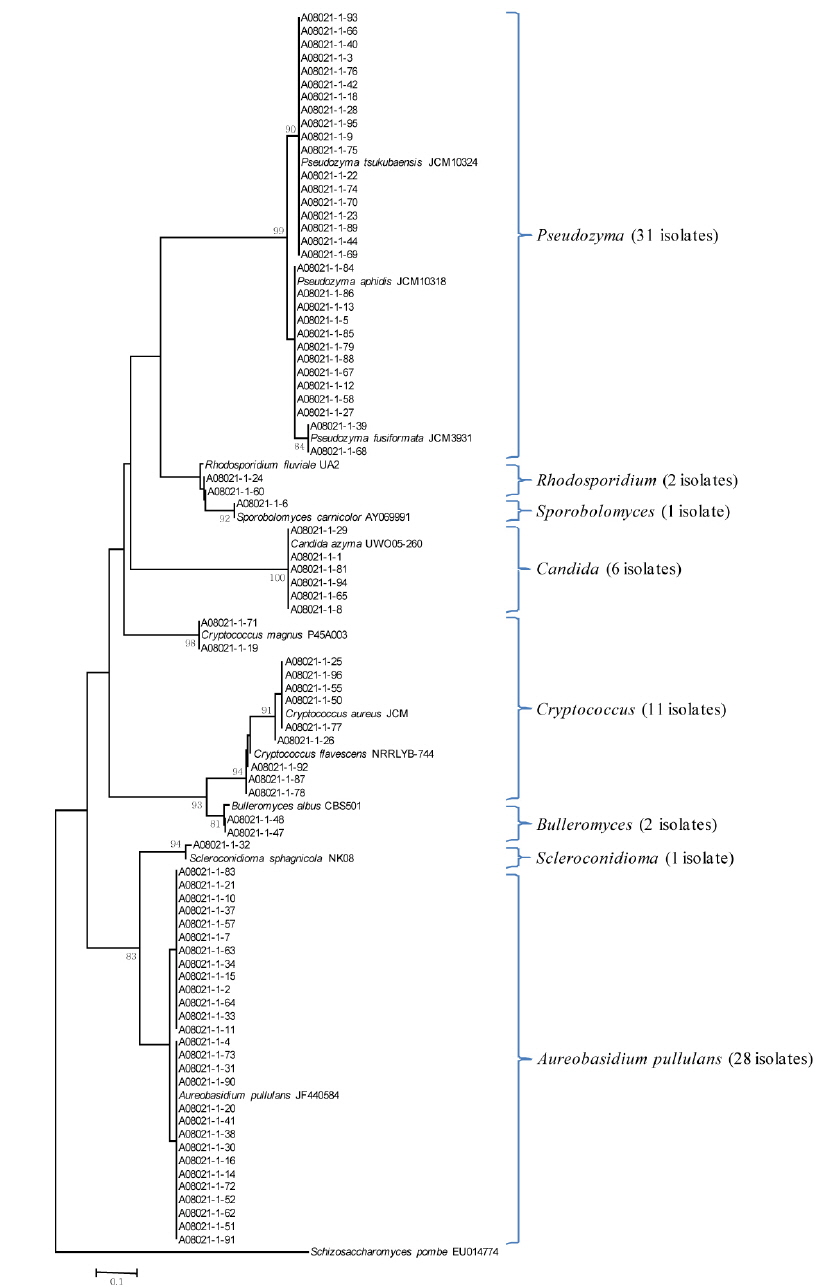
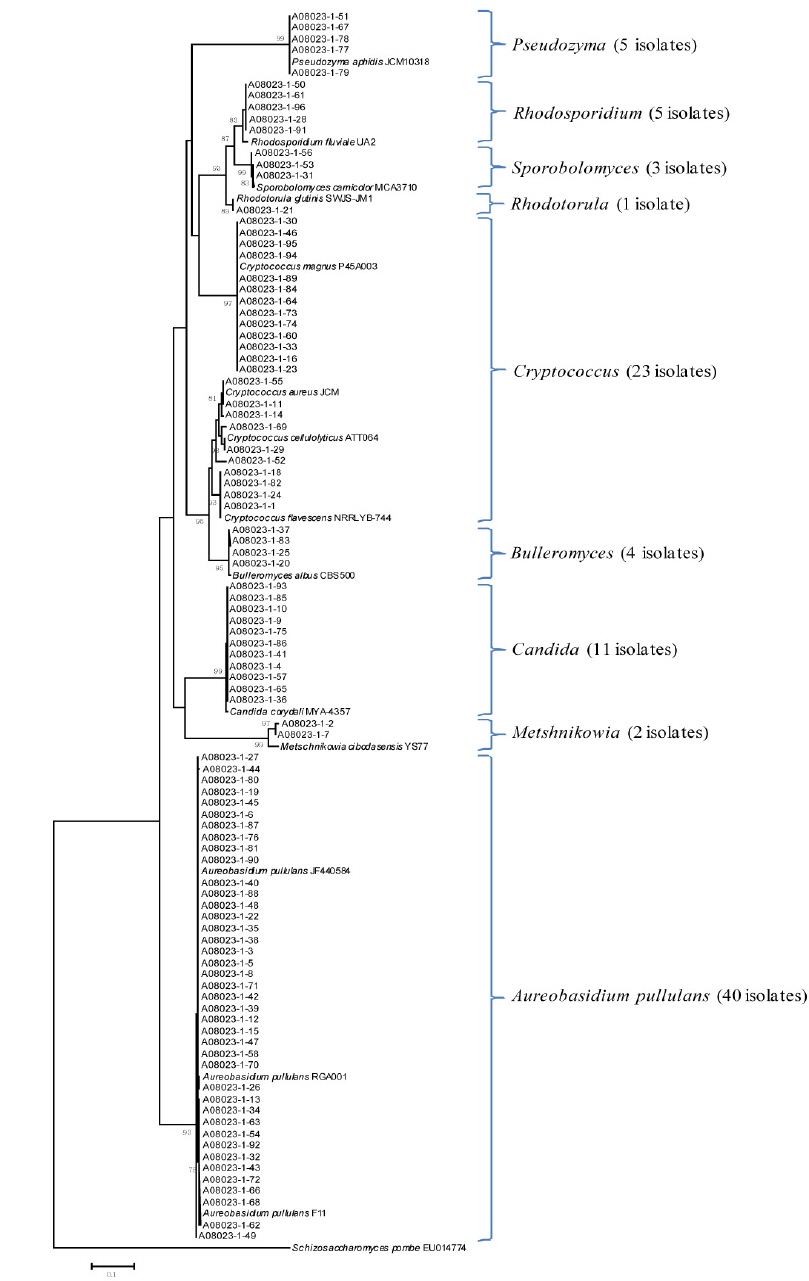
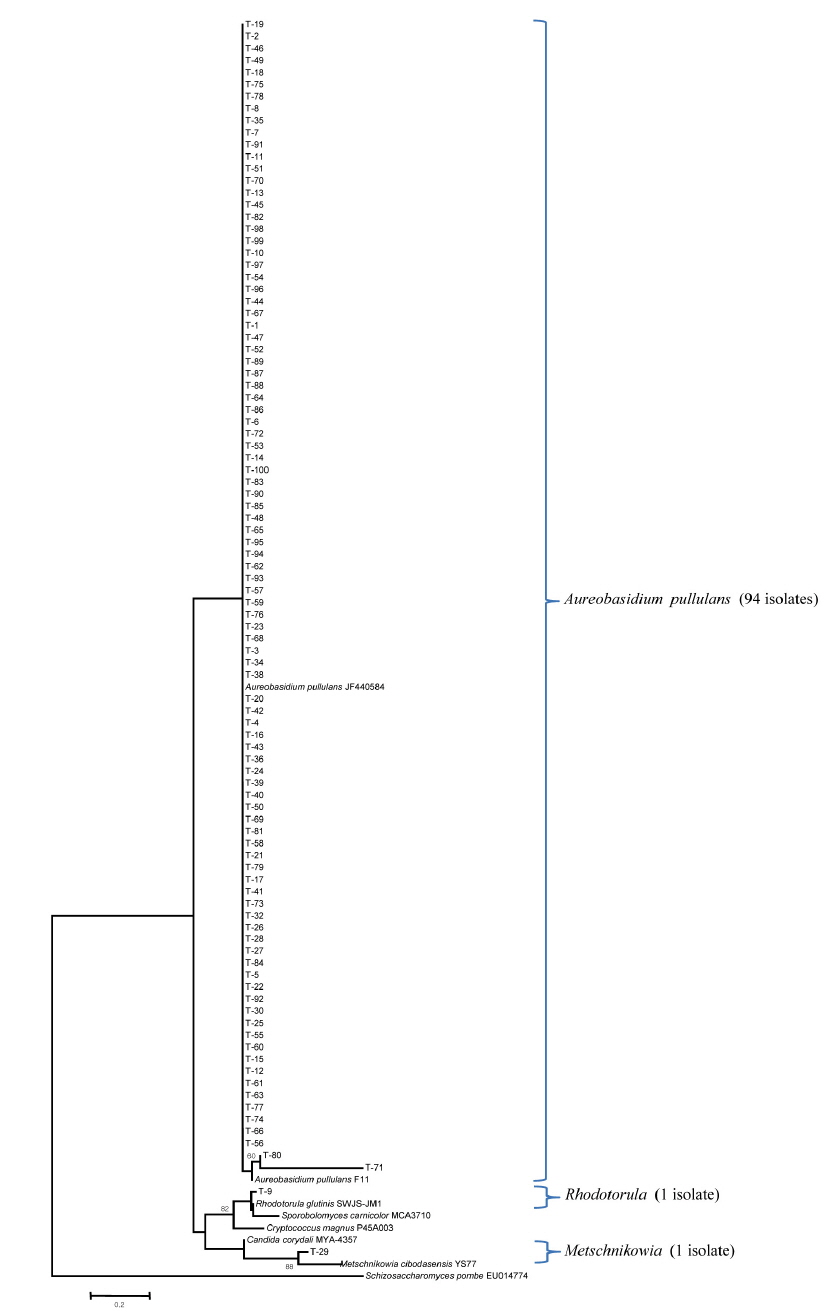
Yeast isolates associated with the leaves, stems, and flowers of the tiger lily needed to be identified using isolation methods that have previously been used effectively in yeast biotechnology. A culture-based approach was necessary for the isolation of many yeast strains associated with tiger lily.
In this study, the homogenized leaves, stems, and flowers of tiger lily were spreaded onto GPY medium containing chloramphenicol, streptomycin, Triton X-100, and L-sorbose. A total of 82 yeast strains from the leaves, 94 and 97 yeast strains from the stems and flowers were isolated, respectively. Yeast isolates were identified by phylogenetic analysis based on internal transcribed spacer region sequencing. The yeast species isolated from the leaves comprised of 31 isolates of the genus
We identified site-specific yeast communities associated with tiger lily. These yeast isolates may have high potential for application in the field of biotechnology.
참나리는 백합과(
효모는 일반적으로 토양, 수생환경, 식물잎, 곤충, 극한 환경 등의 다양한 환경에서 서식하는 것으로 알려져 있으며 (Botha, 2011; Fonseca and Inacio, 2006; Raspor and Zupan, 2006), 약품, 주류, 효소, 음료, 제빵, 농업 및 산업에 중요한 영향을 미치는 등, 인류의 식생활에 많은 이익을 주고 있다(Deak, 2009; Tamang and Fleet, 2009). 이와 같은 효모들을 대량으로 분리하기 위해서 선행연구에서 세균과 곰팡이의 배양을 억제하고 효모만을 분리하는 기술을 알로에 잎에 정착하는 효모를 분석하여 확인하였다(Choi
참나리로부터 효모의 분석은 거의 연구되지 않았으며 그 결과가 유용할 것이며, 효모 바이오테크놀로지에의 기초자료로서 활용될 것으로 사료된다. 따라서 본 연구에서는 참나리 잎과 줄기, 꽃으로부터 기존에 확립된 효모 분석법을 이용하여 효모 군집 분석을 수행하여 보고하는 바이다.
본 연구에 사용된 참나리는 2011년 7월에 울진(N 36° 59' 54.49" and E 129° 24' 17.28")에서 채취하였다. 채취된 참나리는 크린벤치내에서 멸균된 가위와 핀셋을 사용하여 각각 잎과 꽃, 줄기 세 부분으로 나누어 효모 분리를 위한 시료로 준비하였다. 각 부위별 시료는 0.1 M potassium phosphate buffer를 사용하여 3회 washing 하여 멸균된 용기에 실험 전까지 보관하였다. 효모 분리용 배지는 GPY agar (4% glucose, 0.5% peptone, 0.5% yeast extract, 1.5% agar) 를 이용하여 배양하였다. 특히 효모의 생육에 영향을 미치지 않고 세균의 억제를 위해서 Choi
먼저 GPY 고체배지를 멸균하여, 245×245×25 mm 크기의 사각 플레이트(Nunc Bio-Assay Dish, Thermo Scientific, Roskilde, Denmark)에 분주하여 고체배지를 준비하였다. 각 부위별 시료를 파쇄하기에 알맞은 크기로 잘게 절제한 다음 멸균된 용기에 넣고 최종 볼륨이 10 mL이 되도록 10 mM potassium phosphate buffer를 첨가하여 준 후 멸균된 homogenizer (T10 basis, IKA, Germany)를 이용하여 파쇄하였다. 각 부위별 파쇄된 참나리 원액 1 mL를 미리 준비한 배지에 도말한 후 25℃에서 2~5 일간 배양하여 효모를 분리하였다.
>
ITS (internal transcribed spacer) 영역의 염기서열 분석 및 분자계통학적 분석
염기서열은 Choi
본 연구에서는 유용 효모의 바이오테크놀로지 응용을 위하여 다수의 야생효모를 분리하였다. 이를 위해서 참나리에 서식하는 효모 균주를 분리하고 계통분류학적인 분석을 실시하였다. 그 결과, 참나리 잎에서는 82 균주, 줄기에서 94 균주, 꽃에서는 97 균주를 각각 분리하였다. ITS 유전자의 염기서열 분석을 통한 계통학적 분석 결과, 참나리 잎에서는 82 균주가
Fig. 3에서 보는 바와 같이 참나리 꽃에 정착하는 효모의 군집이 극히 단순한데, 이는 본 연구에서 처음으로 밝히는 사실이다. 특히 잎에서는 8 종류의 효모가 그리고 줄기에서는 9 종류의 효모가, 꽃에서는 3 종류의 효모가 정착하고 있어 효모 군집 구조 분석에 대해서는 앞으로 더 많은 연구가 요구된다. 특히 유용 알로에인
이들에 대한 보다 많은 정보가 축적되었을 때 참나리 또는 대상 식물 고유의 기능과 정착하고 있는 효모 기능의 상호관계의 규명 또는 그 기능의 바이오테크놀로지에의 활용이 가능할 것이다.
효모의 유용 기능을 탐색하기 위해서 참나리에 정착하는 효모 군집을 분석하였다. 본 연구에서는 잎에서 총 82 균주, 줄기에서 총 94 균주, 꽃에서는 총 97 균주를 분리하였다. 분리된 균주를 ITS 1과 4 primer를 사용하여 ITS 영역 염기서열의 계통분석을 실시한 결과, 참나리 잎에서는