Sorghum (
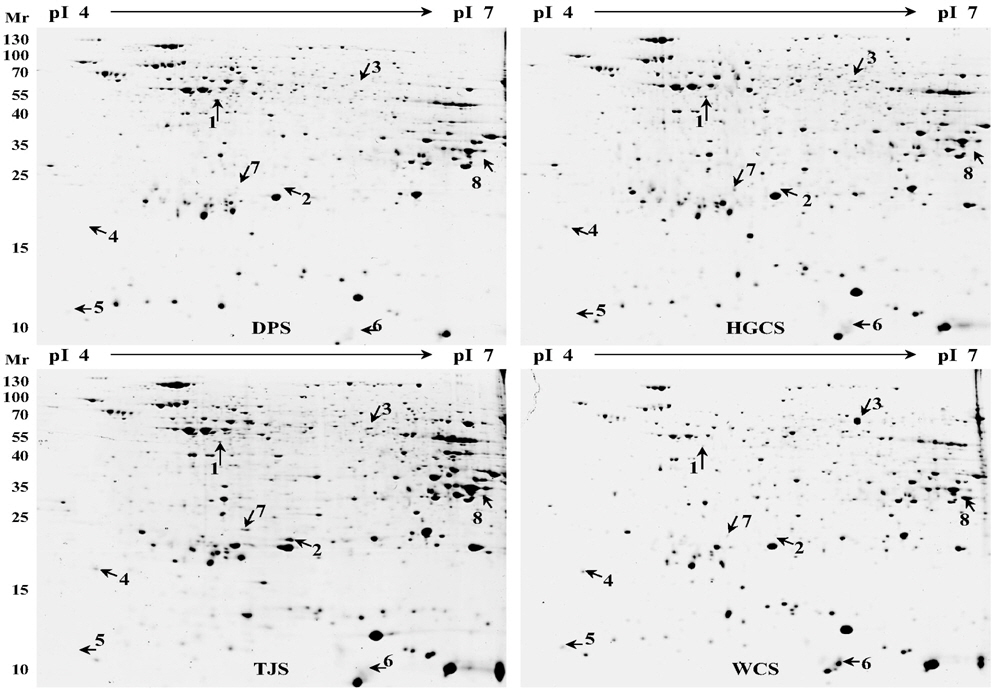
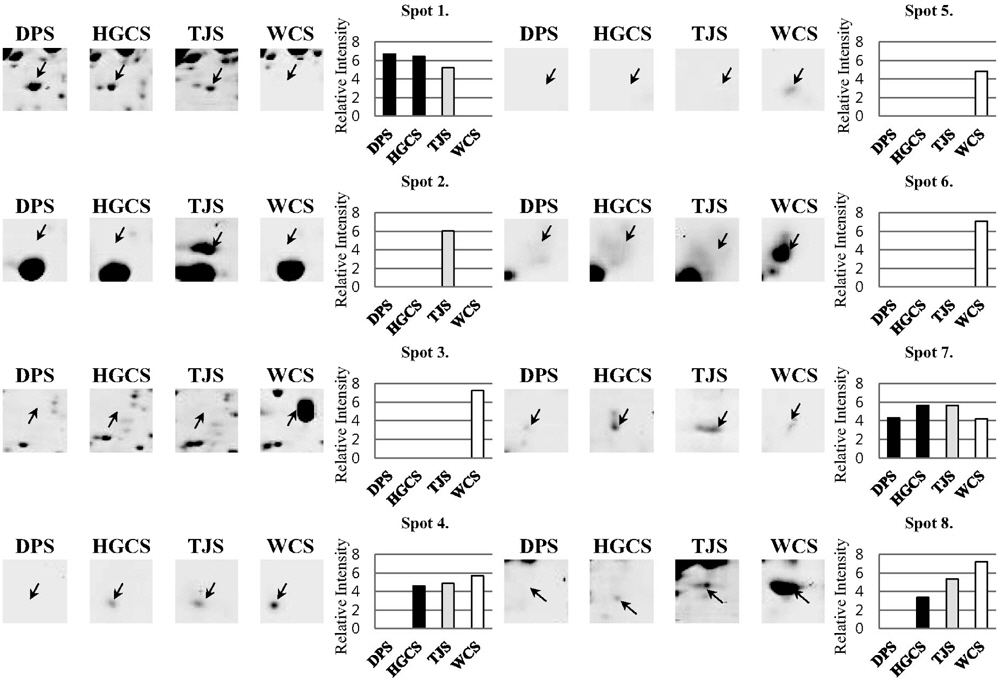
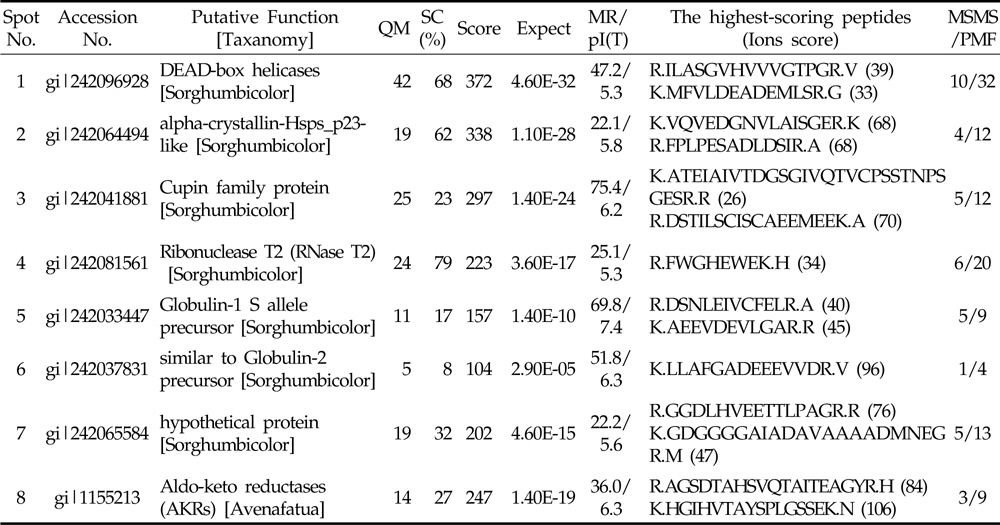
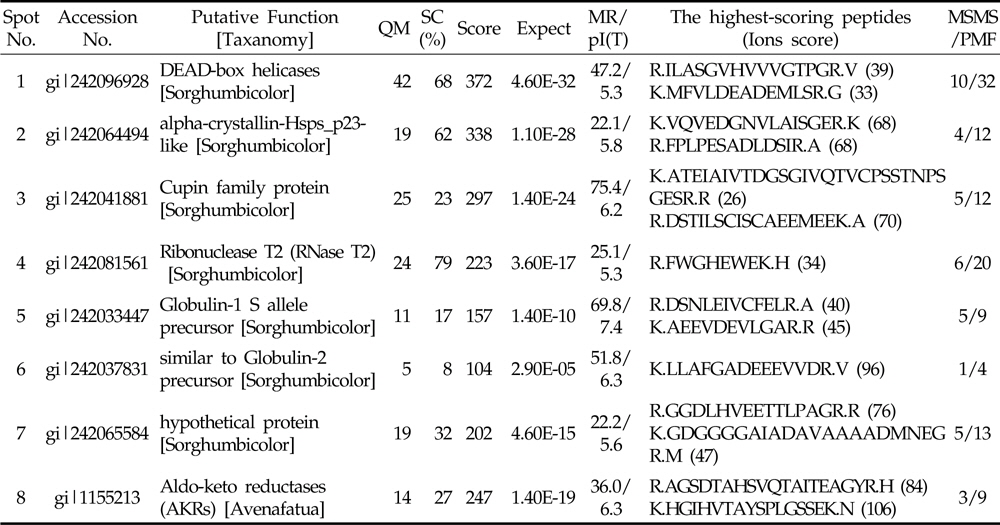
Proteins were extracted from sorghum seed, and separated by 2-DE. Total 652 spots were detected from 4 different sorghum seed after staining of 2-DE with colloidal Coomassie brilliant blue (CBB). Among them, 8 spots were differentially expressed and were identified using MALDI-TOF/TOF mass spectrometry. They were involved in RNA metabolism (spot1, spot 4), heat shock proteins (HSPs, spot 2), storage proteins (spot 3, spot 5, and spot 6), and redox related proteins (spot 8). Eight of these proteins were highly up-regulated in Whinchalsusu (WCS). The HSPs, Cupin family protein, and Globulin were specifically accumulated in WCS. The DEAD-box helicase was expressed in 3 cultivars except for WCS. Ribonuclease T2 and aldo-keto reductase were only expressed in 3 cultivars except for Daepung-susu (DPS).
Functions of identified proteins were mainly involved in RNA metabolism, heat shock protein (HSP), and redox related protein. Thus, they may provide new insight into a better understanding of the charactreization between the cultivars of sorghum.
수수 (
프로테오믹스는 단백질 수준에서 유전자 발현의 특성을 파악하여, 단백질 발현에 대한 정보 획득 및 그 발현 단백질의 기능 해명에 있어 유용한 기술이라고 할 수 있다 (Pandey and Mann, 2000). 이러한 프로테오믹스적 접근법은 이차원 전기영동 (2‐DE) 분석으로 특정 조건에 따라 다르게 발현되는 단백질을 등전점 (pI)과 분자량에 따라 분리한 후 각각의 단백질을 질량 분석기를 이용하여 아미노산 서열을 결정하고 이를 바탕으로 단백질이나 genome database를 bioinformatics적 분석을 수행하여 단백질을 동정하는 과정이다 (Rabilloud, 2002).
현재까지 수수 단백질체에 대한 분석 연구는 미미한 실정이다. 최근 Kumar 등 (2011)은 salt stress에 반응하는 수수단백질에 대한 프로테옴 분석을 수행하였다. Ndimba 등 (2010)은 salinity stress 하에 발현되는 heat shock protein 70 (HSPs70)에 대한 프로테옴 분석을 수행하여 보고하였다.
본 연구는 수수 품종에서 품종 별로 발현 정도에 차이와 품종 특이적으로 발현되는 단백질들을 2‐DE로 분리한 후, MALDI‐TOF/TOF MS를 이용하여 동정함으로써 수수 종자의 품종 간 차이를 보이는 단백질을 확보하고 이들 단백질을 이용하여 수수의 기능성 강화 및 품종 판별 기술 개발을 위한 유용 유전자를 확보하고자 수행하였다.
본 실험에서 사용된 수수는 대풍수수 (DPS), 황금찰수수(HGCS), 흰찰수수 (WCS), 토종수수 (TJS) 등 4 품종으로, 사용된 수수 종자는 농촌진흥청 국립식량과학원 기능성 작물부에서 분양 받았다.
1 g (약 5 립)의 종자에 액체 질소를 부어 완전히 파쇄한 후, 파쇄된 가루에 Mg/NP‐40 단백질 추출용액 [0.5 M Tris ‐HCl, 2% Nonidet P (NP)‐40, 20 mM MgCl2] 20 ml을 첨가시켜 균질화 시키고 나서 12,000 rpm으로 4℃에서 10분 간 원심분리를 수행하여 상등액만 분리하였다. 추출한 단백질에 물로 포화된 phenol 10 ml을 첨가 하여 현탁 시킨 후 원심 분리하여 phenol 층을 취하고 0.1 M ammonium acetate를 포함한 methanol을 phenol 층 부피의 4배 양으로 첨가하여 균질화 후 1 시간 동안 ‐20℃ 에서 침전시켰다. 침전 후 4℃에서 12,000 rpm으로 5 분 간 원심 분리하여 얻은 침전물에 0.1 M ammonium acetate를 포함한 methanol 10 ml을 첨가하여 현탁 후 침전물을 얻는 방법과 같은 조건으로 다시 원심 분리하는 세척 과정을 세 번 반복하고, 80% acetone으로 두 번 더 세척하였다. 마지막으로 80% acetone 1 ml을 첨가하여 ‐20℃에서 추출 된 단백질을 보관하였다 (Kim
추출된 단백질은 2‐DE Quant Kit (GE Healthcare, Piscataway, NJ, USA)를 사용하여 정량 분석하였다. 80% acetone에 침전 되어 있는 단백질을 원심 분리를 수행하여 acetone을 완전히 제거하고, rehydration 용액 [7 M Urea, 2 M Thiourea, 4% (v/v) CHAPS, 2 M dithiothreitol (DTT), 0.5% (v/v) IPG buffer pH 4‐7 (GE Health, Waukesha, WI, USA)]에 녹인 후, 2 분간 원심 분리를 수행하였다. 500 μl precipitant (GE Healthcare, Piscataway, NJ, USA)를 첨가하여 섞어 준 뒤, 상온에 2‐3 분 간 놓아 둔 후 500 μl co‐precipitant (GE Healthcare, Piscataway, NJ, USA)를 첨가하여 가볍게 현탁 시킨 후 10,000 rpm 이상으로 5 분간 원심 분리하여 상등액을 완전히 제거하였다. 100 μl copper 용액 (GE Healthcare, Piscataway, NJ, USA)과 400 μl 증류수를 첨가 후, 침전물이 완전히 용해될 때까지 혼합하였다. 각 튜브당 1 ml working color reagent (GE Healthcare, Piscataway, NJ, USA)를 첨가한 즉시 부드럽게 섞어 주었다. 상온에서 15~20 분간 놓아 둔 후, spectrophotometer (Mecasys, Daejeon, South Korea)를 이용하여 480 nm 파장에서 단백질 농도를 측정하였다.
2‐DE 분석은 Kim 등 (2012)에 쓰여진 방법으로 수행 하였다. 각각의 단백질 시료 (500 μg)는 24 cm IPG strip (GE Healthcare, Piscataway, NJ, USA) 이용하여 등전점 전기영동을 실시하였다. Rehydration 용액에 6 시간 동안 Rehydration 시킨 strip을 이용하여 전기영동을 실시했다. 전기 영동은 50 V 4 시간, 100 V 1 시간, 500 V 1 시간, 1000 V 1 시간, 2000 V 1 시간, 4000 V 2 시간, 8000 V 5시간, 8000 V 9 시간, 50 V 6 시간으로 수행 하였으며 IPGphore II platform (GE healthcare, Waukesha, WI, USA)에 의해 실시되었다. 전기 영동 후 strip은 equilibration 용액 [30% (v/v) glycerol, 2% (v/v) sodium dodecyl sulfate (SDS), 50 mM Tris‐HCl (pH 6.8), 100 mM DTT, 6 M Urea, 0.1 mg/ml bromphenol blue]에 equilibration 과정을 거친 뒤 55 mM iodoacetamide (Amresco, Solon,OH, USA)를 첨가 후 암실에서 30 분간 한번 더 equilibration를 수행하였다. Strip은 13% SDS‐PAGE gel 위에 수평으로 올려 놓고 전기영동을 수행하였다. 전기영동은 gel 당 2 W로, 500 V, 300 mA 에서 30 분을 걸어 준 뒤, gel 당 16 W에서 700 V, 300 mA로 loading dye가 gel 아래 0.5 cm로 내려갈 때까지 걸어 주었다. Gel은 colloidal Coomassie Briliant Blue (CBB) 염색 용액 [34% (v/v) methanol, 17% (w/v) ammonium sulfate, 3% (v/v) phosphoric acid, 0.1% (w/v) CBB G‐250]으로 염색하였다.
2‐DE gel은 transmissive scanner (PowerLook 1120, UMAX)를 사용하여 스캔 후 Image Master 2D Platinum software 6.0 (GE Healthcare, Waukesha, WI, USA)를 이용하여 이미지를 분석하였다. 분석 과정에는 spot 판별 과정과 부피 측정이 포함되어 있다. 첫 번째로 품종마다 양적변화를 보이는 spot을 판별 한 후 각 spot의 pixel 값을 측정하였다. 단백질 spot의 발현도는 p‐value가 0.05 이하인 spot들로 선정하였다.
Kim 등(2004)에서 보고된 바와 같이 2‐DE gel에서 Ingel tryptic digestion을 수행한 뒤 MALDI‐TOF/TOF mass spectrometer 분석을 수행하였다. Gel에서 colloidal CBB로 염색된 protein spot을 잘라내어 50% (v/v) ACN 이 포함 된 0.1 M NH4HCO3에 세척하여 SpeedVac Concentrator (Thermoscientific, Bremen, Germany) 으로 건조 시켰다. 건조 된 gel 조각에 10 mM DTT이 포함된 0.1 M NH4HCO3을 처리한 후 55℃에서 45분간 환원시킨 후 원심 분리로 상등액을 제거하고 55 mM iodoacetamide 이 포함 된 0.1 M NH4HCO3을 처리하여 암실에서 30분간 알킬화 반응을 시켰다. Gel 조각을 50% ACN이 포함된 0.1M NH4HCO3로 세척 작업을 수행한 뒤 digesting solution [25 mM NH4HCO3, 0.1% noctylglucoside (w/v), and 12.5 ng/μㅣ trypsin]을 처리하여 37℃ 에서 over night 하여 digestion 시킨 후 건조 시켰다. 단백질 동정은 ABI 4800 Plus TOF‐TOF Mass spectrometer (Applied Biosystems, Framingham, MA, USA)를 이용하여 355nm에서 200 Hz ND;YAG laser를 가동시켰다. 모든 데이터는 4700 Proteomics analyzer를 위한 Mass Standard Kit (Applied Biosystems, Framingham, MA, USA)를 이용하여 분석하였다. MS/MS spectra는 Database 검색은 database 검색 프로그램인 MASCOT에서 NCBI database 를 바탕으로 검색하였으며 검색 척도에 대해서 experimental peptide mass value에 대한 오차를 ±50 ppm으로 정하고 methionine oxidation과 carboxyamidomethylation을 variable modification으로 지정하였으며 miscleavage는 1로 지정하여 검색하였다.
수수 품종 별로 발현 정도에 차이가 나는 단백질을 비교하기 위하여 농촌진흥청 국립식량과학원 기능성 작물부로부터 분양 받은 4 품종의 수수 종자로부터 pheonol 추출법을 이용하여 단백질을 추출하였다 (Fig 1). 추출한 단백질은 이차원 전기영동을 실시한 후 colloidal CBB 염색법을 통해 품종별로 발현의 차이를 보이는 단백질 spot들을 관찰할 수 있었 다 (Fig 2). 총 652개의 spot들이 관찰 되었고, 이들 중 일부는 품종별로 발현량에 차이를 나타내었다. 이들 이차원 전기영동 gel을 ImageMaster software를 이용하여 품종별로 비교한 결과 8 개의 단백질이 품종별로 발현에 차이를 보였다 (Fig 3). 대부분은 흰찰수수 (WCS)에서 발현되는 경향이 컸으며 이들 단백질의 발현 정도의 차이를 ImageMaster software를 이용하여 비교한 결과, spot 3, spot 5와 spot 6은 흰찰수수(WCS)에서 유일하게 발현 되는 단백질임을 확인하였다. 또한 spot 1은 흰찰수수 (WCS)를 제외한 나머지 세품종에서 발현되었고 spot 4 와 spot 8은 대풍수수 (DPS)를 제외한 나머지 세 품종에서만 발현됨을 확인하였다. spot 2 는 토종수수 (TJS)에서 유일하게 발현되었다. Spot 7은 모든단백질에서 발현이 되는 것을 확인하였으나 그 중 토종수수(TJS)와 황금찰수수 (HGCS)에서 높게 발현 되는 것을 나타내었다. 또한 Anova 검정을 통하여 모든 spot은 품종별로 발현에 대한 차이에 있어 유의성이 있음을 확인하였다. 이와같이 품종별로 특이적으로 발현하는 단백질 spot들을 gel에서 잘라내어 tryptic digestion 과정을 거쳐 MALDI TOF/TOF MS로 분석을 실시하고 NCBI database를 기반으로한 MASCOT 검색 engine을 이용하여 단백질을 동정하였다 (Table 1). 동 정한 결과, RNA metabolism (spot 1, spot 4), HSP (spot 2), 저장 단백질 (spot 3, spot 5, spot 6), 산화‐환원 관련 단백질 (spot 8)들을 동정하였다.
[Table 1.] Proteins identified by MALDI-TOF/TOF MS.
Proteins identified by MALDI-TOF/TOF MS.
DEAD‐box helicase (spot 1)는 대풍수수 (DPS)에서 다른 세 품종에 비해 발현 정도가 가장 컸다 (Fig 3). RNA helicase는 이중 가닥 RNA를 풀어주기 위해 nucleotide triphosphate의 가수분해로 전달 받은 에너지를 사용하는 효소와 관련이 있는 것으로 보고된 단백질로 모든 단계의 RNA Metabolism에 관련되어 있다 (de la Cruz
Alpha‐crystallin‐Hsps_p23‐like protein (Spot 2)과 hypothetical protein (Spot 7)은 토종수수 (TJS)에서 발현 정도가 높은 단백질이다(Fig. 3). Alpha‐crystallin‐Hsps_p23‐like protein은 heat shock proteins family로서 alphacrystallin protein (ACD) 구조는 매우 다양한 기능을 가지고 있는 것으로 알려져 있으며 water stress에 반응하는 것으로 보고되어있다 (Maqbool
흰찰수수 (WCS)에서 Spot 3, Spot 4, Spot 5, spot 6 그리고 Spot 8이 다른 세 품종에 비해 특이적으로 높은 발현 정도를 보이고 있음을 관찰 하였다 (Fig. 3). Cupin family protein (Spot 3)과 Ribonuclease T2 (Spot 4), Globulin‐ 1 S allele precursor (Spot 5), Aldo‐keto reductases (Spot 8)은 흰찰수수 (WCS)에서 주로 보이는 단백질이다. 특히 Cupin family protein (Spot 3)은 병에 대한 저항성에 관련이 있으며 저장단백질 기능 및 종자의 발아 및 초기 생육에 관여한다 (Dunwell et al., 2001). 또한 알레르기를 유발 할 수 있다고 보고되었다 (Breiteneder and Mills, 2005). 흰찰수수 (WCS)에서만 특이적으로 보이는 단백질이므로 품종 marker로서의 활용 가능성도 기대할 수 있을 것으로 사료된다.
Ribonuclease T2 (Spot 4)는 인산이 결핍될 때 이 유전자가 발현하기 때문에 nutritional stress 조건 하에 식물을 구하기 위해 phosphate scavenging system RNA를 분해서 인산을 공급하는 체계의 한 부분일 것이라고 추측된다 (Taylor