Several yeast species have potential applications in biotechnology and the identification of such yeast species is of great interest. The first step in the identification of yeasts is the establishment of an effective isolation method. Thus, we compared the efficacy of different yeast media in the isolation of yeast associated with
In this study, we spread homogenized
All the isolates obtained using large agar plate containing chloramphenicol, streptomycin, Triton X-100 and L-sorbose were identified as yeast. Therefore, we concluded that this method is useful for selective screening of yeast species.
알로에는 식물분류학상 백합과에 속하는 상록의 다년초로서 다육질의 열대 약용식물이다(Reynolds, 2004). CO2의 수용과 고정단계를 시간에 따라 분리하여 광합성을 하는 대표적인 CAM (Crassulacean Acid Metabolism) 식물의 하나이며, 전 세계적으로 약 400 여종이 알려져있지만 건강식품 및 화장품용으로 사용되는 알로에 종은 5~6종에 불과하다(Grindlay and Reynolds, 1986; Kluge
알로에는 다당류, 지베렐린 등과 같은 많은 성분을 포함하지만 종에 따라 95% ~ 99%는 물이므로 유효 고형분의 함량은 매우 낮다(Grindlay and Reynolds, 1986; Reynolds and Dweck, 1999). 알로에 겔의 가장 풍부한 성분은 가용성 단당 및 복합 다당의 탄수화물이고, 알로에 외피 바로 밑에 위치한 유관속의 유세포에는 노란색 액즙의 삼출물 성분들이 존재하며, 약 300여종의 알로에로부터 80여종의 화합물이 동정되었다(Rodriguez
효모는 토양, 수생환경, 식물체, 곤충, 극한 환경 등의 다양한 환경에서 서식하고 있으며(Botha, 2011; Raspor and Zupan, 2006), 음료, 약품, 효소, 농업 및 산업에 중요한 영향을 미치고 있으며, 인류의 식생활에 많은 이익을 주고 있다(Deak, 2009; Tamang and Fleet, 2009). 이와 같은 효모의 연구를 수행하기 위해서는 세균과 곰팡이의 배양을 억제하고 효모만을 분리하는 기술이 요구된다. 하지만 효모를 선택적으로 분리해내기 위해서는 어떻게 세균과 곰팡이의 생장을 억제하고 효모만을 배양 및 분리할 수 있을 것인가가 관건일 것이다. 본 연구에서는 세균 및 곰팡이를 억제하는 방법으로 항생제 등의 화합물질을 이용해서 세균 및 곰팡이를 억제하고 알로에에 정착하고 있는 효모군집 분석을 수행하였다. 알로에로부터 효모의 분석은 거의 연구되지 않았으므로 그 결과가 유용할 것이며, 효모를 활용하는 산업 분야에서 중요한 기초 데이터로서 이용될 것으로 사료된다.
본 연구에 사용된 알로에는 알로에 베라(
먼저 DG18, DOB with CSM, GPY, SCG agar 배지를 멸균하여, 245 x 245 x 25 mm 크기의 사각 플레이트에(Nunc Bio‐Assay Dish, Thermo Scientific, Roskilde, Denmark)분주하여 고체 배지를 준비한다. 채취한 샘플을 파쇄하기에 알맞은 크기로 잘게 절제한 다음 멸균된 용기에 넣고 최종 볼륨이 10 mL이 되도록 10 mM potassium phosphate buffer를 첨가한 후 멸균가능한 homogenizer (T10 basis, IKA, Germany)를 이용하여 파쇄한다. 이와 같이 잘 파쇄된 알로에 원액 1 mL를 배지에 멸균된 유리 도말봉을 이용하여 도말한 후 25℃ 배양기에 2 ~ 5 일간 배양한다.
위와 같이 배양된 각각의 플레이트에서 효모만 선별하여 단일 균주를 얻기 위해 3차 분리까지 실시하였다. 배양된 적정 콜로니수의 플레이트로부터 전체의 효모를 분리하였다. 분리된 단일 균주들은 InstaGene Matrix (Bio‐Rad, Hercules, CA, USA)로 Genomic DNA를 추출하였으며, PCR은 EF
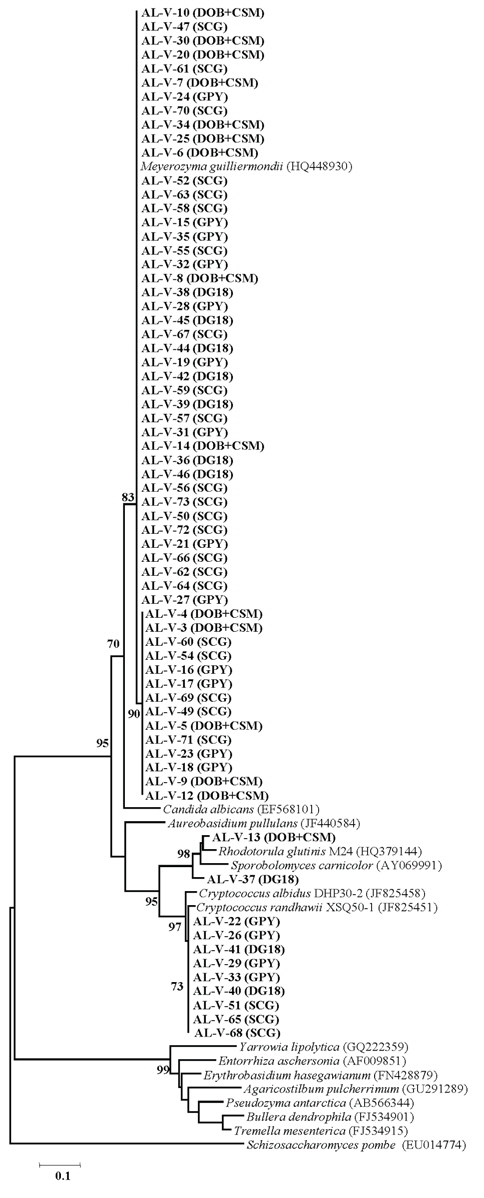
본 연구에서는 두 종류의 알로에에 서식하는 효모 균주를 분리하고 분류학적인 위치를 밝히고자 하였고 이를 위해 ITS 유전자의 염기서열 분석을 통한 계통학적 분석을 종합하여
Fig. 1에서 보이는 바와 같이
또한 본 연구에서는 세균과 곰팡이를 배제하고 효모만을 분리하는 스크리닝 법을 개발하고자 하였다. 이를 시험하기 위해서