한국 잔디 신품종 개발 목적으로 전라남도 장성군 삼서면 일대 한국잔디 중지류 생산포장에서 선발한 생육 및 피복속도가 빠르고 밀도 또는 가을철 녹색기간이 긴 2개 우량계통을 대상으로 DNA 수준에서 기존 중지류 또는 한국잔디들과 차이를 살펴보고 신품종 특이적으로 차이를 보이는 DNA 염기서열을 기반으로 RAPD-SCAR 마커를 개발하고자 본 연구를 실시하였다. RAPD 프라이머를 스크리닝 한 결과 N8021와 N8001 프라이머가 CY6069와 CY6097 품종 각각에 특이적인 DNA 절편을 약 600 bp와 700 bp 부근에서 발견할 수 있었다. 특이 밴드는 TA-cloning vector에 삽입 후 대장균에 형질전환하여 배양하였고, 이로부터 추출된 플라스미드 DNA의 염기서열 분석을 실시하여 최종적으로 중지류 신품종 특이 SCAR 마커 프라이머를 작성하였다. CY6069 선발 품종 특이 SCAR 마커(CY6069_550)는 다른 한국잔디 들잔디(야지), 한국잔디 중지, 갯잔디, 금잔디에서는 보이지 나타나지 않았던 식별 가능한 밴드를 보였고(550 bp), CY6097 선발 품종 식별을 위한 SCAR 마커(CNU70-6_1500)도 CY6097 계통에서만 특이적으로 나타나는 DNA 밴드를 약 700 bp 부근에서 증폭할 수 있었다. 형태 및 생육특성 차이와 함께 본 연구를 통해 개발된 RAPD-SCAR 마커를 활용하면 선발된 중지류 한국잔디 CY6069와 CY6097 계통을 실험실내에서 PCR을 통해 유전자원 식별 및 원산지 증명이 가능해졌고 영양번식을 통해 주로 번식하는 난지형 잔디 생산 포장에서 품종의 혼입으로부터 정확하게 분리해 낼 수 있을 것으로 판단된다.
Two medium-leaf ecotypes (CY6069, CY6097) belonging to one species (
한국 잔디(Korean lawngrass;
우리나라에서 생산되는 중지류 잔디는 안양중지, 삼덕중지, 평동중지, 장성중지 등 생산지역의 이름을 따라 여러 가지로 불리우고 있는데 생육속도가 빨라 피해를 입었을 때 지표면 피복과 회복 속도가 월등하고 수확 횟수가 일반 들잔디에 비하여 빠르기 때문에 농민들과 잔디 수요자들이 주로 선호하는 초종이다(Choi and Yang, 2005; Choi et al., 2012). 중지류 한국잔디는 엽폭이 3-4 mm 범위에 속하는 그룹으로 기존 한국잔디의 피해복구로부터 재생능력이 떨어지는 단점을 보완할 수 있는 가능성을 보였는데 최근 이들 중지류 한국잔디 생산 포장에서 기존의 중지보다 형질의 변이를 보이는 2 계통(CY6069, CY6097)들이 선발되었다(Choi et al., 2012). 이들 선발 계통들은 안양중지에 비교하여 8-10% 이상 피복율이 빠르고, 금잔디(
한국잔디류는 대부분 지하경 또는 포복경을 이용하여 영양번식을 하기 때문에 동일한 생산지역내에서도 다른 품종의 혼입으로 장기간 동일장소에서 재배할 경우 최초의 품종과 다른 형태가 섞여있을 가능성이 높다. 이러한 경우 기존의 관리방법에 따라 시비, 병해충 방제 및 물관리를 할 경우 관리 결과가 달라질 수 있어 잔디 관리에 혼란을 초래할 수 있어 생산 및 유통되는 품종에 대한 정확한 식별이 필요하다고 하겠다. 하지만 엽폭과 형태와 생육습성만을 가지고 이들 중지류 계통의 구분은 현실적으로 쉽지 않고 향후 계속되는 중지류 신품종의 출현을 대비하여 계통별 구분이 가능한 분자수준에서의 마커개발이 요구된다고 하겠다(Yerramsetty et al., 2008)
DNA 지문법(finferprinting)이나 이제까지 Radom amplified polymorphic DNA (RAPD)를 활용한 잔디 유전자원의 식별은 유전체 전체를 조사한다는 점에서 유리할 수 있으나 서로의 유사도를 설명할 수 있어 유전다양성 연구에 많이 활용되어 왔지만(Anderson et al., 2001; Caetano-Anolles et al., 1995), 최근 특정 품종 또는 유전적 계통에만 특이적으로 보이는 DNA 절편만을 이용하여 종(species) 특이 또는 농작물의 원산지 증명 등에 많이 활용되는 새로운 PCR(polymerase chain reaction) 기반 마커가 개발되고 있다(Busconi et al., 2003; Parodi et al., 2002). 그 중 보편화된 종 또는 품종식별을 위하여 RAPD 기반 sequence characterized amplified region (RAPD-SCAR) 마커의 개발을 들 수 있는데 한지형 잔디에 비하여 난지형 잔디의 경우는 상대적으로 미흡하였고(Abraham et al., 2005; Hyun et al., 2010; Scheef et al., 2003), 특히 한국잔디에 이용한 보고는 극히 제한적이라고 할 수 있다(Chung et al., 2013; Kang et al., 1999).
본 연구의 목적은 앞서 보고된 중지류 한국잔디 선발신품종을 식별할 수 있는 PCR기반 DNA 마커의 개발과 그 검증에 있다. 이를 위하여 선발된 중지류2개 출원 품종(CY6069, CY6097)을 대상으로 하여 제한효소를 이용하여 제놈(genome)을 무작위 절단하여 얻어진 특이 DNA절편을 PCR 조건에 적합한 염기서열 기반 프라이머로 제작하고자 하였다. 본 연구결과를 통하여 얻어진 PCR 기반 RAPD-SCAR 마커는 클론 또는 영양번식을 통해 주로 번식하는 한국잔디에서 신품종 개발자의 권리를 보호하고 잔디 유통시장에서 고유의 특성을 보이는 특정 품종을 손쉽게 식별할 수 있게 되어 원산지 증명 및 품종의 진위를 판단할 수 있는 근거로 활용될 수 있을 것으로 기대된다.
선발된 한국잔디 중지류 식별을 위한 SCAR 마커개발을 위하여 단국대학교 녹지조경학과에서 보유하고 있는 CY6069와 CY6097 2개 계통을 활용하였다(Choi et al., 2012). 이들 타겟 선발종과 함께 본 실험에 이용된 대조 한국잔디 종에는 충남대학교에서 보유하고 있는 한국 들잔디(
SCAR marker 개발을 위해 사용한 자연 선발 생태종(CY6069와 CY6097)과 4개의 대조 한국잔디 유전자원 잎으로부터 CTAB 방법을 이용하여 DNA를 추출하였다(Kang et al., 2008). 실험에 이용된 한국잔디 변이계통의 특이적 SCAR 마커 개발을 위하기 위하여 40여 개의 RAPD 프라이머를 구입하여(바이오니아, 대전) 조사하였고, 그 중 다른 한국잔디와 달리 2계통에서 특이적 다형성을 보이는 2개의 10-mer 오페론 프라이머(N8021과 N8001)가 각각의 선발 신품종을 위하여 최종 선정되었다(Table 1). 선정된 프라이머와 DNA 증폭기(Takara TP600, Takara, Japan)를 이용하여 RAPD-PCR을 수행하였다.

RAPD primers to detect DNA fragments distinctly amplified in the medium-leaf zoysiagrass ecotypes used in this study.
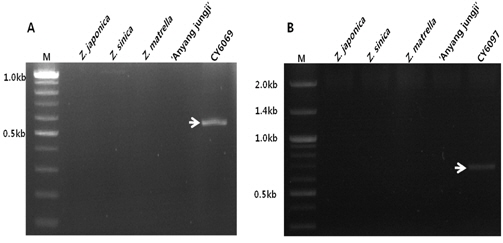
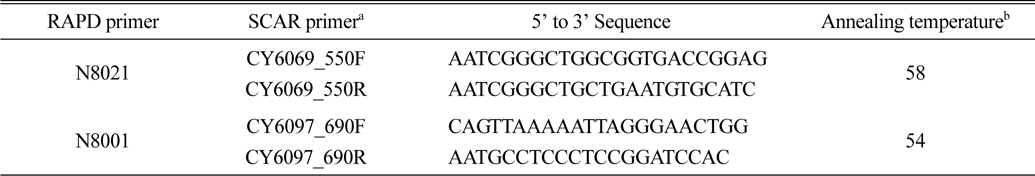
PCR 조건은 94℃에서 5분간 일차적으로 변성을 유도한 후 94℃에서 30초간 DNA 이중가닥을 분리하였고, 36℃에서 1분간 DNA 복원(annealing), 72℃에서 2분간 연장과정을 총 35회 반복하여 수행하였다. 최종적으로 72℃에서 7분간 PCR 산물의 안정화를 수행하였다. PCR 완료 후 1.5% 아가로즈 젤에서 100 v로 2시간 동안 전기영동을 수행하여 선발 신품종에서 보이는 특이 DNA 절편을 확인하였다(Fig. 1).
RAPD 분석을 통해 확인된 특이 DNA 절편은 밴드 위치의 DNA 절편을 잘라 TA-cloning vector에 삽입하였다(Life Technology, Carlsbad, CA). Vector에 삽입된 특이 밴드의 염기서열 분석을 위해 42℃에서 열 충격 (heat-shock) 방법을 이용하여 대장균 (
특이밴드가 삽입된 것으로 확인된 대장균 균락들 중 3개를 선별한 후 항생제(ampicillin)가 포함된 각각의 액체 LB 배지에 넣고 37℃ 진탕 배양시킨 후 15시간 동안 배양 후 대장균에 포함된 DNA를 추출하였다. 이렇게 추출된 플라스미드 DNA는 염기서열 분석 장치 ABI 3130XL(Applied Biosystems, Foster City, CA, USA)를 이용하여 염기서열 분석을 실시하였다. 염기서열 분석에 필요한 프라이머는 TA-cloning vector에 포함된 M13 primer 쌍을 사용하였다. 이 후 분석된 특이밴드의 염기서열을 바탕으로 Forward(F)와 Reverse(R)를 한 쌍으로 하는 선발 신품종 2계통 특이 SCAR 마커 프라이머를 디자인하였다(Table 2).
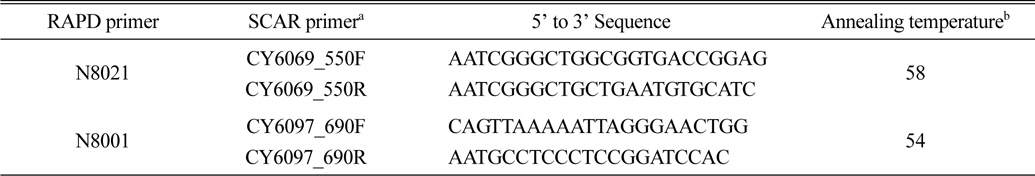
SCAR marker primer pairs specifically designed from the amplified and cloned RAPD fragments for detecting the medium-leaf zoysiagrass ecotypes used in this study.
제작된 SCAR 프라이머 쌍을 이용한 특이적 밴드의 패턴 여부는 PCR 방법을 이용하여 검정하였다(Fig. 4). N8005와 N8032 프라이머를 이용한 RAPD 결과로부터 이 염기서열을 바탕으로 제작한 SCAR 마커 프라이머 쌍으로 PCR을 수행하였다(Table 2). CY6069 신품종 식별을 위한 RAPD-SCAR 마커의 PCR 조건은 94℃에서 5분간 일차 변성을 실시한 후 94℃에서 30초간 DNA 이중가닥의 분리, 58℃에서 30초간 DNA 복원, 72℃에서 40초간 연장과정을 총 25회 반복하여 수행하였고 최종적으로 72℃에서 7분간 PCR 산물의 안정화를 유도하였다. 반면에 CY6097 신품종 식별을 위하여 제작된 프라이머의 증폭을 위해서 위와 동일한 방법으로 실시하였으나 DNA 재결합(annealing)은 54℃에서 30초간 실시하였다. PCR을 완료한 후 1% 아가로즈 젤을 이용한 전기영동을 통해 특이 DNA 절편의 증폭 결과를 최종 확인하였다.
장성군 삼서면 일대 중지류 재배 생산포장으로부터 수집한 중지형 한국잔디에서 우수형질을 갖는 CY6069 계통과 CY6097 계통 각각의 선별체와 positive control로 이용된 4개 한국잔디 품종 또는 종(들잔디, 안양중지, 갯잔디, 금잔디)등 총 5개 유전자원을 대상으로 하여 구입한 RAPD primers를 사용하여 CY6069와 CY6097 선발체 특이밴드를 찾고 그 염기서열을 기반으로 하여 SCAR marker를 개발하고자 하였다.
RAPD 마커는 유전체 염기서열 정보가 없는 많은 식물의 유전다양성 및 타겟 유전자의 증폭에 매우 유용한 마커 시스템이지만, 우성 마커군에 포함되므로 원하는 DNA존재의 유(1) 또는 무(0) 형태로 구분하고, 특정 유전자좌(gene locus)에 있는 대립유전자가 동형접합체(homologous)인지 이형접합체(heterologous) 형태인지를 알기가 어렵다(Kumar and Gurusubramanian, 2011). 또한 PCR 반응 조건(DNA 양, PCR 반응 혼합물질, 증폭 횟수 등)에 따라 효소반응이 영향을 받으므로 결과의 재현성이 떨어지고 준비과정이 복잡하다. 이러한 문제점을 해결하기 위하여 PCR 기반 염기서열에 기초한 SCAR 마커의 개발이 필요하게 되었다.
N8021 RAPD primer를 이용한 PCR 반응 결과 CY6069 선발 신품종은 약 600 bp 부근에서 특이 밴드를 관찰하였고, N8001 RAPD primer를 이용한 PCR 반응 결과 CY6097 신품종 특이 DNA 절편을 약 700 bp 부근에서 관찰할 수 있었다(Fig. 1). CY6069와 CY6097 선발 신품종 특이 다형성을 보인 DNA 절편은 클로닝 벡터에 삽입 후 대장균에서 증식한 후 삽입된 부위의 절편을 염기서열 분석한 결과 CY6069 선발종 특이 다형성 DNA 절편은 556 bp 크기로 증폭되었고(Fig. 2), CY6069 선발 신품종 특이 DNA 절편은 690 bp로 증폭되었다(Fig. 3).
선발된 2계통의 밝혀진 염기서열을 기초로 다형성을 보인 DNA 절편 전체를 증폭하기 위하여 SCAR 마커 프라이머 쌍을 디자인하여 검증하고자 하였다(Table 2). 증폭조건을 달리하여 선발계통과 다른 국내에서 생육하고 있는 4종의 한국잔디(들잔디, 안양중지, 갯잔디 및 금잔디)를 대상으로 PCR 증폭을 실시한 결과 기대했던 크기의 DNA 절편을 얻을 수가 있었다(Fig. 4). 증폭된 DNA 절편은 기대했던 것처럼 비교구의 다른 한국잔디에서는 나타나지 않고 선발 계통에서만 예상했던 크기로 증폭되는 것을 확인할 수 있었다(Fig. 4). RAPD에서 보인 특이밴드의 크기와 일치하는 약 550 bp 부근에서 CY6069에서만 보이는 특이적인 밴드가 나타났으며, 약 700 bp 근처에서 CY6097에 특이적인 밴드가 나타났다. 또한 제작된 SCAR 마커 프라이머를 위한 PCR 조건을 적용할 때 다른 비특정 밴드의 출현을 볼 수 없었다.
동일한 프라이머를 이용해 DNA를 증폭해도 PCR 결과에 있어 차이(bias)가 발생할 수 있기 때문에 개발된 SCAR 마커의 이용 편이를 위해서는 정확한 증폭조건을 확립하는 것이 중요하다. 이러한 PCR 차이는 생물종별 DNA 또는 PCR 자체의 원인(변성된 DNA 이중가닥의 재결합 온도, PCR 증폭횟수, 주형 DNA 가닥에 결합하는 프라이머 성질, DNA 합성효소)에 의해 발생할 수 있다(Ishii and Fukui, 2001). 본 연구에서 개발된 2개의 SCAR 마커는 PCR 증폭조건이 대부분 동일하지만CY6069_550 SCAR 마커는 PCR 증폭 시 변성된 DNA 재결합(annealing)을 위해 58℃가 필요하지만 CY6097_690 SCAR 마커는 54℃가 적당하다는 것을 알 수 있었다(Table 2).
본 연구에서와 같이 단일 잔디 샘플을 분리하여 증폭하는 경우는 비 특이적 밴드를 비교적 쉽게 제거할 수 있지만 동일한 잔디 생산포장에서 여러 품종이 혼재되어 추출된 DNA 자체가 섞여 있는 경우나 또는 한가지 잔디 품종에 여러 프라이머를 동시에 스크리닝 할 경우는 특히 PCR 증폭조건이 중요하다고 할 수 있다. 제작된 프라이머가 주형 DNA와의 결합효율에 문제가 있는 경우 그 효율을 높이기 위해서는 DNA 재결합 온도를 낮추어 해결할 수 있지만 프라이머 특이성이 떨어져 비특이 밴드의 출현이 많을 수 있고 이 때 주형 DNA의 G+C 함량이 또한 영향을 줄 수 있다(Ishii and Fukui, 2001). CY6097 특이 SCAR 마커는 더 긴 DNA 절편을 증폭하였지만 낮은 DNA 재결합 온도에서 밴드의 출현을 보인 것은 주형 DNA에 결합하는 프라이머의 낮은 G+C 비율 때문인 것으로 보인다(Table 2).
본 연구를 통하여 확보된 RAPD-SCAR 마커 개발 기술은 향후 국내 잔디 신품종 수요의 급증에 따라 새로운 품종이 지속적으로 출시될 경우를 대비하여 유용하게 활용될 수 있을 것으로 기대된다. 일본에서와 같이 잔디 품질을 등급화하여 유통한다면 고급 잔디를 생산하는 생산자는 상대적으로 더 많은 수익을 보장받을 수 있고, 잔디수요자들은 생산된 잔디를 신뢰하고 이용할 수 있게 될 것이다. 이러한 잔디산업 시장의 변화는 결국 지속적인 고품질 잔디의 생산과 잔디산업 전체의 활성화를 도모할 수 있다고 하겠는데 문제는 품종의 다변화를 대비한 품종 고유의 식별이 형태만으로는 시각적으로 구분이 쉽지 않다는 점이다. 특히 영양번식을 주로 증식 수단으로 하는 한국잔디의 경우 품종 별 식별 마커의 개발은 잔디 품종의 등급화, 원산지 증명 및 잔디산업 활성화에 필수적인 이용수단이 될 것으로 보인다. 또한 지하경이나 포복경 등 영양체의 일부를 이용하여 대량 증식 및 활용을 할 경우 육종가의 권리가 침해되기 쉬우므로 본 연구에서 개발된 PCR 기반 분자마커의 지속적인 개발과 검증이 필요할 것으로 사료된다.
![RAPD amplification profiles of five zoysia species or medium-leaf selections [CY6069 (A) and CY6097(B)] using RAPD primer N8021 (left) or N8001 (right). Zoysiagrass species compared were Zoysia japonica ‘Anyang Joonggi’ (Lane 1), Zoysia japonica (wild type; lane 2), Zoysia matrella (lane 3), Zoysia sinica (lane 4), CY6069 (lane 5 of gel A), and CY6097 (lane 5 of gel B). The arrows indicate specific DNA fragments which were cloned and sequenced. M; 100bp plus DNA marker.](http://oak.go.kr/repository/journal/15062/JDHHCQ_2013_v2n2_191_f001.jpg)