Norovirus (NoV) is a major cause of food poisoning outbreaks in Korea. Most NoV outbreaks originate from environmental contamination, but bivalves such as oysters are also important vectors. Oyster
우리나라는 주요 패류생산국가 중의 하나로 연간 400천 톤 이상의 패류가 생산되며, 품종별로는 굴이 300천 톤 이상으로 전체 패류 생산량의 70% 정도를 차지하고 있다. 또한 우리나라에서 생산되는 굴은 90% 이상이 천해양식으로 생산되며, 주로 경상남도와 전라남도의 남해안에 집중되어 있다(MOF, 2013). 우리나라에서 굴이 생산되는 양식장은 대부분 연안에 위치하고 있으므로 각종 오염원들로부터 영향을 받을 가능성이 크다. 이전 보고에 따르면 대규모 하천이나 인구가 밀집되어 있는 지역의 해역에서 세균함량이 높고, 강우발생에 따라 육상에서 유래한 분변성 오염물질이 해역으로 유입되어 해역에 영향을 나타낸다고 보고하였다. 특히 인축의 분변에는 장관계 바이러스 등의 식중독 원인체가 함유될 가능성이 있으며, 분변이 오염된 해역에서 생산된 굴은 이러한 식중독의 원인물질을 함유할 가능성이 크다. 최근 노로바이러스에 의한 식중독이 가장 문제가 되고 있으며, 오염해역에서 생산된 굴이 노로바이러스의 매개체 역할을 할 가능성이 크다(Lee et al., 2010; Oh et al., 2012; Iritani et al., 2014).
노로바이러스는
노로바이러스는 비세균성 장염의 90% 이상의 원인으로 알려져 있으며, 연령이나 성별에 무관하게 가장 많이 장염을 일으키는 원인물질로 알려져 있다(Fankhauser et al., 2002). 미국의 경우 2009년부터 2012년까지 식품과 관련된 식중독의 48%가 노로바이러스에 의해 발병되는 것으로 알려져 있으며, 일본의 경우에는 2001년부터 2007년까지 발생한 식중독 중 노로바이러스로 기인한 환자수가 가장 많은 것으로 보고되고 있다(Jo et al., 2009; Hall et al., 2014). 국내에서도 최근 원인물질별 식중독 발생현황을 살펴보면 2005년까지는 노로바이러스가 병원성 대장균의 다음 순위를 차지하였으나, 2006년 이후 노로바이러스에 의한 식중독이 크게 급증하여 2002년부터 2013년까지 노로바이러스에 의한 식중독 발생건수가 전체 식중독 발생건수의 15% (437/2,858)로 원인물질들 중 가장 큰 비중을 차지하고 있다(MFDS, 2014).
노로바이러스 식중독의 원인물질로는 식수, 과일, 채소, 패류 등이 있으며 주로 분변 및 구강 경구로 감염된다. 과일이나 채소류는 오염된 물이나 오염된 식품을 조리하는 사람에 의해 바이러스에 오염되며, 패류는 서식환경 즉 해수가 오염되어 있을 경우 여과섭이(filter feeding) 과정 중에 체내에 바이러스를 농축하게 된다. 이들 패류 중 굴은 생굴의 형태로 소비되는 경향이 있고, 다양한 나라에서 소비가 이루어지기 때문에 이와 관련된 노로바이러스 발생 사례가 일본 등의 여러나라에서 보고된 바 있다(Shin et al., 2010; Alfano-Sobsey et al., 2012; Iritani et al., 2014). 우리나라에서는 식중독 발생의 역학조사가 부족하여 굴 섭취로 인한 노로바이러스 식중독 발생 사례는 아직 보고된 바없지만, 해역에서 생산·유통되는 굴에 대한 노로바이러스 검출사례는 보고된 바 있다(Moon et al., 2011; Shin et al., 2013). 하지만 이러한 보고들은 RT-PCR (reverse transcription-polymerase chain reaction)을 이용하여 바이러스 유전자를 검출한 결과로 노로바이러스의 정성분석만을 보고하였다. 노로바이러스는 숙주세포를 이용한 배양기술이 개발되지 않아 RT-PCR을 이용한 유전자 검사법이 일반적인 방법이지만, 시간 소모가 많고 정량분석이 불가능하다는 단점이 있어 최근에는 형광물질을 이용하여 유전자 증폭과정을 실시간으로 모니터 하는 real-time RT-PCR이 많이 사용되고 있다(Patel et al., 2009).
본 연구에서는 우리나라 굴의 주 생산지인 남해안에서 분변오염 정도가 높은 것으로 확인된 통영시 인평동 인근의 굴 양식장을 대상으로 2012년 1월부터 12월까지 1년간 노로바이러스에 대한 모니터링을 실시하였으며, real-time RT-PCR을 이용하여 바이러스 유전자를 정량적으로 분석하였다. 또한 노로바이러스에 감염된 굴의 가열처리 조건을 구명하기 위하여 가열처리 후 노로바이러스의 농도변화를 분석하였다.
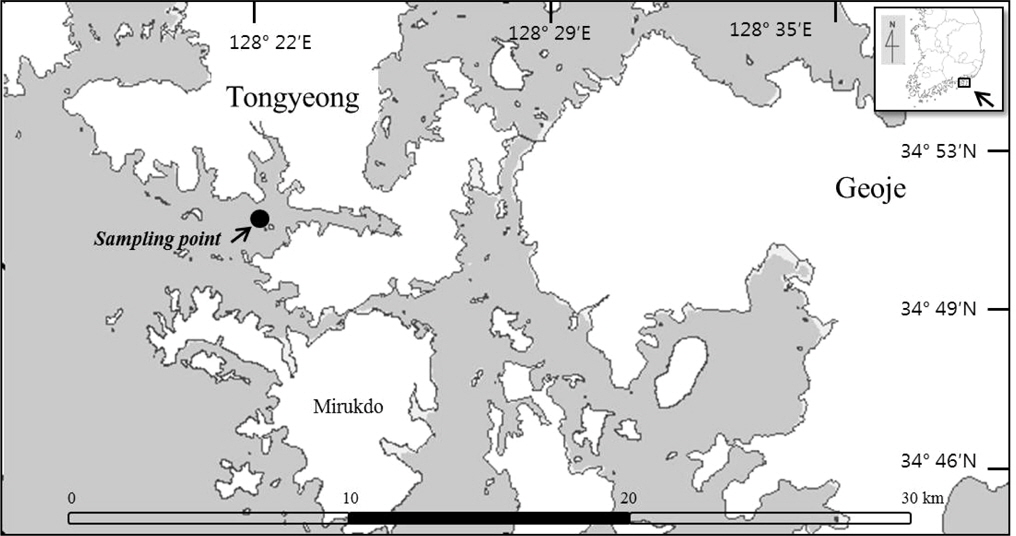
우리나라에서 생산되는 굴은 남해안에 집중되어 있으며, 도심의 하수가 해역으로 유입될 가능성이 큰 통영시 인근의 굴 양식장을 대상으로 샘플링을 실시하였다(Fig. 1). 2012년 1월에서 12월까지 경상남도 통영시 인평동 인근 굴 양식장 1지점에서 총 21개의 참굴(
가열처리에 따른 노로바이러스 농도변화를 분석하기 위한 굴의 바이러스 감염은 Naturally-contamination방법을 이용하여 인위적으로 감염시켰다(Oh et al., 2012). 즉, 경남 통영시 일원에서 채취한 참굴(
가열처리는 굴을 가열하여 섭취할 경우 굴에 미칠 수 있는 온도 및 바이러스의 외피단백질에 손상을 미칠 수 있는 온도 등을 고려하여 60, 68, 70 및 100℃의 4개의 구간으로 설정하였으며, 각각 15분간 항온수조에 오염된 굴을 반응시켰다(Alfano-Sobsey et al., 2012; Nuanualsuwan et al., 2002). 오염된 굴은 패각을 분리하여 알굴 상태로 가열처리를 실시하였으며, 처리후 즉시 노로바이러스 분석을 실시하였다.
굴에서 노로바이러스 분리는 Jothikumar et al. (2005)을 일부 변형하여 사용하였다. 즉 분리된 중장선에 3 g에 동량의 300 µg/mL Proteinase K solution (Promega, USA)을 첨가하였으며, 호모게나이저를 사용하여 완전히 균질화하였다. 균질화된 샘플은 37℃, 320 rpm의 조건으로 1시간 반응시킨 후 Proteinase K의 불활성화를 위하여 65℃에서 15분간 추가로 반응시켰다. 반응액은 3,000 g에서 5분간 원심분리 후 상등액을 RNA 추출에 사용하였다. 자연감염을 통해 노로바이러스가 검출된 샘플을 양성 대조군으로 사용하여 실험의 신뢰성을 확보하였다.
RNA 추출에는 Vrial RNA mini kit (QIAgen, USA)을 사용였다. 샘플 300 µL에 AVL buffer (Guanidine thiocyanate 함유) 1,120 µL를 첨가하여 혼합한 후 실온에서 10분간 반응시켰다. 반응액에 95-100% 에탄올 1,120 µL를 첨가하여 혼합하였으며, 혼합액을 630 µL를 spin column tube로 옮겨 6,000 g에서 1분간 원심분리하였다. 남은 혼합액을 동일한 방법으로 처리 한 후, 동일한 spin column tube에 AW1 buffer (Guanidine hydrochloride 함유) 500 µL를 첨가하여 6,000 g에서 1분간 원심분리하였다. 또한 AW2 buffer (Sodium azide 함유) 500 µL를 각각 첨가하여 20,000 g에서 3분간 원심분리하였으며, 원심분리 후 spin column에 걸러진 용액은 제거하였다. 다음으로 spin column을 새로운 tube로 옮긴 후 AVE buffer (sodium azide 및 poly A 함유)을 60 µL를 넣고 1분간 반응시켰다. 마지막으로 6,000 g로 1분간 원심분리하여 realtime RT-PCR을 수행하기 위한 template로 사용하였다.
노로바이러스 유전자의 정량분석을 위한 표준곡선은 pET30a vector에 해당 유전자의 sequence (GI형 96 bp, GII형 98 bp)를 삽입하여 제작된 plasmid DNA (Tarara, Korea)를 이용하였다. plasmid DNA는 GI형과 GII형 각각 106-102 copies/reaction 농도로 희석한 후 realtime RT-PCR을 실시하였으며, Thermal cycler dice realtime software (Takara, Japan)을 이용하여 표준곡선을 산출하였다.
Realtime RT-PCR 반응을 위하여 OneStep RT-PCR kit(QIAgen, USA) 및 RNase inhibitor (Ambion, USA) 시약을 사용하였다. 폴리오바이러스의 RNA를 Internal control RNA(IC, US FDA)로 첨가하여 반응이 적절히 이루어지는지 확인 하였으며 음성대조군으로 RNase-free water를 사용하여 실험의 신뢰성을 확보하였다.
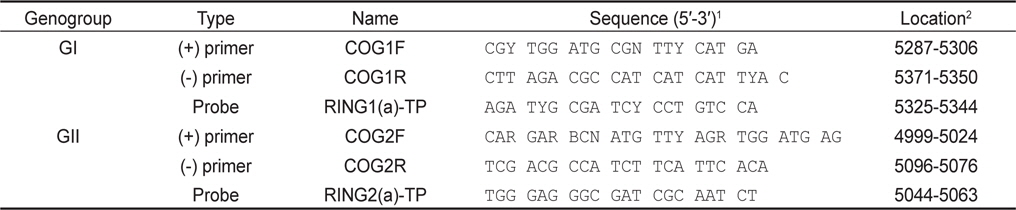
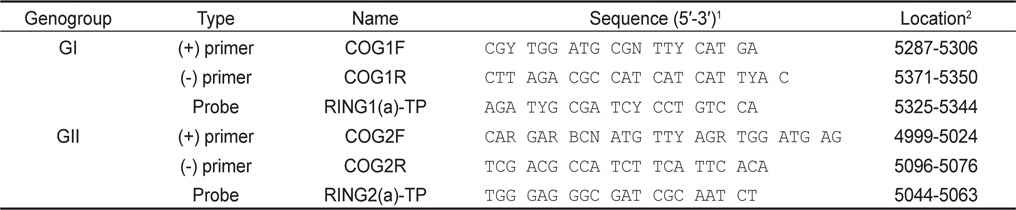
5 units/µL Enzyme Mix 1 µL, 5X RT buffer 5 µL, 10 mM dNTP 1 µL, 25 mM MgCl2 0.75 µL, 5 units/µL RNase inhibitor 0.25 µL, 10 µM primer 1 µL, 10 µM IC primer 0.5 µL, 10 µM probe 0.5 µL, IC RNA 1 µL 및 template RNA 5 µL로 반응액을 조성한 후, DW를 첨가하여 최종적으로 25 µL의 반응액을 조성하였다. 반응조건으로 50℃ 50분간 역전사 반응 후, 95℃에서 15분간 DNA를 변성하였다. 이 후 95℃ 10초, 53℃ 25초 및 62℃ 70초간 반응을 45 cycle 반응시켰으며, PCR 반응은 Thermal cycler dice TP800 (Takara, Japan)를 이용하였다. 본 연구에 사용된 primer 및 probe는 Kageyama et al. (2003) (Table 1)을 참고로 하였으며 probe는 노로바이러스 및 IC에 FAM-TAMRA detector와 ROX-BHQ2 detector 적용하였다. GI형 및 GII형 각각에 IC를 첨가하여 duplex realtime RT-PCR을 실시하였으며 샘플마다 3회의 PCR 반응을 실시하였다.
[Table 1.] Primers and probes used this study
Primers and probes used this study
2012년 1월에서 12월까지 경상남도 통영시 인평동 인근 굴 양식장 1지점에서 총 21개의 참굴을 샘플링 하여 노로바이러스를 분석하였다.
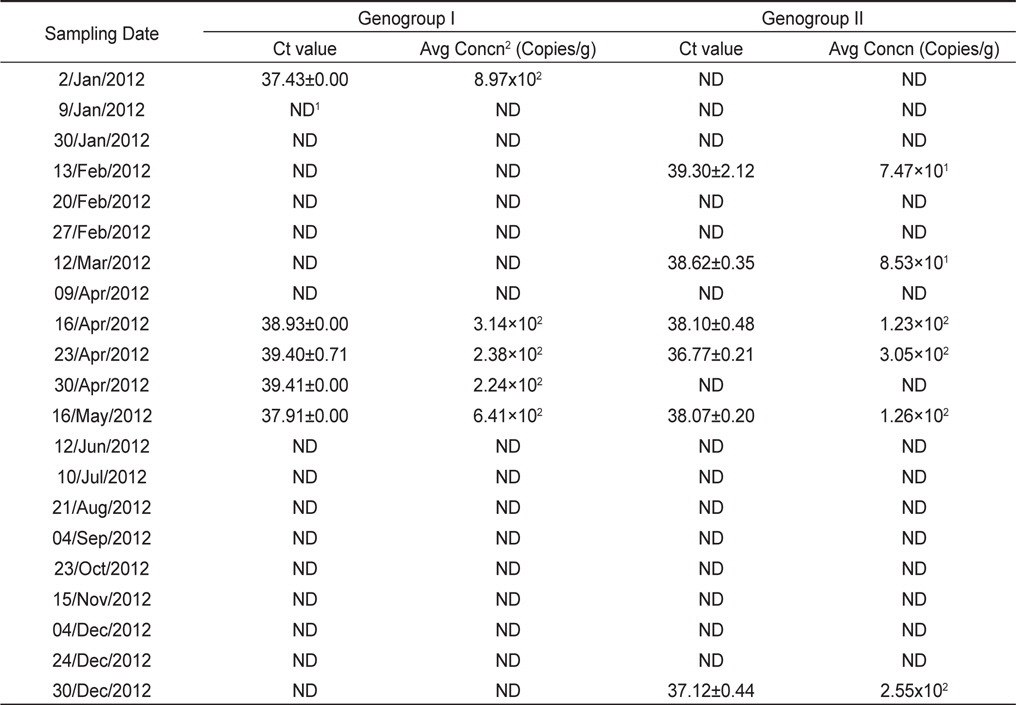
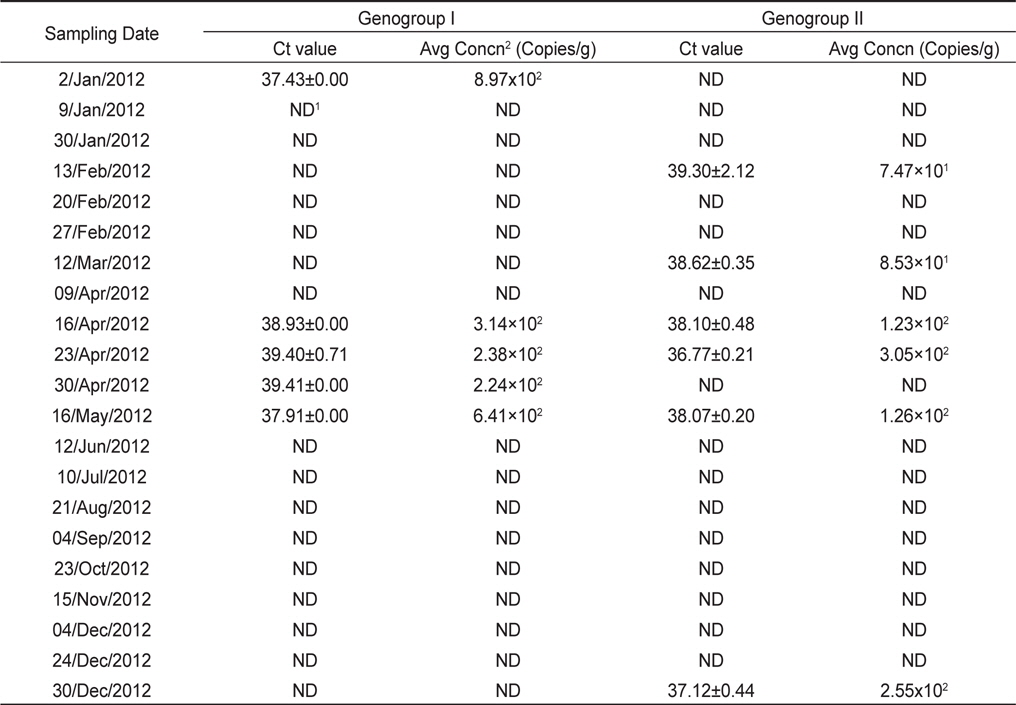
모니터링 결과, 2012년 1월에서 12월까지 총 21개의 샘플 중 8개의 샘플에서 노로바이러스가 검출되어 38% (8/21)의 검출율을 나타내었다. 유전형별로는 GI형이 24% (5/21), GII형이 29% (6/21)의 검출율을 나타내었으며, 14% (3/21)의 샘플에서 GI형과 GII형이 동시에 검출되었다. 시기별로는 1-5월, 12월에 집중적으로 검출되는 경향을 나타내었다(Table 2).
[Table 2.] Quantitative analysis of NoVs detected in Korean oysters Crassostrea gigas in 2012
Quantitative analysis of NoVs detected in Korean oysters Crassostrea gigas in 2012
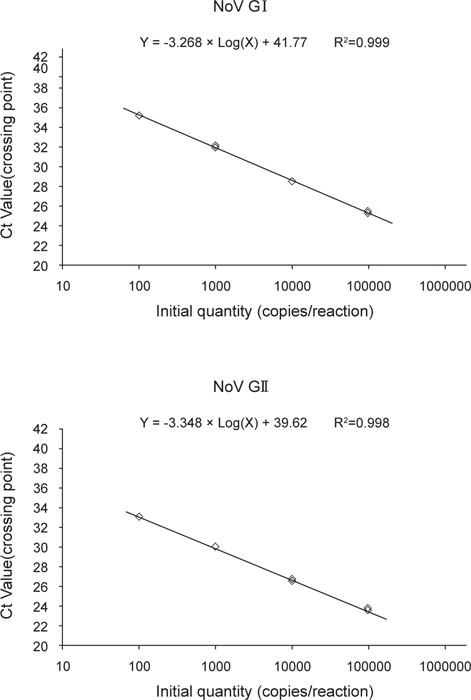
노로바이러스 정량분석을 위한 표준곡선은 GI 및 GII형 plasmid DNA를 각각 106-102 copies/reaction로 희석하여 분석하였다. 표준곡선의 선형분석 결과, GI형은 상관계수(R2) 0.999, 기울기가 −3.286으로 나타났으며, GII형의 경우 상관계수(R2) 0.998, 기울기는 −3.348로 나타났다(Fig. 2). 표준곡선에 realtime RT-PCR 결과의 Ct value를 대입하여 샘플의 노로바이러스 농도를 정량하였으며, 분석된 샘플의 최종 결과값은 샘플당 3회 실험결과의 평균으로 나타내었다.
노로바이러스가 검출된 샘플의 Ct value는 GI형이 37.43-39.41, GII형이 36.77-39.30로 나타났으며, 이를 표준곡선에 대입하여 계산된 노로바이러스의 농도는 GI형이 8.97×102−2.24×102, GII형이 3.05×102−7.47×101 copies/g으로 나타났다. 가장 높은 농도로 검출된 샘플은 GI형이 8.97×102 copies/g으로 검출되었으며 가장 낮은 농도로 검출된 샘플은 GII형이 7.47×101 copies/g으로 검출되었다(Table 2).
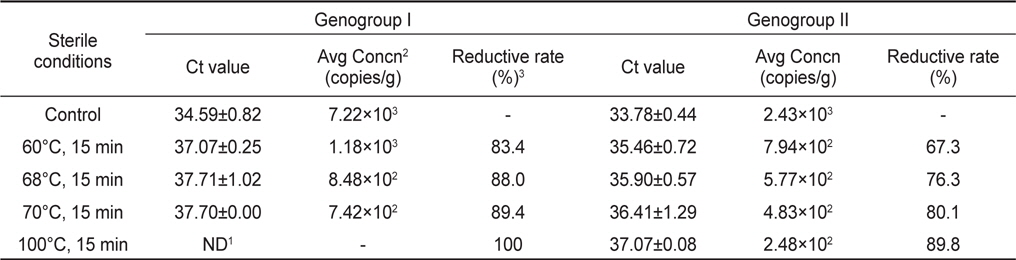
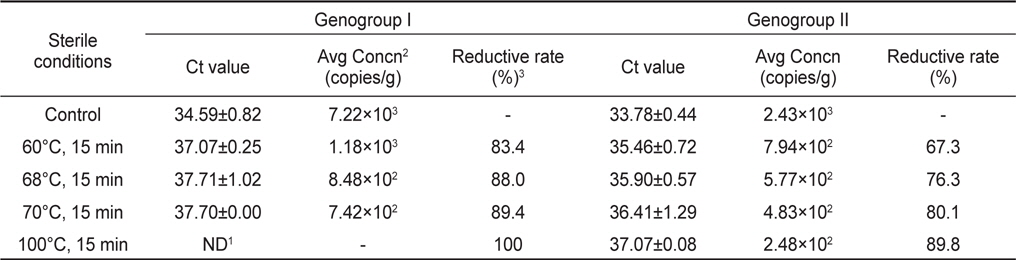
가열처리에 따른 노로바이러스 농도변화를 관찰하기 위하여 굴을 사각채롱에 넣어 하수의 유입이 많은 연안에 수하한 후 자연적으로 노로바이러스에 감염시켰다. 7일간 하수에 노출된 굴에서 노로바이러스 GI형과 GII형이 각각 이 7.22×103 및 2.43×103 copies/g 농도 감염된 것으로 확인되었으며, 이를 각 가열조건에 따라 처리하였다.
노로바이러스에 감염된 굴을 각 구간별로 가열처리하여 realtime RT-PCR로 분석한 결과 Ct value는 GI형이 37.07-37.71, GII형이 35.46-37.07로 나타났으며, 이를 표준곡선에 대입하여 계산된 노로바이러스의 농도는 GI형이 1.18×103−7.42×102, GII형이 7.94×102−2.48×101 copies/g으로 나타났다. 노로바이러스의 농도는 60, 68, 70℃에서 처리한 결과 GI형이 각각 83.4, 88.0, 89.4% 감소되었으며, GII형은 60, 68, 70, 100℃에서 각각 67.3, 76.3, 80.1, 89.8% 감소되는 것으로 나타났다. GI 및 GII형 모두 높은 온도에서 처리할 경우 더 높은 감소효과를 나타내는 것으로 확인되었지만, 100℃에서 처리한 GI형의 경우를 제외하고는 가열처리 후에도 일부 유전자가 검출되는 것으로 나타났다(Table 3).
[Table 3.] Quantitative analysis of heat-treated NoVs in contaminated oysters Crassostrea gigas
Quantitative analysis of heat-treated NoVs in contaminated oysters Crassostrea gigas
노로바이러스 식중독은 감염환자의 분변이 환경이나 식품등을 오염시키고, 오염된 환경이나 식품을 통하여 환자가 발생하는 순환고리를 형성하고 있다. 이러한 원인들 중 인축의 분변에 오염된 해역에서 생산된 패류는 노로바이러스 식중독의 매개체 역할을 할 가능성이 크며, 패류 중에서도 생굴섭취가 많은 굴에 의한 노로바이러스 식중독 사고가 일본을 포함한 여러나라에서 보고된 바 있다(Alfano-Sobsey et al., 2012; Iritani et al., 2014).
우리나라에서는 노로바이러스 식중독과 원인식품과의 상관관계에 대한 역학조사에 대한 보고가 부족하여 굴 섭취로 인한 노로바이러스 식중독 또한 보고된 바가 없다. 하지만 최근 오염된 해역에서 생산된 파래(Enteromorpha spp)에 의한 노로바이러스 식중독 사고가 보고된 바 있으며, 우리나라 해역에서 생산·유통되는 굴에서 노로바이러스가 검출된 사례가 있으므로 우리나라에서도 굴이 노로바이러스 식중독의 매개체 역할을 가능성이 크다(Moon et al., 2011; Shin et al., 2013; Park et al., 2014).
노로바이러스는 숙주세포에 의한 인공배양법이 개발되지 않아, 현재 널리 이용되는 검출방법은 RT-PCR을 이용한 유전자 분석법이다. RT-PCR법에 따른 노로바이러스 검출법은 2회 이상의 PCR 반응 및 agarose를 이용한 전기영동을 수행해야하는 번거로움이 따르고 정량분석이 불가능하다는 단점이 있다. Realtime RT-PCR은 이러한 단점을 보완하기에 적합한 분석법으로 기존의 RT-PCR에 형광물질의 probe를 적용하여 단시간에 결과를 모니터링할 수 있고, 정량분석이 가능한 장점이 있어 최근 realtime RT-PCR을 적용한 노로바이러스 분석법이 유럽의 국제표준(International Organization for standardization)으로 등재된 바 있다(ISO, 2013).
본 연구에서는 선행연구의 정성분석 결과를 보완하고자 realtime RT-PCR을 적용하였으며, 통영시 인평동 인근의 양식장 1지점에서 2012년 1월부터 12월까지 21회에 걸쳐 굴을 샘플링하여 노로바이러스를 분석하였다. 대상지점은 연중 분변 오염정도가 높은 것으로 확인되어(Shim et al., 2009; Oh et al., 2012), 인체의 분변에 기인하는 노로바이러스 또한 오염될 가능성이 클 것으로 판단되었다. 분석결과 38% (8/21)의 샘플에서 노로바이러스가 검출되었으며, 노로바이러스가 검출된 샘플의 농도는 8.97×102−7.47×101 copies/g으로 나타났다. 모든 샘플이 103 copies/g 이하의 농도에서 검출되었으며, 102 copies/g보다 낮은 농도로 검출된 샘플이 9.5% (2/21)로 확인되었다. 이와 유사한 연구로 영국의 자료에 따르면 2009년부터 3년간 영국에서 생산된 굴의 76% (643/844)가 노로바이러스에 오염된 것으로 나타났으며, 바이러스의 농도는 102 copies/g 이상이 36.5%이며 103 copies/g 이상이 24.9%로 보고한 바 있다(EFSA, 2012). 따라서 본 연구에서의 샘플링 지점은 분변오염 정도가 높은 곳임을 고려하면 검출된 노로바이러스의 농도가 높지 않은 것으로 판단할 수 있으나, 이러한 결과는 단기간의 국소적인 결과이므로 우리나라에서 굴이 생산되는 해역에 대한 집중적인 조사가 필요 할 것으로 판단된다.
본 연구에서는 가열처리에 따른 노로바이러스 농도변화를 관찰하기 위하여 굴을 노로바이러스에 자연감염 시킨 후 가열처리조건별에 따라 노로바이러스의 농도변화를 분석하였다. 노로바이러스 GI형 및 GII형의 초기농도가 7.22×103 및 2.43×103 copies/g인 감염굴을 60, 68, 70, 100℃에서 각각 15분간 처리 하였으며, 그 결과를 감소율로 나타내었다. GI형의 경우 전 구간에서 80% 이상의 감소율을 나타내었으며, GII형은 67-90%의 감소율을 나타내었다. GI 및 GII형 모두 높은 온도에서 처리할 경우 더 높은 감소효과를 나타내는 것으로 확인되었지만, 가열처리 후에도 일부 유전자가 검출되는 것으로 나타났다. 바이러스는 72℃ 이상의 온도로 처리할 경우 외피단백질이 파괴된다고 알려져 있으나, 본 연구에서는 100℃에서 15분 처리시에도 바이러스의 유전자가 검출되는 것으로 나타났다. 이는 굴의 지방질과 같은 성분들이 노로바이러스의 외피단백질이 파괴되는 것을 보호한 결과로 판단된다(Nuanualsuwan et al., 2002). 노로바이러스는 감염가를 판단할 수 있는 숙주세포가 개발되지 않아 가열처리 등에 의한 불활성화 연구는 murine calicivirus(MCV)나 feline calicivirus (FCV)와 같은 대체바이러스에 집중되어 있다(Bozkurt et al., 2013; Nims and Plavsic, 2013). 이러한 연구들에서 MCV나 FCV를 65℃에서 2분 또는 72℃ 1분간 가열하였을 경우 감염가가 6.7 Log10 이상 감소한다는 보고가 있어 본 연구의 가열처리 후 검출된 노로바이러스들도 상당수는 감염력을 상실했을 것으로 판단된다. 또한 본 연구에서 가열처리에 사용된 굴은 자연감염을 통해 다소 높은 농도의 바이러스가 감염된 굴이므로 실제 해역에서 분리되는 농도와 같이 초기농도가 낮은 샘플은 가열처리에 따라 그 감소율이 더 높아 질 수 있을 것이다. 따라서 바이러스의 감염농도를 조절하여 추가적인 연구가 진행되어야 할 것으로 판단된다.
본 연구는 대체바이러스가 아닌 노로바이러스에 대한 직접적인 연구결과로서 감염된 굴의 가열처리 조건에 대한 농도변화를 보고하였다. 이러한 연구결과는 식품에서의 노로바이러스 처리에 관한 기초적인 결과로 모니터링에 의한 정량분석 결과와 함께 노로바이러스의 위해평가자료에 활용될 것으로 판단된다. 또한 식품에서의 노로바이러스 식중독의 감염농도를 구명하기 위하여 지속적인 정량분석과 역학조사가 진행되어야 할 것으로 사료된다.