김(Pyropia; 기존 Porphyra속에서 학명이 변경됨. Sutherland et al., 2011)은 우리나라의 대표적인 양식 수산물로서, 다양한 형태의 수산식품으로 판매되고 있다. 최근 한국, 일본 및 중국에서는 주요 종인 방사무늬김의 생산성과 품질을 높이기 위하여 다양한 신품종을 개발하고 있으며, 수출용 김의 비중도 늘리고 있다(Noh, 2014). 이에 따라, 개발된 신품종의 유통과정에서 품질 유지를 위한 수산식품 관리시스템의 중요성이 강조되고 있다. 국내외에서 판매되고 있는 김은 마른김 뿐만 아니라 조미김 등 다양한 형태로 유통되고 있으며, 특히 김을 주재료로 가공한 조미김 중에는 ‘파래김’, ‘감태김’ 등으로 특성화한 상품도 판매되고 있다.
조미김인 ‘파래김’과 ‘감태김’은 생산과정에서 녹조 파래류(Ulva속)의 해조류를 김 가공과정에 첨가하여 제조한 것이다. ‘파래김’의 경우는 파래류(Ulva spp.)를 포함한 것으로 알려져있으나 정확한 종명은 알려진 바 없으며, ‘감태김’은 파래류 중 ‘감태’를 포함하는 것으로 판매되고 있으나 이에 대한 실체는 분석된 바 없다. 특히 ‘감태김’의 경우, U. prolifera가 해조류목록에서 ‘감태’로 보고된 적이 있으나, 현재는 ‘가시파래’로 분류학적으로 처리되어 ‘감태’라는 종명(국명)을 가진 파래류는 존재하지 않는다(Lee and Kang, 2001; Bae, 2010). 또한, 갈조류 감태(Ecklonia cava)와 동일한 이름으로 인해서 수산 가공식품 유통에서 혼동을 유발할 수 있다. 따라서, 파래류를 포함하는 김 가공식품인 ‘파래김’과 ‘감태김’의 품질 관리를 위해서는 식품에 포함된 해조류의 정확한 종분류학적 실체를 파악할 필요성이 있다.
파래류(Ulva속)는 국내에서 14종이 자생하고 있으나 형태적인 변이가 많아 형태·해부학적 특징 만으로는 정확한 동정에 한계가 있다(Bae, 2010). 최근 해조류의 계통분류학적 연구가 활발히 진행됨에 따라 생물종이 가진 특정 유전자 부위를 종동정(species identification)의 분자마커(molecular marker)로 개발하여 이용하는 기술이 널리 보급되고 있다(Maggs et al., 2007; Saunders and Moore, 2013). 해조류의 종간 구분에 활용되고 있는 분자마커로는 핵유전체의 ITS, 18S rDNA 부위와 엽록체 유전체의 rbcS (Rubisco small subunit coding gene) 및 rbcL (Rubisco large subunit coding gene), 미토콘드리아 유전체의 cox1 등이 사용되고 있으며(Lee et al., 2001; Maggs et al., 2007), 다양한 염기변이 정보를 활용한 DNA 바코드 연구도 활발이 진행되고 있다(Zuccarello et al., 1999; Lee et al., 2011; Saunders and Moore, 2013). 또한, 미토콘드리아를 비롯한 세포소기관 유전체 해독을 통하여 유용해조류 품종 구분을 위한 분자마커 개발 연구도 진행되고 있다(Wang et al., 2013; Hwang et al., 2013; Hwang et al., 2014).
분자마커는 기존 계통분류학적 연구뿐만 아니라 다양한 분야의 연구에 활용되고 있는데(Hebert et al., 2003; Hebert and Gregory, 2005), 특히 생물종의 분자동정(molecular identification)은 농수산식품에 포함된 생물종의 동정 및 기원 추적에도 활용될 수 있다(Lee et al., 2002; Wasser et al., 2007; Agrimonti et al., 2011). 수산식품에서는 주로 어류의 종 동정에 분자마커가 활용되어 왔으나(Lockley and Bardsley, 2000; Hanner et al., 2011) 해조류 가공식품에 대한 연구는 미진한 실정이다.
국내외에서 다양한 형태의 가공식품으로 소비되고 있는 김의 경우, 가공된 상태로 유통되기 때문에 원래 생물종이 가진 형태를 유지하고 있지 않을 뿐 아니라 다른 종류의 해조류가 포함되어 있는 경우도 있다. 본 연구에서는 엽록체 유전자를 기반으로 DNA 바코드 분자마커를 개발한 후, 이를 이용하여 현재 유통되고 있는 조미김 중 ‘파래김’과 ‘감태김’에 포함된 파래류에 대하여 정확한 종분류학적 실체를 규명하고자 하였다.
가공식품으로서 조미김에 포함된 파래류의 종 동정을 위하여 시판되고 있는 ‘파래김’ 3종류와 ‘감태김’ 1종류를 구입하였다. 유전자 분석과정은 Lee et al. (2001, 2011)에서 제시된 방법을 이용하였다. 확보된 김 시료는 Tissue Lyzer (Qiagen, Germany)를 사용하여 마쇄하고, DNeasy Plant Mini Kit (Qiagen)을 이용하여 total genomic DNA를 추출하였다. 추출된 total genomic DNA에는 김 유래의 DNA와 파래류 유래의 DNA가 모두 포함되어 있으므로, PCR 과정에 사용되는 프라이머조합에 홍조와 녹조 특이적인 프라이머를 도입하여 김과 파래류 유전자를 각각 분리하였다. PCR반응은 amfiXpand PCR master mix (GenDEPOT, USA)를 사용하였으며, 반응조건은 95℃에서 3분 처리한 후, denaturation (94℃/30초), annealing (50℃/30초) 및 extension (72℃/1분) 과정을 40회 반복하였으며, 최종 72℃에서 7분간 final extension 과정을 수행하였다(Thermal Cycler 9700; Applied Biosystems, USA).
본 연구에서 김과 파래류의 분자동정(molecular identification)을 위한 분자마커로 엽록체 유전자인 rbcL을 선정하였다. 김의 rbcL 유전자 기반 분자마커 개발을 위해서 김 유전자 증폭에 적합하도록 rbcL 유전자의 5’ 말단을 증폭하는 프라이머들을 새로이 디자인하였으며(forward primer, rbcL-2F-Por; reverse primer, rbcL-772R-Por), 이를 위해 GenBank에 등록된 방사무늬김의 엽록체 유전체 전체 서열을 참조하였다(NC_007932). 한편, 파래류의 유전자 증폭을 위해서는 녹조 특이적인 프라이머 조합 rbcL-7F-Ulva와 rbc590 (Hayden et al., 2003)을 사용하였는데 이 중 forward 프라이머인 rbcL-7F-Ulva는 본 연구에서 파래류 증폭을 위하여 새로이 디자인한 것이다. 또한, ‘파래김’과 ‘감태김’에 포함되어 있는 것으로 추정된 잎파래와 가시파래의 염기변이를 기초로 하여, rbcL 유전자 부위 중 두 종에 특이적인 짧은 단편을 증폭하기 위하여 forward 프라이머를 제작하였다.
PCR 산물은 정제 후 시컨싱을 수행하였으며(Genotech, Korea), 시컨싱 결과인 chromatogram은 Sequencher 5.2.4 소프트웨어(Gene Codes Corparation, USA)를 이용하여 assembling 한 후 contig서열을 추출하였다. 결정된 rbcL 염기서열은 Gen-Bank database (National Center for Biotechnology Information, NCBI)의 BLAST 검색기능을 사용하여 database 내에 축적된 염기서열 정보들과 비교 분석하였다. 염기서열 비교분석에는 GenBank database 중 정확한 종명을 포함하고 있고 분자계통분류학적 연구를 통해 검증된 시료의 염기서열만을 사용하였으며, Sequencher 5.2.4와 MEGA 5.2.2 (Kumar et al. 1994)프로그램을 이용하였다.
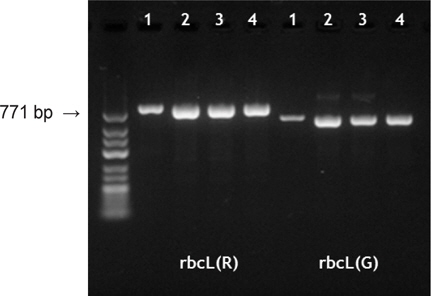
시판 중인 조미김 중 ‘파래김’과 ‘감태김’에서 추출한 total genomic DNA를 주형으로 김과 녹조류에 각각 특이적인 프라이머 조합을 이용하여 성공적인 PCR 반응결과를 얻었다(Fig. 1). 김을 대상으로 한 PCR은 rbcL 유전자 증폭용 프라이머 조합 rbcL-2F-Por (5'-TGTCTCAATCCGTAGAATCAC-3')와 rbcL-772R-Por (5'-CTCTCTCATACATATCTTCCAT-3')을 제작하여 적용한 결과, 김 특이적 771 bp의 rbcL 유전자가 증폭되었다(Fig. 1). 증폭된 rbcL 유전자 염기서열 분석 결과는 김속 식물을 대상으로 진행된 Hwang et al. (2005)의 연구결과 중 한국산 방사무늬김(Pyropia yezoensis)과 100% 일치하였다. 한편, 녹조 파래류 rbcL 증폭을 위하여 forward 프라이머(rbcL-7F-Ulva, 5'-CCACAGACTGAAACAAAAGC-3')인 rbcL-7F-Ulva를 새로이 디자인하여 적용하였으며, rbcL-7FUlva와 rbc590 프라이머 조합을 이용하여 조미김에서 584 bp 의 크기의 PCR 밴드를 확인하였다(Fig. 1). 증폭된 파래류 기원rbcL 유전자를 시컨싱한 결과, ‘파래김’ 유래 rbcL 염기서열은 GenBank에 보고된 파래 계통분류학적 연구결과 중 잎파래[U. linza; Japan (AB741533, AB830520), China (HM046607)]와 100% 일치하였고, ‘감태김’은 가시파래[U. prolifera; China (JQ867403, FJ042888, HM046608), Japan (AB598810, AB830521) 등]와 100% 일치하였다. 조미김 유래의 염기서열과 GenBank 참고서열을 비교분석한 결과, 잎파래와 가시파래간에는 증폭된 rbcL 유전자 말단의 프라이머 서열들을 제외한 544 bp 염기서열 중 단 1개의 서열에서 차이가 나타났다. 즉, rbcL 유전자 5'말단의 개시코돈부터 465번째 염기가 잎파래(U. linza)에서는 'A'로, 가시파래(U. prolifera)에서는 'G'로 분석되었다(Fig. 2). 이러한 종간 염기변이는 분석된 시료와 참고서열 모두에서 일관성 있게 나타났으므로 분류학적으로 두 종을 구분할 수 있는 식별형질(informative site)로 확인되었다.
본 연구에서 발견된 rbcL 유전자의 염기서열 차이를 기초로하여 잎파래와 가시파래에 각각 특이적인 PCR 증폭을 위하여 염기변이를 포함하는 forward primer를 제작하였으며[U. linza specific primer, rbcL-448Fms1-Ulin(5'-CCACCGCATGGTATTCCA-3'); U. prolifera specific primer, rbcL-448Fms1-Upro(5'-CCACCGCATGGTATTCCG-3')], 이러한 종특이적 forward 프라이머들과 기존의 rbc590 조합을 적용한 결과 ‘파래김’에서는 잎파래 특이적인 프라이머(rbcL-448Fms1-Ulin)만 반응하였고, ‘감태김’에는 가시파래 특이적인 프라이머(rbcL-448Fms1-Upro)만 반응함으로써 143 bp 크기의 증폭결과를 얻을 수 있었다(Fig. 3). 50℃, 55℃ 및 60℃로 annealing temperature를 조정한 결과, 50℃에서는 rbcL-448Fms1-Upro의 경우 낮은 특이성이 나타났으나 rbcL-448Fms1-Ulin은 높은 특이성을 나타내었다(Fig. 3). 60℃에서는 PCR 반응산물이 전혀 나타나지 않은 반면 55℃에서는 두 개의 프라이머 모두 안정적인 종특이적 PCR 밴드를 보였다.
본 연구에서는 가공식품인 조미김에 포함된 김과 파래류를 정확하게 동정하기 위한 분자마커로서 rbcL 유전자 부위를 도입하였다. 이는 핵 유전체 기원의 분자마커인 18S rDNA와 ITS 대신에 엽록체 기원의 분자마커를 발굴하여 적용한 것인데, 김을 포함하는 홍조식물과 파래류를 포함하는 녹조식물은 엽록체 유전체의 진화양상에서 뚜렷이 구분되는 계통군을 형성하므로(Yoon et al., 2002), 혼입된 시료에서도 엽록체 유전체 분자마커들을 동시에 적용함으로써 김과 파래류 기원 rbcL 유전자를 각각 성공적으로 추출·분석하는 것이 가능하였다.
rbcL은 해조류 종간 유연관계 및 계통분류학적 연구에 활용되어 왔고(Maggs et al. 2007) GenBank에 비교 분석이 가능한 많은 염기서열이 포함되어 있다. 우선 김 특이적인 rbcL 분자마커를 이용하여 조미김(‘파래김’ 및 ‘감태김’)에서 추출된 rbcL 염기서열을 분석한 결과, 김은 방사무늬김(Pyropia yezoensis)으로 확인되었다. 현재 국내에서 양식되고 있는 방사무늬김은 한국 자생품종과 일본품종으로 구분되며 ‘파래김’과 ‘감태김’에 포함되어 있는 김은 그 중 자생품종으로 확인되었는데, 이로써 분자마커를 이용하면 종 수준 뿐 만 아니라 양식품종까지 정확히 동정할 수 있음을 시사해주고 있다. 방사무늬김 외에도 국내에서 양식되는 김에는 잇바디돌김(Pyropia dentata)과 모무늬돌김(Pyropia seriata)이 있으나, 분석된 ‘파래김’과 ‘감태김’에서는 발견되지 않았다. 조미김과 같이 가공된 상태의 시료는 DNA가 손상되는 문제점이 있으나 본 연구에서 사용된 rbcL 증폭 프라이머 조합을 이용하여 성공적으로 분석가능 하였으므로, 향후이 프라이머 조합은 높은 효율의 증폭결과를 요구하는 시료의 연구에 널리 활용될 수 있을 것으로 판단된다.
한편, 녹조류 분자마커를 이용하여 ‘파래김’과 ‘감태김’에 포함된 파래류 특이적인 rbcL 유전자 서열들을 발굴하였다. 본 연구에서 조사된 시료를 기준으로 볼 때 현재 ‘파래김’으로 시판되고 있는 조미김에는 잎파래(U. linza)가 사용되고 있고, ‘감태김’에는 가시파래(U. prolifera)가 사용되고 있는 것으로 확인되었다. 또한, 갈조류 감태(Ecklonia cava, 다시마목)의 종명이 ‘감태김’에 잘못 사용되고 있으므로(Lee and Kang, 2001; Bae, 2010), 앞으로 이러한 분류학적 종명과 실제 유통되는 해조류 명칭 간의 오류는 수정되어야 할 것으로 판단된다.
잎파래와 가시파래에 특이적으로 제작된 프라이머는 ‘파래김’과 ‘감태김’에서 각각 잎파래와 가시파래만 특이적으로 반응하였다(Fig. 3). 특히, 잎파래와 가시파래를 구분하는 염기변이와는 별도로 프라이머 서열 3'말단에 인위적으로 염기변이를 추가하였는데(3’-end mismatch primer; Kwok et al., 1990), 이 프라이머는 PCR 과정에서 DNA 주형과 결합하는 55°C annealing temperature에서 각종에 대한 높은 특이성을 나타내었다. 최근 일본 연구팀이 수산가공식품에 포함된 파래류의 종 감별을 위하여 분자마커를 개발한 바 있다(Kawashima et al., 2013). 이들의 연구는 파래류로만 구성된 수산식품을 대상으로 하였으나, 잎파래와 가시파래를 구분할 수 있는 수준의 해상력을 제공하지 못하였다. 이에 비해 본 논문에서 적용된 분자마커는 조미김에 포함되어 있는 김 유전자와 혼입된 상태에서도 파래류 를 확인할 수 있을 뿐 아나라, 잎파래와 가시파래 각각을 정확히 동정할 수 있는 해상력을 갖고 있다. 따라서, 개발된 잎파래 및 가시파래 특이적 프라이머 조합은 조미김 등 시판되고 있는 수산가공식품 내에 포함된 파래류 감별에 유용한 분자마커로 활용될 수 있다.
본 연구에서는 전통적인 분자계통분류 연구를 위하여 준비된 생체 및 건조시료가 아니라 여러 단계의 과정을 거쳐 식품으로 가공되어 판매되고 있는 가공식품을 대상으로 유전자 분석을 시도하였다. 식품으로 가공되는 과정에서 김과 파래류가 가지고 있는 유전자가 손상되어 있음에도 불구하고, 본 연구에서 개발된 분자마커는 조미김에 포함된 파래류의 종류를 정확히 동정할 수 있었다. 따라서, 향후 이러한 DNA 바코드 기반 분자마커 기술은 다양한 수산가공식품에 포함된 생물종에 대하여 정확한 종 정보와 품질관리를 위한 자료를 제공할 뿐 아니라, 분자마커의 해상력이 좀 더 높아진다면 수산식품의 원산지 추적에서도 중요한 역할을 담당할 수 있을 것으로 기대된다.





![rbcL region PCR products using Ulva species specific primers [Samples (1-3 'Parae-gim', 4 'Gamtae-gim')]. Forward primers [Ulin: U. linza specific primer (rbcL-448Fms1-Ulin), Upro: U. prolifera specific primer (rbcL-448Fms1-Upro)].](http://oak.go.kr/repository/journal/14788/KSSHBC_2014_v47n5_522_f003.jpg)