양식 넙치에서 주로 발생하는 연쇄구균증은 매년 반복적으로 출현하며, 특히 Edwardsiella tarda등과의 혼합 감염이 빈번하여 우리나라의 양식 산업에 큰 피해를 주고 있는 것으로 알려져 있다(Kim et al., 2003). 넙치를 비롯한 해산 양식 어류의 연쇄구균증 주요 원인균으로 Streptococcus parauberis, S. iniae , Lactococcus garvieae가 관여하는 것으로 보고되었으며(Baeck et al., 2006), 최근에는 특히 S. parauberis의 검출 빈도가 뚜렷 하게 증가하고 있다. 이러한 연쇄구균증의 치료를 위하여 양식현장에서는 많은 양의 항생제가 사용되고 있으며, 더구나 새로운 돌연변이체에 의한 어병 발생이 증가함에 따라 항생제의 사용 빈도수가 현재 점차 증가추세에 있다. 현재 현장에서 사용되는 quinolone계 항생제는 nalidixic acid, oxolinic acid 그리고 fluoroquinolone 계로 분류할 수 있는 ciprofloxacin, enrofloxa-cin, norfloxacin, pefloxacin 등을 포함하고 있다.
Quinolones는 주로 세균의 DNA 합성에 필요한 효소인 DNA gyrase(topoisomerase II)와 topoisomerase IV의 작용을 억제하여 DNA 복제를 저해함으로 항균작용을 나타내며(Gellert et al., 1976), DNA gyrase 는 DNA 복제, 재조합, 전사에 필수적인 효소로써 DNA 복제시 DNA의 ATP-dependent negative supercoiling을 촉매하며 gyrA와 gyrB subunit으로 구성된다. Topoisomerase IV는 염색체 복제의 마지막 단계에서 염색체를 분리하는데 필수적인 성분으로 DNA decatenating 및 relaxing activity를 나타내며 gyrA 와 gyrB에 상응하는 parC와 parE subunit으로 구성된다(Drlica and Zhao, 1997).
이러한 quinolone계 항생제에 내성을 보이게되는 기전은 DNA gyrase 유전자인 gyrA와 gyrB 상의 염기변이, topoisomerase IV의 일부인 parC, parE 상의 염기변이, 특히 관련 4개 유전자 각각에서 가장 중요한 부위이며 quinolone 내성 결정 부위인 quinolone resistance-determining region (QRDR)에서의 변이가 가장 중요한 요소로서 작용하는 것으로 알려져 있다(Piddock, 1999; Tanaka et al., 2000).
High-resolution melting (HRM)기법은 PCR 수행 중 생성되는 amplicon에 intercalating fluorescent dye를 결합시키고, 그 후 온도 상승을 통해 target 부위의 염기서열에 따라 각기 다르게 발생하는 florescence signal의 변화 특성을 분석하는 방법이다(Kristensen and Dobrovic, 2008; Tajiri-Utagawa et al., 2008; Wittwer et al., 2003). 따라서 항생제 내성균의 내성 유전자에서 나타나는 mutation의 분석은 HRM 기법의 적용을 통해 보다 빠르고 손쉽게 확인할 수 있을 것이다.
본 연구에서는 어류 양식현장에서 연쇄구균증 원인체로서 대부분을 차지하고 있는 S. parauberis의 quinolone에 대한 내성정도를 현장에서 분리된 균주 및 in vitro 변이주를 사용하여 비교 분석하였다. 또한 내성균주에서 나타나는 quinolone 내성 관련 유전자의 변이를 HRM 분석을 이용하여 효율적으로 규명 할 수 있는 방법을 개발하였다.
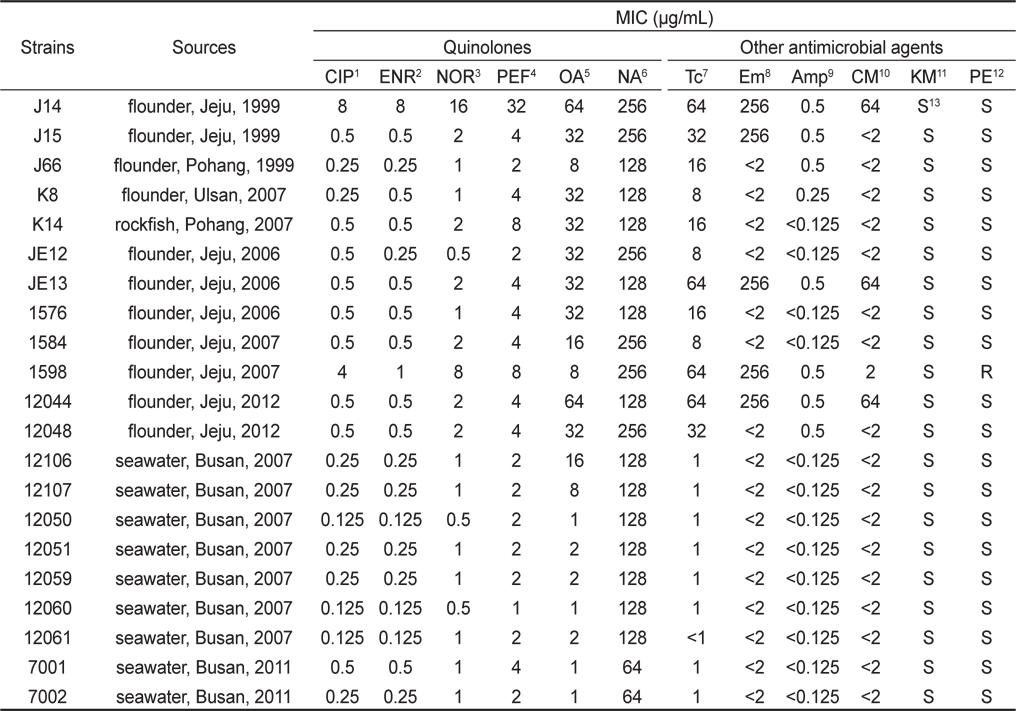
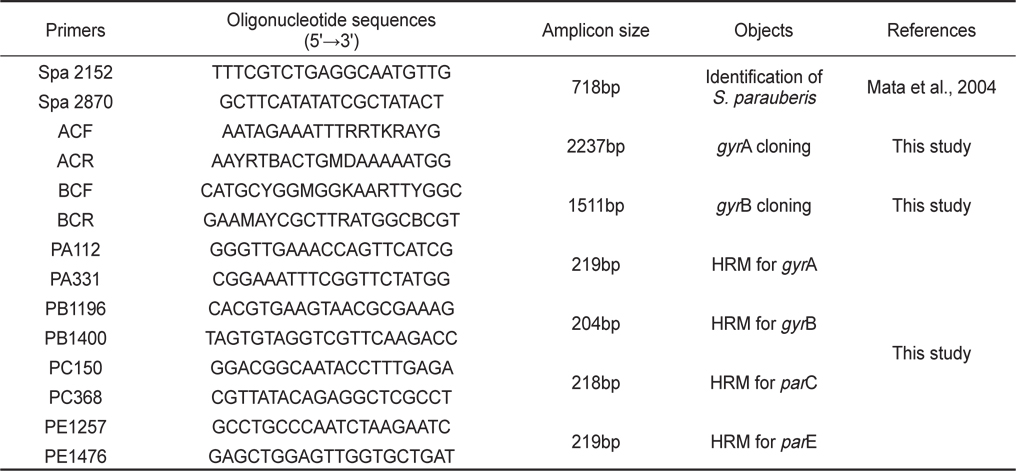
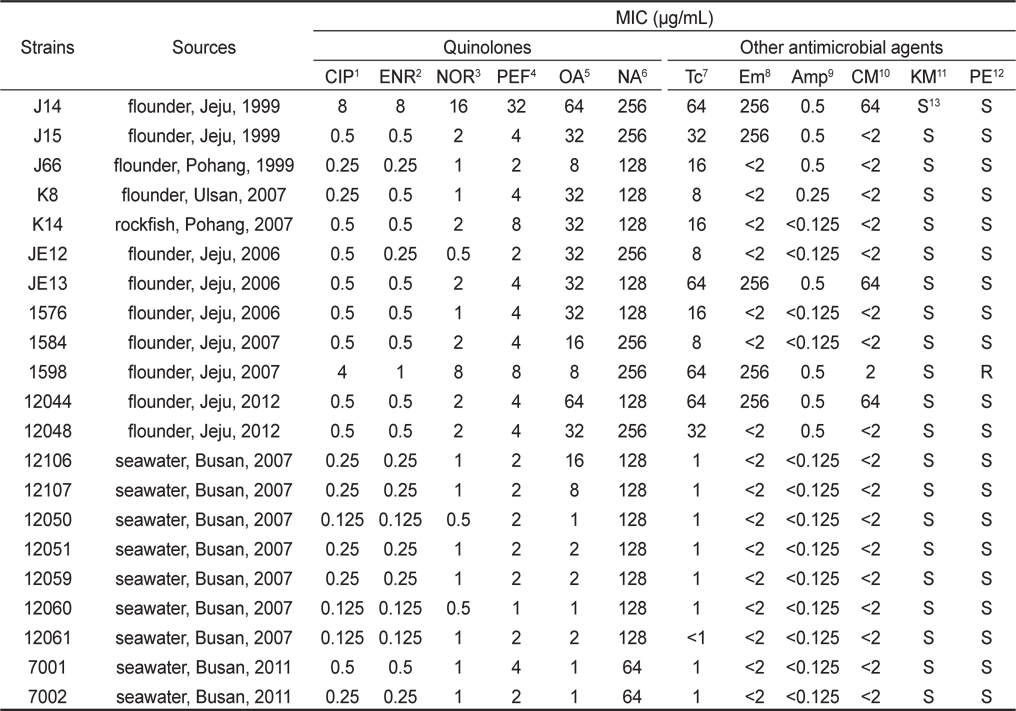
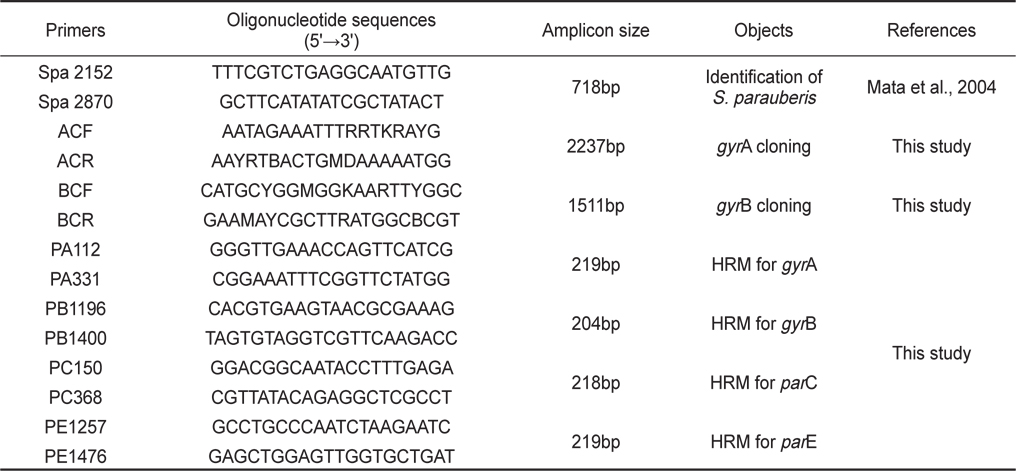
본 실험에서 사용된 21개의 S. parauberis 균주들은 1999년에서 2012년까지의 기간 동안 우리나라 여러 지역의 넙치, 돌돔을 비롯한 병어에서 전형적으로 연쇄구균병의 증상이 나타나는병어의 비장조직과 해수로부터 분리하였다(Table 2). 균의 분리 및 배양에는 1% 의 NaCl을 첨가한 Tryptic Soy Agar (TSA, Difco, USA)및 Brain Heart Infusion Agar (BHIA, Difco, USA)를 이용하였다. 병어의 조직과 해수는 TSA 및 BHIA에 접종하여 28°C에서 24-48시간동안 배양하였다. 이후 TSA 및 BHIA에서 순수 분리된균은 형태학적 검사로써 colony형태와 현미경 상에서의 균의 형태, 크기 및 운동성을 관찰하여 간이 동정하였고, S. parauberis에 특이적인 primer를 사용해 PCR을 실시하여 추가적인 동정을 실시하였다(Table 1). 또한 실험 균의 보존을 위해 배양액에 20% glycerol (Sigma–Aldrich Korea, Korea)를 첨가한 후, -70 °C에서 사용 전까지 보관하였다.
Quinolone 계 항생제 중 fluoroquinolne인 ciprofloxacin (CIP), enrofloxacin (ENR), norfloxacin (NOR) 그리고 pefloxacin (PEF) 4종과 oxolinicacid (OA), nalidixic acid (NA) 2종, 그리고 비 quinolone계 항생제로서 tetracycline (Tc), erythromycin (Em) ampicillin (Amp) chloramphenicol (CM) kanamycin (KM) 그리고 penicillin (PE) 6종 (Sigma–Aldrich Korea, Korea)에 대한 S. parauberis의 내성 수준을 Minimum Inhibitory Concentration (MIC)값을 broth dilution 법으로 측정하여 결정하였다(Gruson, 2005).
Ciprofloxacin 내성 분리 균주를 대상으로 HiGeneTM Genomic DNA prep kit For Bacteria (Biofact, Korea)를 사용해 제조사의 manual에 따라 total DNA 추출을 실시하였다. Topoisomerase 관련 유전자의 검출을 위한 PCR은 10× PCR buffer 2 μ L, 200 μ M 의 각각의 dNTP, 1 μ M의 sense primer와 antisense primer, Taq DNA polymerase (Cosmogentech, Korea) 및 DNA template 1 μL를 첨가한 후 distilled water로 최종액의 volume이 20 μL가 되도록 하였다. 이때 사용한 primer set는 gyrA, gyrB 와 parC, parE 의 QRDR을 포함하는 부위를 target으로 하였으며 HRM 분석에서도 적절한 길이가 형성 될 수 있도록 PCR 산물의 크기가 약 200 bp가 되도록 primers를 제작하였다(Table 1). PCR 혼합물은 Perkin-Elmer 2400 thermal cycler (Perkin-Elmer, Norwalk, CT, USA)를 사용하여 94°C에서 3분간 pre-denaturation 시킨 후, 94°C에서 30초 denaturation, 55°C에서 30초 annealing, 72°C에서 30초 extension의 반응을 30 cycle 수행한 후 72°C에서 7분간 post-extension시켰다.
PCR 생성물은 Gene-allTM GEL SV elution kit (General biosystem, Korea)를 사용하여 agarose gel로부터 분리, 정제한 후 pGEM-T Easy Vector System I (Promega, USA)를 사용하여 제조사의 manual에 따라서 클로닝 시켰다. Competent cell (E. coli DH5α) 100 μL와 혼합하여 열충격에 의하여 transformation을 실시한 후 IPTG 1 mM, X-gal 40 μ g/mL과 Amp 50 μ g/mL이 첨가된 Luria-Bertani (LB, Difco, USA) agar 배지에 도말하여 흰색의 집락을 선택하여 Amp 50 μ g/mL 이 첨가된 LB broth 배지에 배양하였다. 배양액 1.5 mL로부터 Gene-all TM plasmid SV mini kit (General biosystem, Korea)를 사용하여 플라스미드를 분리하였다. 염기 서열 분석은 Big Dye Terminator Cycle DNA Sequencing Kit (Applied Biosystems, USA)를 사용하여 분리된 플라스미드 내에 삽입된 염기서열을 밝힌 후, MACAW program (Version 2.0.5., National Center for Biotechnology Information, National Institutes of Health, Bethesda, MD, USA)을 사용하여 비교분석하였다.
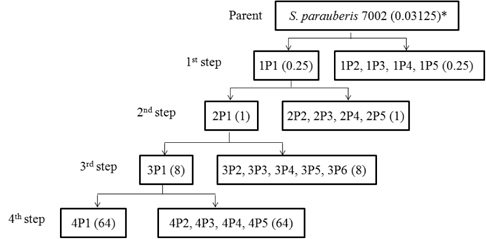
실험균주로는 보유 균주 중 ciprofloxacin을 포함한 다른 quinolone 계열 항생제들에 대해 가장 낮은 MIC를 보이는 S. parauberis 7002 균주를 wild type으로 사용하였으며 항생제가 첨가된 Mueller Hinton (MH, Difco, USA) agar 배지를 이용하여 항생제에 단계적으로 노출시켰다. 먼저 wild type의 균주를 MH broth에서 16-18시간 배양 후 각 plate당 MIC농도의 1-16배의 ciprofloxacin이 첨가된 MH plate에 도말하였다. 집락이 나타나는 최고농도의 항생제 배지에서 5개의 집락을 분리하고 그 중 하나의 균주(1P1)를 MH broth에 배양하였다. 이러한 과정을 1st step mutant 분리로 하였다. MH broth에서 2-3회 계대배양된 균주는 다음 단계인 2nd step mutant 분리에 사용하였다(Fig. 1). 또한 3rd 그리고 4th step의 mutant 분리도 연속적으로 동일한 방법을 이용하여 실시하였다.
HRM을 시행하기 위한 반응액에는 10 μL의 MeltDoctor HRM Master Mix, 3.5 mM MgCl2, 5 μM 의 sense primer 와 5 μM의 antisense primer, template (20 ng/ μL) 1 μL를 첨가 후 distilled water로 최종액의 volume이 20 μL가 되도록 했다. HRM 분석은 StepOne Real-Time PCR System (Applied Biosystems, USA)을 사용하였고 아래와 같은 방법으로 실시하였다.
먼저 95°C에서 5분간 pre-denaturation 시킨 후, 95°C에서 10초 denaturation, 56°C에서 10초 annealing, 72°C에서 15초 extension의 반응을 45 cycles 수행하였다. 총 45 cycles 반응이 끝난 후 60-90°C에서 0.1°C /s의 속도로 Tm (melting temperature) 값을 측정하였다. melting-curve 분석은 High-Resolution Melt Software (Ver. 3.0) (Applied Biosystems, USA)를 사용하였다.
1999년에서 2012년까지 분리된 S. parauberis의 21개의 균주 중 2균주 (10%)가 CIP에 대해 내성을 나타내는 것을 확인 할 수 있었다(Table 2). 그러나 내성균 모두는 12종의 현장 분리균에서 나타났으므로, 실제 현장 분리균에서의 내성균 비율은 17% 로 나타났다. 현재 S. pneumoniae를 제외하고 다른 Streptococcus spp.에 대한 ciprofloxacin의 MIC 판독 기준은 European Committee on Antimicrobial Susceptibility Testing (EUCAST) 및 Clinical Laboratory Standards Institute (CLSI)에서도 확립된 바가 없다. S. pneumoniae의 경우 ciprofloxacin 내성에 대한 breakpoint를 EUCAST 에서는 2 μ g/mL 이상으로, CLSI는 4 μ g/mL 로 규정하고 있다(EUCAST. 2011, CLSI. 2010). 따라서 현장 분리 quinolone 내성균의 판별은 CIP에 대한 MIC 값이 2 μ g/mL 이상인 것으로 하였다. 또한 비록 현재까지 S. parauberis 의 OA에 대한 내성 기준에 대한 보고는 없지만, 현장 분리균에서 OA-MIC가 8 μ g/mL을 기준으로 내성균과 wild type 두 group으로 나눌 수 있었다(Table 2). 그러므로 OA-MIC가 8 μ g/mL을 기준으로 14종의 균주 중 67% 정도가 내성을 나타내었으며 특히 12종의 현장 분리균 전부가 내성균으로 판명 되었다. OA는 수산 생물에의 사용이 허가된 수산용 항균제 중 하나로서 이전부터 많이 사용되어져 왔으므로 내성의 정도가 다소 높게 나타나는 것으로 보여지며, 이후 개발된 CIP는 OA에 비해 항균력이 더 높으며 누적 사용량 또한 적으므로 내성의 기준이 낮게 나타난 것으로 보여진다(Table 2). 뿐만 아니라 21개의 균주 중 현장 분리균 12개 균주 모두는 Tc에 대한 내성을, 5종의 균주는 Em, 그리고 Amp, Cm등에 대한 내성도 나타내어 현장 분리균 모두는 다재 내성균으로 판별 되었다. 그러나 수중 생태계에서 발견된 7균주 모두는 사용된 항생제에 대하여 내성을 나타내지 않아 수권 환경이 아직까지는 안전한 상태에 있음을 추정 할 수 있었다.
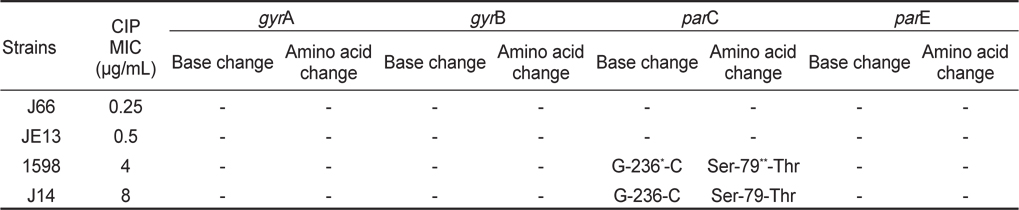
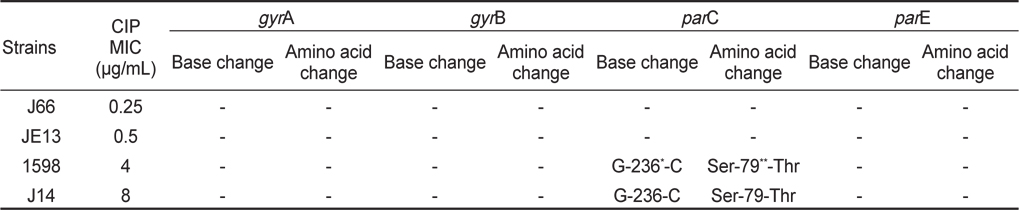
현장 병어에서 분리된 CIP 내성의 S. parauberis 중 MIC가 0.25-8 μ g/mL 을 보이는 대표적인 4 개의 isolates를 임의로 선택하여 type II topoisomerase관련 유전자인gyrA, gyrB, parC, par E gene 각각의 QRDR 부위를 target로 한 PCR을 실시하였다. 생성된 amplicon을 pGEM-T Easy Vector에 cloning 하고 그 염기서열을 분석함으로써 내성 획득과 각 유전자에서의 mutation 특성 변화와의 상관 관계를 분석하였다.
선택 균주 4종 중 CIP-MIC가 4 μ g/mL 이상으로 나타나는 2개 내성균의 QRDR-parC 부위에서 Ser-79 → Thr으로의 amino acid 변이를 관찰할 수 있었다. 그러나 gyrA, gyrB, 그리고 parE gene의 QRDR 부위의 염기 서열에서는 변이를 발견 할 수 없었다(Table 3). 또한 CIP-MIC가 0.5 μ g/mL 이하를 보이는 현장 분리균 12044 및 K14의 topoisomerase 관련 유전자 모두에서도 QRDR 부위에서의 유전적 변화를 발견 할 수 없었다(data not shown).
Wild type의 S. parauberis 7002를 CIP가 첨가된 MH agar에서 단계별로 총 4 단계를 거쳐 내성균을 유도하여 in vitro CIP-내성 mutant를 분리하였다(Fig. 1). 단계별로 선택된 4종의 변이 내성균들 (1P1, 2P1, 3P1 및 4P1)에 대한 CIP-MIC 분석 및 topoisomerase II 및 IV 유전자 각각에 대한 PCR을 실시하여 QRDR에 대한 염기 서열을 분석하였다(Table 4). 그 결과 두번째 단계에서 CIP-MIC가 1 μ g/mL 을 나타내는 mutant 2P1의 gyrA (C-242-T)와 parE (C-1345-A)의 nucleotide 변이가 검출되었다. 그러나 gyrA의 변이는 아미노산의 변이가 없는 Ser-81 → Ser 의 silent 변이였고, parE이 변이는 QRDR부위의 5 ‘ end 쪽에 있는 부위로서 Arg-449 → Ser 의 변화를 보여 주었다. 또한 세 번째 단계에서의 mutant 3P1 은 parC 79번 부위에서 변이가 일어나 현장 분리 내성균(Ser-79 → Thr)과 동일한 위치였으나 그 변이는 Ser-79 → Ile 으로 나타나 새로운 변이 형태를 보여 주었다. 3P1의 CIP-MIC는 8 μ g/mL를 나타내어 2P1의 MIC에 비하여 8배로 증가하는 명확한 내성 증가를 확인할 수 있었다.
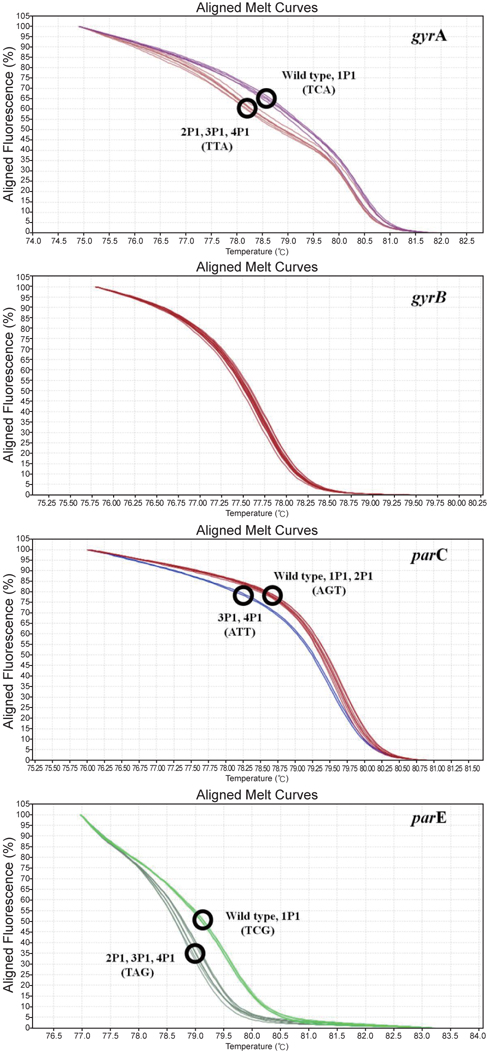
HRM 분석은 DNA 합성시 염기가 결합할 때 생기는 녹는 점의 차이로 인하여 염기가 하나만 틀려도 signal이 다른 양상으로 나타나기 때문에 CIP-내성균의 topoisomerase 유전자의 QRDR 부위에서 나타나는 mutation을 검출하고 분석하는 목적에 활용하였다. gyrA, gyrB, parC, parE gene에서 주요 변이가 나타나는 QRDR의 염기서열이 포함되도록 제작한 primer를 이용하여 (Table 1), in vitro mutant 를 test한 결과에서 변이가 나타난 gyrA, parC, parE gene이 변이가 일어나지 않은 균주와 구별되는 melting curve 를 나타내는 것을 확인할 수 있었다(Fig. 2). 그러므로 본 연구의 primers를 이용한 HRM 분석방법은 S. parauberis의 type II topoisomerase를 encoding 하는 각 유전자들의 확인과 QRDR에서의 변이 또한 비교 할 수 있음을 확인 할 수 있었다.
본 연구에서는 1999년에서 2012년까지의 기간 동안, 우리나라 여러 지역의 넙치, 돌돔을 비롯한 병어로부터 연쇄구균증을 일으키는 원인균인 S. parauberis를 분리하였으며 이에 대해 quinolone의 내성을 조사하고 내성 획득의 원인을 규명하고자 하였다. 이전의 연구들에 따르면 연쇄구균증에 있어 S. iniae와 더불어 L. garvieae가 주요 원인체로 보고되어 왔다(Nakatsugawa, 1983, Heo et al., 2001). 그러나 최근에는 S. parauberis가 몇 년간 검출 빈도가 뚜렷하게 증가하여 현재 가장 다수를 차지하며, 우리나라에서 빈번히 연쇄구균증을 발생시키는 원인체로 보고되고 있다. 또한 그 발생 양상이 제주를 중심으로 우리나라 전역으로 확산되고 있어 피해가 심각한 것으로 알려져 있다(Baeck et al., 2006).
DNA gyrase와 topoisomerase IV의 gyrA, parC 유전자에서 변이는 quinolone 내성기작에 우선적으로 중요한 요소이며, gyrB와 parE 역시 내성에 관여하는 요소이다(Pan and Fisher, 1998). 현재까지 S. parauberis의 topoisomerase 관련 유전자에 대한 특성 분석은 이루어지지 않고 있다. 그리고 본 연구실에서 그람 양성균의 gyrA 와 gyrB 에서의 conserved region에 대한 degenerated primer (Table 1)를 사용하여 2237 bp 와 1511 bp (GenBank number; GU324257.1 와 GU324260.1)를 cloning 하였으나 Nho et al. (2011)의 S. parauberis full sequence에 대한 특성 규명에서 gyrA 와 gyrB 로 추정 ∙ 지정한 부위와의 염기 배열이 gyrA는 98.8%, gyrB는 99.3% 동일하였다. 그러므로 parC 와 parE 의 염기 배열도 본 연구의 isolates의 것들과 동일할 것으로 추정 할 수 있어 이에 대한 유전자 cloning은 실시하지 않고 Nho et al. (2011)의 유전자 염기 배열을 사용하여 PCR primer를 제작하였다. 이러한 primer sets는 S. parauberis의 DNA gyrase 및 topoisomerase IV 유전자들의 변이 특성 및 정도를 알아보기 위하여 각 유전자들의 QRDR의 염기 서열을 분석하기 위한 PCR 과 HRM 분석에 사용하였다.
S. pneumoniae의 경우 parC의 Ser-79와 gyrA의 Gly-81, Ser-83의 아미노산 변이가 quinolone 내성의 가장 주요한 변이로 알려져 있다(Pan and Fisher, 1996). 이러한 보고를 바탕으로 현장에서 분리한 quinolone 내성 S. parauberis의 parC와 gyrA를 포함한 QRDR 부위의 mutation을 분석한 결과 parC의 Ser-79 부위에서 Thr으로의 변이가 나타났으며, parC 이외의 gyrA, gyrB, parE 의 QRDR 부위에서는 다른 변이가 관찰되지 않았다.
이러한 변이에 대한 분석과 함께 in vitro 상에서 quinolone 에 대해 내성균을 유도하는 실험을 실시하였다. Wild type 인 7002를 ciprofloxacin이 첨가된 MH 배지에 단계적으로 노출시킴으로써 실험적으로 유전자들의 변이를 유도하는 실험으로 각각 총 4 단계를 거쳐 mutant 를 제작하였다. 각 단계별로 5 균주를 임의적으로 선택하여 gyrA, gyrB, parC, parE gene의 아미노산 변이를 분석한 결과 (Table 4), 먼저 2번째 단계에서 parE (Arg-449 → Ser)의 변이가 나타나고, MIC 값은 4배로 증가하였다. 가장 quinolone 내성에 변화를 많이 나타내는 것으로 알려진 gyrA 에서의 변이는 nucleotide의 변이는 있었지만 아미노산 변이는 나타나지 않았고 이는 silent mutation으로서 ciprofloxacin에 대한 내성의 증가는 gyrA와는 큰 연관성이 없을 것으로 보여진다. parE의 경우 그 위치에 있어 QRDR region의 mutation hot point에서 벗어났으며 Mycoplasma pneumoniae를 제외한 다른 균주에서 보고된 적이 없다(Gruson et al., 2005). 또한 parE 자체가 일반적으로 quinolone 내성에 크게 영향을 미치지 않는다고 보고되어 왔으므로 큰 의미를 가지지 않을 것으로 생각되지만 이에 대해서는 이후 깊이 있는 연구가 이루어져야 할 것이다. 따라서 S. parauberis의 quinolone 내성은 세 번째 단계에서 나타난 parC (Ser-79 → Ile)의 변이가 오히려 중요한 영향을 미칠 수 있었을 것으로 추정한다. 즉, 이러한 결과는 quinolone 의 내성에 있어서 E. coli를 포함한 그람 음성균에 서는 DNA gyrase가 첫 번째 target이 되지만, Streptococcus와 같은 그람 양성균에서는 오히려 topoisomerase IV 가 첫 번째 target이 되어, DNA gyrase 에서의 변이 보다는 parC나 parE에서의 변이가 내성 증가에 우선적으로 영향을 미친다는 보고와 일치한다 (Pan and Fisher, 1998). 다만 S. pneumoniae와 S. suis 등을 포함한 여러 Streptococcus에서 parC의 79번 아미노산이 내성 획득에 관여함이 보고되어 왔으나(Pan and Fisher, 1996; Escudero et al., 2007), 아직 parE의 변이가 첫 번째의 target이 된다는 보고는 없었으므로 이에 대한 명확한 규명이 필요할 것으로 보인다. 더불어 현장 분리균에서 나타난 Ser-79 → Thr 변화 보다는 in vitro mutants에서 나타난 Ser-79 → Ile의 변화가 보다 높은 수준의 내성을 나타내어 QRDR 부위 hydrophobicity 증가가 S. parauberis의 내성 수준을 더욱 높게 하는 중요한 요소가 될 수 있는 가능성을 제시하고 있다고 생각한다. 또한 4P1이 3P1과 동일한 parC 변이를 나타내었으나 4P1의 Ciprofloxacin에 대한 MIC 값은 8배 이상 증가하는 것으로 나타나, quinolone계 항생제에 대한 내성증가는 topoisomerase IV 뿐만 아니라 또 다른 quinolone 내성 발생기작인 efflux pump, plasmid-mediated quinolone resistance (PMQR) 그리고 균의 생리적 변화 등도 함께 영향을 미칠 수 있음을 보여주고 있다.
HRM 분석은 M. pneumoniae의 mutation에 의한 macrolide 내성을 확인하는데 유용하게 사용되었다는 보고가 있었으나 (Wolff et al., 2008), DNA gyrase 또는 topoisomerase 유전자 변이 분석에 적용한 연구는 지금까지 보고되지 않았다. 본 연구에서는 gyrA, gyrB, parC, parE의 QRDR 부위에서 변이가 일어나는 염기서열이 포함되도록 primer를 만들어 HRM 분석에 이용하였다. 그 결과 변이를 보이는 균과 변이를 보이지 않는 균의 염기서열과는 다른 HRM signal 양상을 보여 wild type과 mutation type이 정확하게 구별 가능하였다 (Fig. 2). 그러므로 QRDR에서의 유전자변이를 HRM으로 screening하는 방법은 현장에서 많은 수의 quinolone 내성 isolates를 분석하여야 하는 경우 gyrA, gyrB, parC, parE에서 나타나는 QRDR의 변이를 빠르고 효율적으로 규명 할 수 있는 편리한 방법이라고 할 수 있다.
본 연구에서는 연쇄구균증의 주요 원인균인 S. parauberis의 quinolone 내성 획득에는 parC의 우선적 관여와 함께 parE의 관여 가능성까지도 제시하였다. 그리고 HRM을 이용하여 type II topoisomerase 유전자인 gyrA, gyrB, parC 그리고 parE의 변이를 간단하고 효율적으로 파악할 수 있게 하였다.