After an outbreak of viral disease in an aquafarm, release of virus (es) from infected fish into environmental seawater has been suspected. In the present study, we utilized a negatively charged membrane (HA type) as an efficient method for concentration and detection of fish pathogenic viruses, specifically, megalocytivirus and viral hemorrhagic septicemia virus (VHSV) present in field-collected seawater samples or inoculated into seawater artificially. Positively charged viruses adsorbed onto the negatively charged membrane and were eluted with 1 mM NaOH (pH 10.5) following rinsing with 0.5 mM H2SO4 (pH 3.0). Megalocytivirus and VHSV particles isolated using anegatively charged HA membrane from seawater inoculated with each virus at a concentration of 10 viral particles/ mL were of sufficient quantity to show positive results in atwo-step PCR (or RT two-step PCR); however, despite it being negatively charged, a cellulose acetate (CA) membraneshowed negative results.In quantitative PCR, the detection limits of the HA membrane for megalocytivirus and VHSV in seawater were 1.20E+00 viral particles/ mL and 1.22E+01 viralparticles/mL, respectively. The calculated mean recovery yields from 1 L seawater spiked with known concentrations of megalocytivirus and VHSV particles were 28.11% and 23.00%, respectively. The concentrate of a 1-L sample of culturing seawater from the aquatank of flounder suffering from VHSV showed clear positive results in PCR when isolated with an HA, but not a CA, membrane. Thus, viral isolation using an HA membrane is a practical and reliable method for detection of fish pathogenic viruses in seawater.
경제적으로 가장 많은 피해를 끼치고 있는 어류 및 갑각류 바이러스성 질병에 대한 치료대책은 현재까지 없으며, 질병확산에 가장 큰 영향을 미치는 환경에 대한 정확한 이해와 분석을 통하여, 그 위험성을 최소화하는 방법만이 최선의 예방법으로 생각되고있다.
국내의 경우 매년 고수온기에 megalocyvirus에 속하는 돌돔 이리도바이러스(RBIV)로 인한 돌돔의 대량폐사로 경제적 손실이 매우 크며(Jung and Oh, 2000), 저수온기에는 viral haemorrhagic septicemia virus (VHSV)로 인한 넙치의 감염 및 대량폐사가 발생하고 있는 실정이다 (Kim et al., 2009). 매년 주기적으로 발생하고 있는 megalocytivirus, VHSV와 같은 바이러스성 질병의 경우 발병이 되기 시작하면 주변 양식장으로의 전파가 매우 빠르며 이에 대한 적절한 관리방안이 필요하다.
바이러스성 질병에 감염된 어류나 갑각류의 대량폐사 시 환경수 중으로 많은 양의 바이러스를 배출하게 되며 배출된 바이러스는 환경수 내에서 확산되어 다른 감수성 종에 대한 재감염 가능성이 높다. 즉 바이러스성 어류질병은 사육환경에 바이러스가 존재하다가 감수성이 있는 어종에 노출 시 발병되는 경우가 일반적이다(Jones and Groman, 2001; EI-Matbouli and Soliman, 2011). 따라서 질병 발생지역의 오염된 환경 사육수 그리고 감염어류 및 갑각류로부터 외부로 유출된 바이러스의 존재여부를 확인하는 것은 어류 및 갑각류의 바이러스성 질병예방을 위해 매우 중요하다고 볼 수 있다. 그러나 환경수 중 소량으로 존재하는 바이러스의 검출을 위해서는 최적화 된 바이러스의 효율적 농축방법이 필요하며, ethanol 침전법, PEG 침전법, ultracentrifugation법, 한외여과막법 및 charged membrane을 사용한 filtration법이 일반적으로 알려져 있다(Kitamura and Suzuki, 2000; Lewis and Meticalf, 1988; Oh et al., 2000; Katayama et al., 2002). 이중 charged membrane을 사용하는 흡착 방법은 Poliovirus, Norovirus, Hepatitis A virus (HAV)등의 enteric virus (Katayama et al., 2002; Liu et al., 2007) 및 Adenovirus (Haramoto et al., 2007) 등의 사람에게 감염되는 바이러스에 대하여 보편적으로 많이 적용되어 왔으며, 수산생물질병제어를 목적으로 환경수중의 바이러스의 농축과 검출에 대한 연구로는 White spot syndrome virus (WSSV), Cyprinid herpesvirus 3 (CyHV-3)을 대상으로 한 연구가 있다 (Song et al., 2003, Minamoto et al. 2009). 하지만 국내에서 만연하고 있는 megalovytivirus와 VHSV등에 대한 어류질병바이러스의 환경수 중에 대한 농축 및 검출에 대한 본 방법의 적용 가능성 여부 및 응용에 관한 연구는 아직 이루어 지지 않고 있는 실정이다.
따라서 본 연구에서는 우리나라에서 가장 경제적으로 큰 손실을 입히고 있는 DNA 바이러스인 megalocytivirus, 그리고 RNA 바이러스인 VHSV에 대해 negatively charged membrane을 적용하여 환경수 중에 미량으로 존재하는 바이러스 검출을 위한 보다 편리한 바이러스 농축기법을 개발하고자 하였다. 그리고 농축법의 검출한계 분석 및 VHSV 발생 양식장의 유입수와사육수에 대한 분석을 실시하여 현장 적용에도 접근하였다.
DNA virus인 megalocytivirus와 RNA virus인 VHSV를 각각 grunt fin (GF)와 chinook salmon embryo-214 (CHSE-214) cell line에서 배양하였다. DNA virus인 megalocytivirus의 경우, 본 go연구실에서 2000년 9월에 남해안에서 샘플링한 양식돌돔비장에서 분리한 IVS-1 strain (Jeong et al., 2003; GenBank accession numbers for ATPase gene : AF487899)을 GF cell에 접종하여 배양하였다. VHSV의 경우 2008년 2월에 감포에서 샘플링한 양식 넙치의 신장조직을 CHSE-214 cell에 접종하여 20℃에서 배양하였다. 사용된 VHSV는 G protein gene 분석으로 type IVa로 확인하였다(data not shown).
접종한 각 cell line에서 3-5일 배양한 후 cytopathic effect(CPE)가 일어나 부착된 세포가 떨어지면 회수하여 냉동과 해동의 과정을 3번씩 수행한 후 1,500
해수 농축 방법으로는 Katayama 등(2002) 이 제시한 negatively charged membrane (0.45 μm pore)을 사용한 방법을 변형하여 해수 내에 존재하는 바이러스를 농축하고자 하였다.
GF와 CHSE-214 cell에서 배양된 megalocytivirus IVS-1 strain과 VHSV의 바이러스 상등액을 바이러스가 존재하지 않는 해수 1 L에 1.00E+01 viral particles/mL 이 되도록 접종하였다.
바이러스를 접종한 해수 1 L를 glass microfiber filters (GF/C, 1.2 μm pore) 와 Negatively charged membrane으로 여과하여 바이러스가 여과막에 흡착되도록 하였다. GF/Cmembrane은 전여과지로 사용되었으며, negatively charged membrane은 CA membrane (Cellulose acetate membrane, 0.45 μ m pore) 또는 HA membrane (Nitrocellulose membrane, 0.45 μm pore)을 단일 또는 이중으로 여과하여 사용하였다.
이후 0.5 mM H2SO4를 흘려주어 양이온을 씻어낸 뒤 여과막에 1 mM NaOH 10 mL을 첨가하여 흡착된 바이러스를 회수 하였으며, 회수된 시료를 50 mM H2SO4로 중화한 뒤 100× TE buffer로 안정화시켰다. 10 mL로 농축시킨 시료를 Amicon Ultra-15 Centrifugal Filter Unit with Ultracel-30 membrane(Millipore)을 사용하여 3,000 g에서 10분 이상 원심분리하여 200 μL로 농축하였다. 농축된 200 μL를 nuclease-free 증류수에 1 mL로 현탁하여, 이중 200 μL로부터 DNA와 RNA를 분리하였다.
최적화된 방법의 모색을 위해 GF와 CHSE-214 cell에서 배양된 megalocytivirus IVS-1 strain과 VHSV의 바이러스 상등액을 바이러스가 존재하지 않는 해수 1 L에 1.00E+01 viral particles/mL 이 되도록 접종하였다. 그리고 바이러스 농축을 수행하였으며, 농축된 시료로부터 2-step PCR을 사용하여 바이러스 검출을 확인함으로써 negatively charged membrane에 따른 효율을 비교하였다.
농축한 해수 시료 1 mL 중 200 μL을 사용하여 DNA와 RNA를 각각 분리하였다. Viral DNA를 분리하기 위해 AccuPrepⓇ Genomic DNA Extraction Kit (Bioneer, Lorea)을 사용하였으 며, Viral RNA를 분리하기 위해서는 RNeasyⓇPlus Mini Kit(QiagenHiden, Germany)를 사용하였다. 각각 제조사의 protocol에 따라서 DNA와 RNA를 분리하여 50 μL의 TE buffer에 현탁하였다. 분리된 nucleic acids는 분광 광도계 (BioPhotometer, Eppendorf)를 사용하여 흡광도 측정으로 A260/A280 nm값을 구하여 DNA와 RNA의 양을 측정하였으며, 실험 전까지 −20℃에서 보관하였다.
cDNA 합성을 위하여 M-MLV reverse transcriptase 1 μL (Promega), 5×buffer 2 μL, dNTP 2 μL, Random hexamer primers 1 μL (Promega), RNasinⓇRibonuclease inhibitor (Promega) 1 μL, extracted total RNA 1 μL을 넣고 total volume이 10 μL이 되게 Nuclease-free water을 첨가한 후, 42℃에서 60분, 99℃에서 5분간 반응시켰다. 여기서 만들어진 cDNA는 PCR 반응의 template로 사용하였다.
1-step PCR (Applied Biosystems 2720 Thermal Cycler)은 아래와 같은 방법으로 실시하였다. 10×PCR buffer 2 μL, 200 μM의 각각의 dNTP, 1 μM의 sense primer와 1 μM의 antisense primer (Table 1), Taq DNA polymerase (Taq DNA polymerase, Cosmo, Korea) 및 template 1 μL (DNA 및 cDNA)를 첨가한 후 distilled water로 최종액의 volume이 20 μL가 되도록 했다. PCR 혼합물은 94℃에서 3분간 predenaturation시킨 후, 94℃에서 30초 denaturation, 55℃에서 30초 annealing, 72℃에서 30초 extension의 반응을 30 cycle 또는 35 cycle 수행한 후 72℃에서 7분간 post-extension 시켰다. 2-step PCR amplification은 1-step PCR product 1μL로 사용하여 위와 동일한 방법으로 실시하였다.
[Table 1.] Primers used in this study

Primers used in this study
PCR 후 증폭 산물은 0.5 μg/μL EtBr (Ethidium Bromide)이 첨가된 2% agarose gel을 이용하고, 1×TAE buffer (40 mM Tris-acetate, 1 mM EDTA)를 전기영동을 위한 완충액으로 하여 전기영동을 실시하였다. UV 검출기에서 나타나는 band를 관찰하여 그 증폭여부를 확인하였다.
Megalocytivirus에 감염된 돌돔으로부터 분리된 IVS-1 isolate의 genomic DNA를, 그리고 VHSV에 감염된 넙치로부터 분리된 VHSV IVa subtype의 cDNA를 각각 template로 하여 1-step PCR (30 cycle)을 각각의 바이러스에 대한 특이적인 primer set와 함께 실시하였다(Table 1). 생성된 PCR product (megalocytivirus의 경우 163 bp, VHSV의 경우 157 bp)를 GeneAllⓇExpin Gel SV kit (GeneAll Biotechnology, Korea)를 사용하여, agarose gel로부터 각각 분리 및 정제하였다. 정제된 DNA를 pGEM-T Easyvecotor (Promega, USA)에 ligation 후
준비된 standard DNA를 Quant-iTTMPicoGreenⓇdsDNA Reagent and Kits (Invitrogen Co., Carlsbad, CA, USA)를 사용하여 무게를 측정하고, 이로부터 plasmid copy 값을 결정한 후 DNA virus인 megalocytivirus의 경우 2.00E+05-2.00E+01 copy, 그리고 RNA virus인 VHSV의 경우 5.00E+04-5.00E+00copy로 각각 10-fold 씩 단계 희석하여 표준 검량 곡선을 작성하는데 사용하였다.
농축한 해수 시료에 오염된 megalocytivirus와 VHSV의 정량적분석을 위해 qPCR (Rotor-GeneTM 6000, Corbett Re-search, AUS)을 실시하였다. qPCR을 실시하기 위한 조건은 10×buffer 2 μL, 200 μM의 각각의 dNTP, 1 μM의 각각의 primer, Hot start Taq (HS prime Taq DNA polymerase, Genet Bio, Korea) 및 template (시료로부터 추출한 DNA, 준비한 cDNA, 10-fold 씩 희석된 plasmid standard)를 0.2 mL의 tube에 넣은 후 최종적으로 EvaGreen (Biotium, Korea) 1 μL를 첨가하였다. qPCR은 95℃에서 10분간 pre-denaturation 시킨 후, 95℃에서 10초 denaturation, 55℃에서 15초 annealing, 72℃에서 20초 extension의 반응을 40 cycle 수행하였다. 40 cycle이 끝난 후 72-95℃에서 1℃/sec의 속도로 T
바이러스가 존재하지 않는 것으로 확인된 해수 1 L에 배양된 megalocytivirus IVS-1 strain과 VHSV IVa를 각각 1.20E-01 viral particles/mL seawater부터 1.20E+02 viral particles/mL seawater의 농도로 인위 오염시켰다. 농도 별로 바이러스가 오염된 해수 시료를 가장 검출률이 좋았던 GF/C+HA membrane을 사용하여 이중여과 및 농축하였으며, 농축시료 1 mL에 존재하는 viral particles을 qPCR로 정량하여 바이러스 회수율을 산출하였다.
2011년 2월에 VHSV에 감염된 제주도, 감포의 넙치 양식장에서 사육수 및 유입수를 채수하여 GF/C+HA membrane을 사용하여 해수를 농축하였으며 분리된 RNA를 대상으로 2-step PCR을 실시하였다. 사육수를 채취한 양식장은 VHSV에 감염 된 넙치(10 cm)가 수온 15±0.5℃, 사육수 순환은 16-18 회/day 회전하여 환수하고 있는 10 m×10 m×1 m의 수조에서 양식되고 있었으며, VHSV에 감염되어 누적 폐사율이 50%(입식 후 15일 후) 정도일 때 채수를 실시하였다.
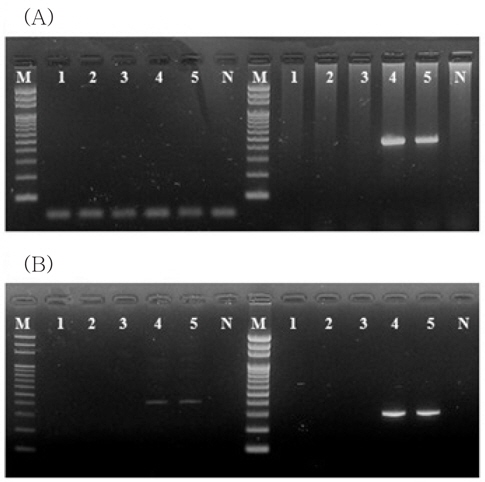
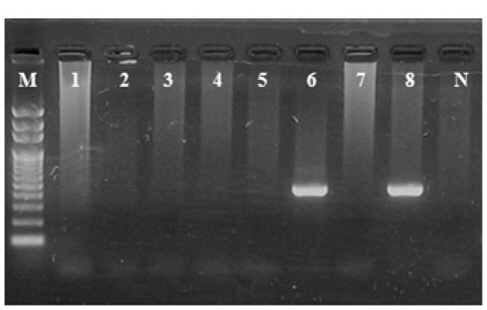
VHSV배양 상등액을 1.00E+01 viral particles/mL seawater 농도로 접종한 1 L의 해수로부터 VHSV를 검출 시, GF/C 또는 CA 각 하나의 membrane만을 사용하였을 경우 2-step PCR에서 검출되지 않았다. 그러나 HA membrane을 함께 사용한 GF/C+CA+HA 또는 GF/C+HA 여과를 실시하여 바이러스를 농축 후 분리된 total RNA를 대상으로 실시된 2-step PCR에서 VHSV에 특이적인 band를 전기영동상에서 확인할 수 있었다(Fig. 1A). 또한, IVS-1상등액을 1.00E+01 viral particles/mL seawater농도로 접종한 1 L의 해수 내의 megalocytivirus를 2-step PCR을 수행하여 검출하고자 하였을 경우, VHSV와 마찬가지로 GF/C 또는 CA 각 하나의 membrane만을 사용하였을 때는 검출이 되지 않았으나 GF/C+CA+HA 또는 GF/C+HA membrane을 사용한 바이러스 농축을 실시하였을 때는 명확한 megalocytivirus의 검출이 가능하였다(Fig. 1B). 따라서 GF/C+CA+HA 또는 GF/C+HA membrane을 사용하였을 때 검출이 가장 잘되는 것을 확인할 수 있었으며,이후의 실험에서는 GF/C+HA membrane을 사용하여 실험을 실시하였다.
이러한 결과는 1 L의 해수에 존재하는 어류질병바이러스(1.00E+01 viral particles/mL)의 검출 시 2가의 음이온으로 구성된 HA membrane을 사용하는 것이 1가 이온으로 구성된 CA membrane을 사용하는 것에 비해 더 적합함을 의미한다. 또한, pore size가 1.2 μm인 GF/C와 HA membrane을 이중으로 사용함으로써 해수 중에 존재하는 부유물과 유기물이 HA membrane에 흡착되는 것을 막을 수 있기 때문에 효율적으로 해수 여과 및 농축이 가능하였다.
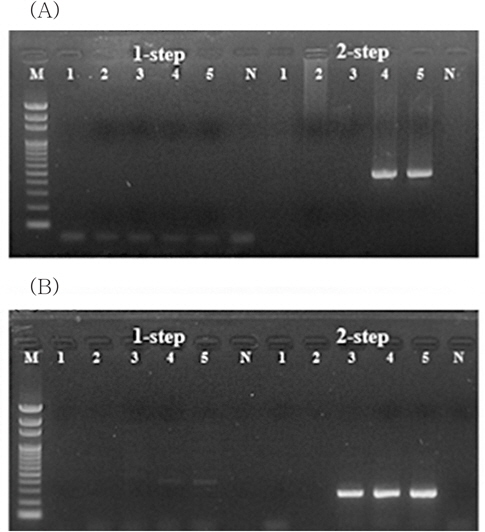
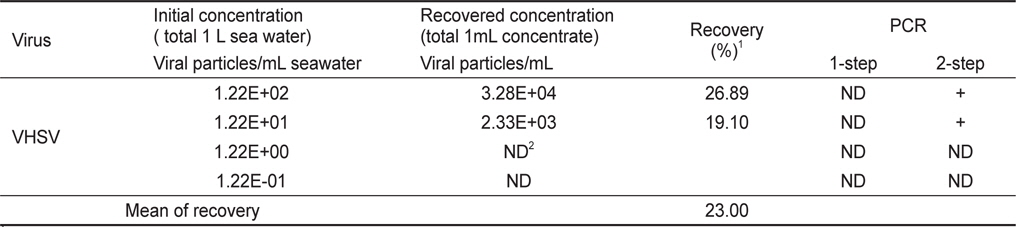
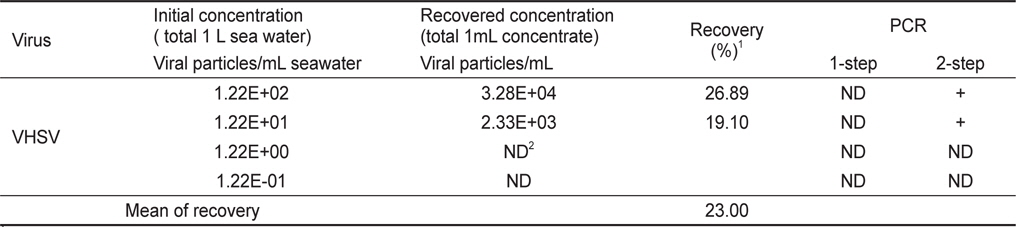
해수중의 VHSV의 검출한계를 분석하기 위해VHSV 배양액을 1 L의 해수에 1.22E+00 viral particles/mL seawater이하의 농도로 인위 오염시켜 1 mL로 농축한 시료에서는 2-step PCR과 qPCR에서 검출되지 않았다(Fig. 2A, Table 2). 그리고 1.22E+01 viral particles/mL이상의 농도로 1 L의 해수에 오염되어 있는 시료를 1 mL로 농축하였을 때 2-step PCR에서 검출이 가능하였으며, 이때 농축시료 1 mL당 3.28E+04, 2.38E+03 viral particles로 나타났다. 이를 통하여 1 L의 해수에 농도별로 접종한 VHSV의 회수율을 산출하였을 때 26.89%, 19.10%로 나타났다. 즉, 본 방법에 의한 평균 VHSV 회수율은 23.00%였으며, 해수 1 L에 존재하는 VHSV의 검출한계는 1.22E+01 viral particles/mL (1 mL로 농축 시 2.38E+03 viral particles)이었다.
[Table 2.] Recovery of virus from 1 L seawater spiked with VHSV
Recovery of virus from 1 L seawater spiked with VHSV
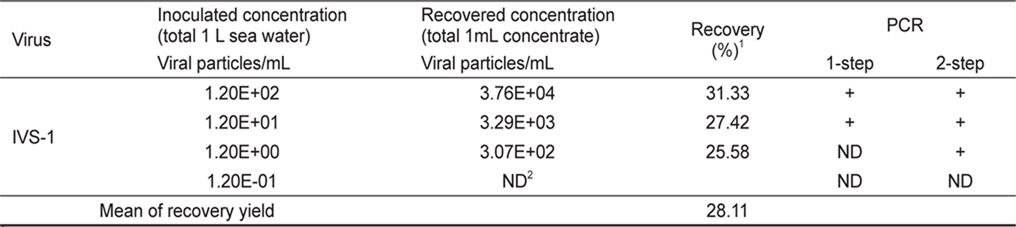
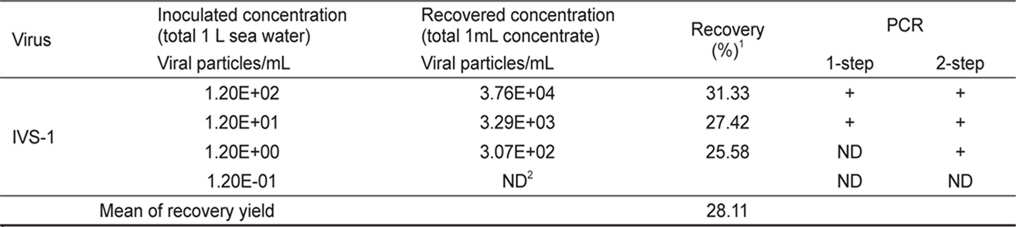
반면 megalocytivirus의 경우 1 L의 해수 중 IVS-1이 1.20E-01 viral particles/mL seawater의 농도로 오염되어 있을 경우 2-step PCR과 qPCR에서 검출되지 않았으며, 1.20E+00 viral particles/mL seawater의 시료에서는 2-step PCR에서 검출이 가능하였다. 또한 1 L의 해수 중에 IVS-1이 1.20E+01 viral particles/mL seawater이상으로 오염되어있을 경우 1-step PCR만으로도 전기영동 상에서 IVS-1에 특이적인 band를 확인할 수 있었다. 그리고 1.20E+00~1.20E+02 viral particles/ mL seawater의 농도로 IVS-1이 오염된 1 L의 해수를 1 mL로 농축할 경우, 농축 시료 mL당 각각 3.07E+02, 3.29E+03, 3.76E+04 viral particles의 바이러스가 회수되었으며, 이를 통하여 회수율을 산출하였을 때 25.58%, 27.42%, 31.33%로 나타났다. 즉 1 L의 해수를 사용할 경우 IVS-1에 대한 회수율은 평균적으로 28.11% 이었으며, 검출한계는 1.20E+00 viral particles/mL seawater이었다(Fig. 2B, Table 3).
[Table 3.] Recovery of virus from 1 L seawater spiked with IVS-1
Recovery of virus from 1 L seawater spiked with IVS-1
VHSV의 검출한계 농도(1.22E+01 viral particles/mL seawater)와 IVS-1의 검출한계 농도(1.20E+00 viral particles/mL seawater)를 비교해볼 때 IVS-1의 검출한계 농도에 있어서 IVS-1이 VHSV에 비해 약 10배 높은 민감성을 보였다.
본 연구에 있어 VHSV를 인위적으로 해수에 첨가한 후 GF/C+HA membrane으로 농축하여 결정한 viral particle값에 있어서, nucleic acid 분리 과정 중 RNA의 안정성 및 cDNA합성수율 문제에 의하여 소실량이 DNA virus에 비하여 높을 수 있으므로 실질적인 VHSV의 양은 본 연구에서 분석된 양보다 높을 가능성이 있다. 또한, 실제로 VHSV에 감염된 넙치양식장의 유입수와 사육수를 대상으로 GF/C+HA membrane을 사용하여 농축 후 바이러스의 오염 정도를 분석한 결과, 유입수에서는 바이러스를 검출할 수 없었으나 사육수에서는 VHSV의 검출이 가능하였다. 그러나 넙치의 VHSV발병 시 감염방법 및 어체의 크기에 따라 누적 폐사율이 일정치 않으며 (Kim et al., 2009; Isshiki et al., 2001), 일반적으로 바이러스 감염 시 질병의 회복기에는 바이러스의 수중 유출이 매우 감소하므로(Grant et al., 2011; Totland et al., 1996) 배출구 위치에 대비한 유입구의 위치, VHSV 질병의 진행정도 등을 고려한 보다 정밀한 분석이 필요할 것이며, megalocytivirus 감염 양어장 또한 비교 분석할 필요가 있을 것이다.
환경수 중 존재하는 바이러스의 회수율을 비교해 볼 때 바이러스 별로 회수율에 대한 차이가 나타나며, entericvirus인 poliovirus의 경우 90% 이상의 높은 회수율을 나타내는 반면, HAV, Feline calicivirus (FCV)의 경우 30-50%의 회수율을 보인다고 보고하고 있으며 어류질병바이러스인 Koi herpesvirus(KHV)의 경우 3.9%의 낮은 회수율을 나타내고 있다 (Hara-moto et al., 2009; Katayama et al., 2002).
2011년 2월에 VHSV에 감염된 제주도, 감포의 넙치 양식장에서 사육수 및 유입수를 채수하여 GF/C+HA membrane을 사용하여 해수를 농축하였으며, 분리된 RNA를 대상으로 2-step PCR을 실시하였다. 사육수를 채취한 양식장은 VHSV에 감염 된 넙치(10 cm)가 수온 15±0.5℃, 사육수 순환은 16-18 회/day 회전하여 환수하고 있는 10 m×10 m×1 m의 수조에서 양식되고 있었으며, VHSV에 감염되어 누적 폐사율이 50%(입식 후 15일 후) 정도일 때 채수를 실시하였다. VHSV에 감염된 넙치사육수 1 L시료를 GF/C+HA membrane을 사용하여 농 축할 경우 VHSV를 검출할 수 있었던 반면, GF/C+CA membrane을 사용할 경우 검출할 수 없었다. 또한 양식장의 유입수를 대상으로 GF/C+CA membrane과 GF/C+HA membrane을 사용하여 농축 후 바이러스의 오염정도를 분석한 결과 배출수 와는 달리 유입수에서는 바이러스를 검출할 수 없었다(Fig. 3).
본 연구에서는 어류질병바이러스의 확산에 가장 큰 영향을 미치는 환경에 대한 정확한 이해와 분석을 통하여 그 위험성을 최소화하기 위해, 국내에서 만연하고 있는 megalocytivirus, VHSV을 대상으로 해수 중 바이러스 농축 방법의 최적화 작업 및 그 검출한계점과 바이러스 회수율에 대한 분석을 실시하였다. GF/C와 HA membrane 을 사용한 해수 중 바이러스 농축법을 어류질병바이러스인 megalocytivirus와 VHSV를 대상으로 적용하였을 때 회수율이 28.11%, 23.00%로 각각 나타나 KHV에 비하여 우수한 결과를 보여주어 해수 중의 여러 어류 질병바이러스의 검출에도 본 방법을 적용할 수 있음을 입증하였다고 할 수 있다.