비타민 D 수용체(vitamin D receptor, VDR, NR1I1)는 리간드에 의해 활성화되는 전사인자인 핵수용체 superfamily에 속한다. 핵수용체는 공활성인자(coactivator) 및 공억제인자(corepressor)와 협동적으로 유전자의 발현을 조절한다. 핵수용체 단백질들은 특징적인 domain 구조를 공유하고 있는데 N-말단부터 부분적인 전사활성을 가진 A/B domain, DNA-binding domain (DBD, C domain), hinge D domain, ligand-binding domain (LBD, E domain) 및 공활성인자/공억제인자 결합 부위이며 전사활성을 나타내는 F domain으로 구성된다. VDR은 다른 핵수용체와 유사하게 레티노이드 X 수용체(retinoid X receptor, RXR)과 이형이량체(heterodimer)를 형성하고 각각의 리간드와 결합한 후 비타민 D 반응 영역(vitamin D responsive element, VDRE, PuGGT/GTCANNNPuGGT/GTCA)에 결합하여 최대한의 전사활성을 나타낸다(Sánchez-Martínez et al., 2006; Koszewski et al., 2010; Landry et al., 2011).
척추동물의 VDR은 콜레스테롤을 전구체로 하며 스테로이드 구조를 공유하는 1α,25-(OH)2-vitamin D3 (calcitriol; 1,25 D3)와 높은 친화력으로 결합하며 칼슘 및 인산염 항상성 조절과 같은 전형적인 calcitriol 효과를 나타낸다(Norman et al., 2002). VDR 유전자들은 포유동물, 조류, 양서류, 파충류, 경골어류 이외에도 무악동물 먹장어(
기능적인 비타민D (1,25 D3)는 자외선B (UVB)에 의해 7-dehydrocholesterol의 광이성질체에 기원을 둔 척추동물 진화 초기에 생체내에서 합성된 호르몬으로 생각되고 있다(Bouillon and Suda, 2014). 또한 1,25 D3는 UVB에 의한 DNA 손상을 예방해 줄 수 있는 해양동물의 효과적인 보호물질로 추정된다(Holick, 2011). 한편, 해양무척추동물 피낭류 유령멍게
본 연구에서는 해양무척추동물 VDR 유전자와 단백질 특성을 규명하기 위하여, 멍게(우렁쉥이,
멍게의 VDR cDNA
>
Polymerase Chain Reaction (PCR)
Full-length HrVDR를 cloning하기 위해, Magest (http://magest.hgc.jp/) database에 등록되어 있는 VDR partial sequence를 바탕으로
[Table 1.] Oligo primers for polymerase chain reaction

Oligo primers for polymerase chain reaction
Total cDNA를 template로 하여 VDR 5′말단과 3′말단 방향으로 각각, 10X BD advantage 2 PCR buffer 2.5 μL, dNTP mix (10 μM) 0.5 μL, 50X BD advantage 2 polymerase mix 0.5 μL, Universal Primer A Mix (UPM) (10X) 2.5 μL, forward primer (MAhrVDR-F), reverse primer (MAhrVDR-R) (10 μM 각각) 0.5 μL, 5′–RACE CDS Primer A, 3′–RACE CDS Primer A (Clontech Lab) (Table 1)를 사용하여 총 25 μL로 5′ RACE-PCR과 3′ RACE-PCR을 수행하였다. Pre-denaturing step 94℃ 3분, denaturing step 94℃ 30초, annealing step 72℃ 2분 5 cycle 실행한 뒤, 다시 denaturing step 94℃ 30초, annealing step 70℃ 30초, annealing step 68℃ 30초, extension step 72℃ 2분 25 cycle 실시한 다음, post-extension step 72℃ 7분 반응하였다. 총 35 cycle을 각각 실행하고 증폭된 DNA 산물은 1% agarose gel로 전기영동 분석하였다.
증폭된 PCR산물을 pGEM-T easy vector (Promega, Madison, WI, USA)에 삽입하고,
>
Luciferase reporter assay 분석
실험 24시간 전에 분주한 human embryonic kidney 유래의 HEK293 세포(5×104)에 HrVDR, HrRXR, human RXR (hRXR), human steroid receptor coactivator (hSRC1) (Maeng et al., 2012b) 및 전남대학교 이영철 교수님으로부터 제공받 은 human VDR 발현용 vector (hVDR), osteocalcin promoter 유래의 VDRE로 구성된 VDRE-luciferase reporter (VDRE-Luc) (Kim et al., 2009), β-galactosidase 발현용 pRSV-β-gal vector (Maeng et al., 2012a)를 LipofectamineTM 2000 (Invitrogen, Grand Island, NY, USA)으로 transfection하였다. 3시간 후에 10% FBS와 1% 항생제를 포함하는 배지로 교환하였다. 1α,25-(OH)2-vitamin D3 (1,25 D3), 7-dehydrocholesterol (7DHC), Cholecalciferol (CHOLC), Ergocalciferol (vitamin D2), 25-hydroxyvitamin D3 [25(OH)D3], 6-formylindolo [3,2-b]carbazole (FICZ), fanesoid X receptor agonist (GW4064), benzoic derivative retinoic acid (AM580), 9-cis retinoic acid (9-cis RA) (Sigma-Aldrich)를 EtOH에 희석하여 처리하고 24시간 동안 37℃, CO2 5% incubator에 배양하였다. 세포배양액 중의 EtOH 농도는 0.1%이하로 유지하였다. 24시간후, 1X cell lysis reagent (Promega)을 사용하여 세포를 용해 시키고, 실험군 간의 형질 도입률을 보정하기 위하여 β-galactosidase assay buffer (200 mM sodium phosphate buffer pH 7.3, 2 mM MgCl2, 100 mM β-galactosidase (98%), 1.33 mg/mL O-nitrophenyl-β-D-galactopyranoside (OPNG)를 처리하여 흡광도 405 nm에서 β-galactosidase의 활성을 조사하였으며, luciferase assay buffer (1M KH2PO4 pH 7.8, 0.5 M MgCl2, 0.1 M ATP, 10 mM luciferin)와 luminometer (Berthold Co., Wildbad, Germany)를 사용하여 luciferase reporter의 활성도를 측정하였다. HrVDR 벡터의 단백질 발현을 확인하기 위하여 HA-epitope를 인식하는 monoclonal 1차항체(mouse anti-HA, H9658, Sigma-Aldrich, St. Louis, MO, USA), goat 2차항체(anti-mouse IgG-HRP, sc2005, Santa Cruz Biotech., Dallas, TX, USA)를 각각 5,000배 희석하여 상법에 따라 western immunoblot을 실시하였다.
HrVDR을 암호화하고 있는 cDNA 전체 염기서열은 5′ 및 3′ RACE 방법으로 얻은 cDNA 산물을 NCBI ORF Finder와 Conserved Domain Search (CD Search) program, CLUSTAL W 및 MultAlin multiple sequence alignment program을 사용하여 분석하였다.
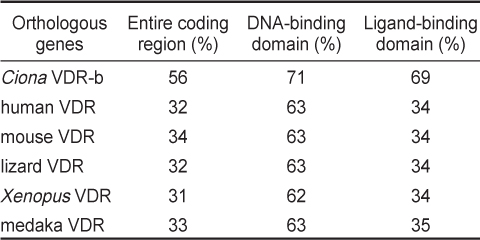
[Table 2.] Amino acid sequence identities with Halocynthia roretzi VDR, HrVDR
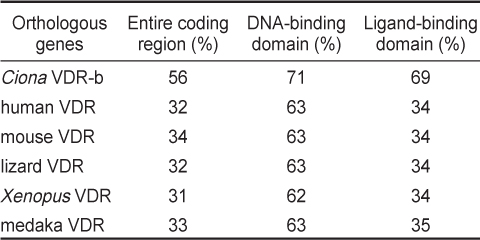
Amino acid sequence identities with Halocynthia roretzi VDR, HrVDR
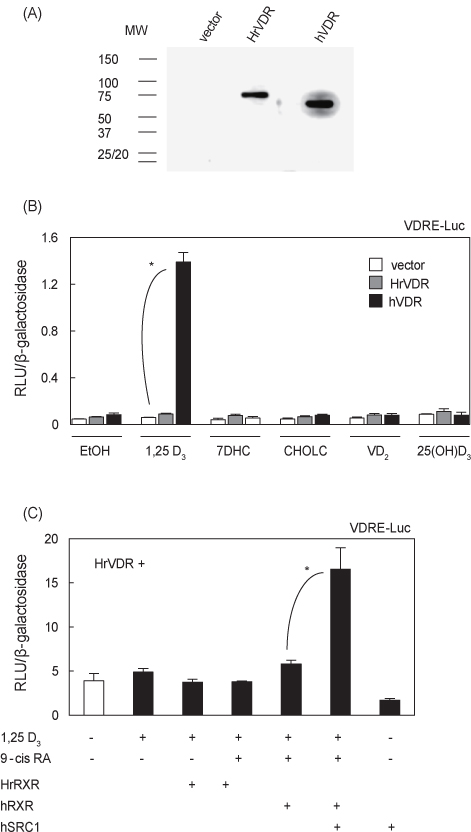
Luciferase reporter assay를 실시하기에 앞서 HEK293세포에 transfection된 HA-HrVDR 발현벡터가 발현되는지 HA epitope 항체를 이용한 western immunoblot 방법으로 조사하였다. 예상한 바와 같이 약 70 kDa 부근에 HrVDR 단일 밴드로 검출되었고, 동일하게 vehicle vector를 transfection한 세포에서는 밴드가 관찰되지 않았으며 hVDR 도입한 세포에서는 약 60 kDa 부근에 나타났다(Fig. 3A).
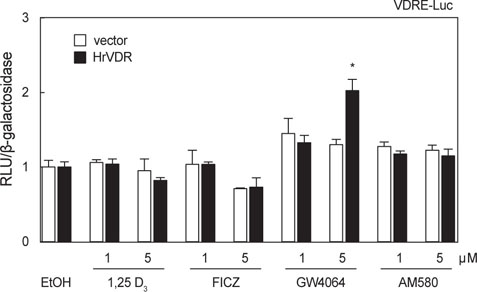
VDRE-luc reporter를 이용한 luciferase assay에서는 positive control hVDR을 도입한 HEK293 세포에서만 활성형 비타민 D3 (1,25 D3)에 의해 전사활성이 보였고, 다른 비타민D3 전구체 7DHC, CHOLC, 25(OH)D3 및 식물성 비타민 D2에 의한 활성은 보이지 않았고, HrVDR은 실험에 이용된 모든 리간드에 반응을 나타내지 않았다(Fig. 3B). 척추동물의 VDR은 RXR과 heterodimer를 형성하여 최대한의 전사활성을 나타내며 공활성인자가 관여하기 때문에(Sánchez-Martínez et al., 2006), HrRXR, hRXR 및 hSRC1을 transfection하고 전사활성을 추가적으로 조사하였다. HrVDR/HrRXR, HrVDR/hRXR transfection 세포에 1,25 D3 및 9-cis RA 처리에 의해 변화가 없었지만, 공활성인자 hSRC1의 cotransfection은 전사활성을 유의적으로 증가시켰다(Fig. 3C;
본 연구에서는 멍게 VDR을 암호화하는 full-length cDNA (
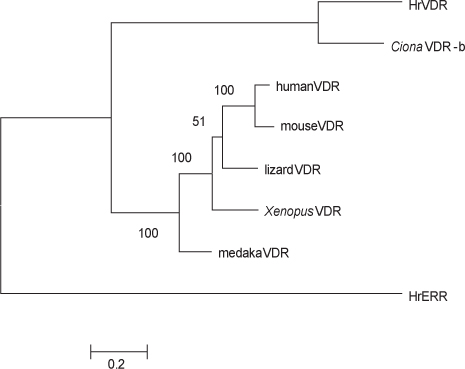
VDR 유전자는 인간, 마우스, 닭, 도마뱀,
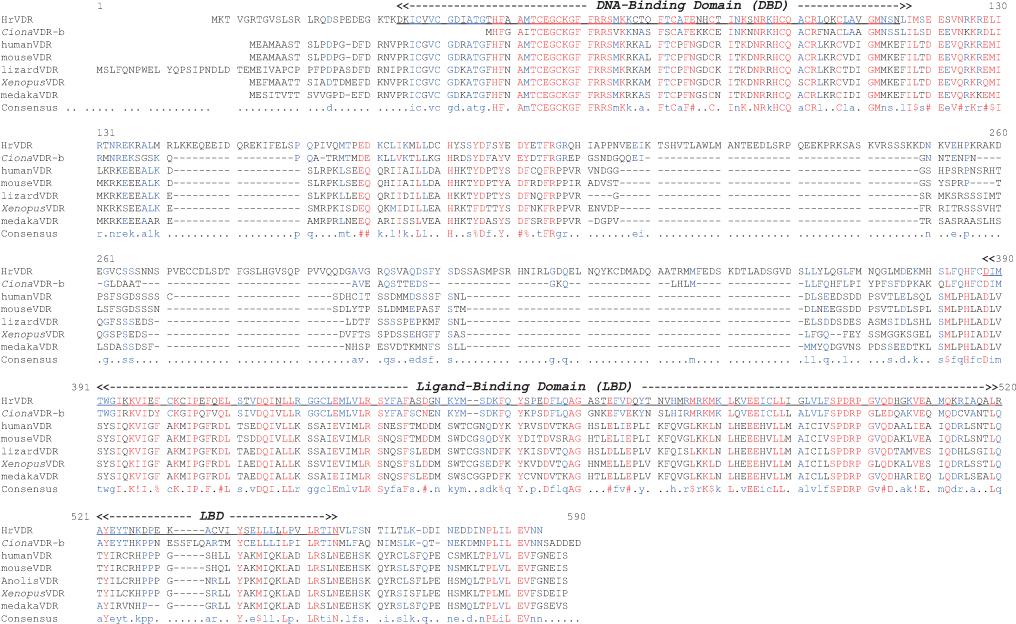
다른 척추동물의 VDR 단백질의 핵심적인 기능 domain들로 판단되고 있는 DBD 및 LBD 영역에 대하여 HrVDR의 대응 영역과 비교하였을 때, 다소간 아미노산서열의 변이가 높은 수준임을 알 수 있었다. 비록 NCBI Blast 분석 결과 척추동물의 가장 유사한 분자로서 VDR 단백질들이 검색되었지만, 호르몬 결합 부위인 LBD의 변이가 많았고 유사 종
척추동물의 VDR과 현저한 아미노산 변이를 보이는 HrVDR 및