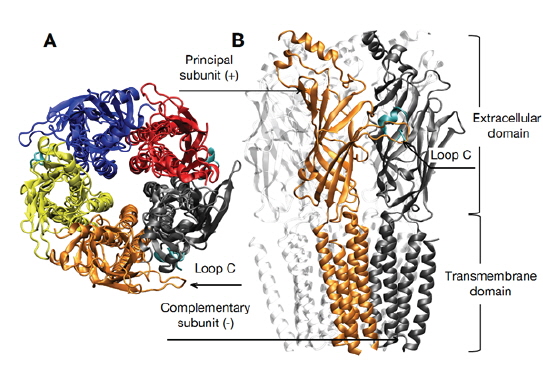
Nicotinic acetylcholine receptor (nAChR)는 신경 전달 물질인 acetylcholine이 extracellular (EC) domain에 결합되어 활성화 되며, 이어서 transmembrane (TM) domain에 의해 형성된 채널이 열리면서 이온을 통과시켜 presynaptic 말단으로부터 온 신호를 postsynaptic 말단으로 전달하게 된다(Fig. 1). 따라서 금연 및 통증 치료, 알츠하이머, 파킨슨 질병 등과 같은 신경전달 및 인지현상 등과 관련된 많은 질병들의 표적이 되어왔고 또한 신약 개발의 잠재적인 표적으로 간주된다(Albuquerque et al., 2009). 다섯 개의 subunit으로 구성된 nAChR는 다양한 alpha (α2-α10)와 beta (β2-β4) subunit의 조합으로 다양한 homo- 혹은 heteropentameric 수용체의 subtype을 형성한다.
해양 무척추 동물인 다양한 청자고둥 종류에서 분리된 독소인 conotoxin들은 다양한 수용체와 이온채널 등에 특이성을 가지고 있어 발견 초기부터 제약 산업계로부터 관심을 많이 받았고, 그 중 하나는 만성 통증 완화제로 개발되어 승인을 받고 판매 중이며, 일부는 알츠하이머, 파킨슨 질병 등의 치료제로 임상 단계에 들어갔다(Clark et al., 2012). 특히 α-conotoxin은 포유류의 nAChR를 표적으로 하며(Azam and McIntosh, 2009),
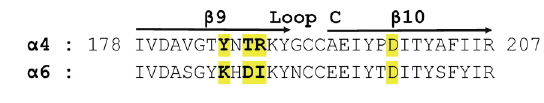
최근 한 연구에서 α-conotoxin BuIA에 대한 α6 subuint의 특이성을 결정하는 주요 아미노산 잔기를 돌연변이 유도와 전기 생리학적 실험을 통해 밝혔다(Kim and McIntosh, 2012). 실험을 통해 연구자들은 α-conotoxin이 α6β2 수용체에 대해 α4β2 수용체에 비해 60,000 배의 선택성을 가지며, α6 subunit의 세 아미노산 잔기 Lys185, Asp187 및 Ile188가 결정적인 역할을 하는 것을 밝혔다. 또한 구조 모델링을 통해 독소와 이들 아미노산이 10 Å 이상 떨어져 있어서 직접적으로 결합력에 영향을 주는 것이 아닌 것으로 예측 하였지만, 어떻게 이들 세 아미노산이 α-conotoxin에 대한 선택성에 영향을 주는지 그 기작을 설명하지는 못하였다.
해양에서 많은 천연물들이 분리되고 있고 이들의 생리활성에 관한 연구가 진행됨에 따라 천연물의 기작에 대한 연구가 필요하고 이를 물리화학적으로 분석하는 연구가 필요하다. 또한 많은 경우 실험적 분석만으로 모든 것을 설명하지는 못한다. 본 연구는 분자 모델링과 더 나아가 분자동역학 전산모사(all-atom molecular dynamics simulation)를 통해 실험적으로 설명할 수 없었던 α-conotoxin의 α6 subunit에 대한 특이성을 결정하는 기작을 원자적 수준에서 이해하고, 실험적 분석을 보완하고자 수행되었다.
본 연구에서는 시궁쥐(
보다 정확한 분자 모델링과 mutation으로 인한 구조 변화를 연구하기 위해 homology 모델링으로부터 얻어진 수용체의 구조를 분자동역학 전산모사를 이용하여 더욱 이완시켰다. 전체 전산모사 시스템의 빠른 equilibration을 위해 모델링으로 준비된 수용체의 TM domain을 pre-equilibrated 1,2-dioleoyl-snglycero-3-phosphatidylcholine (DOPC) lipid bilayer (지질 이중막)에 삽입시켰다. 이 과정에서 중첩되는 물과 DOPC 지질 분자들을 제거하여 수용체가 위치할 공간을 준비하여 최종적으로 368개의 DOPC 지질 분자가 이중막을 형성하게 되었다. 각각 총 44,679개와 44,671개의 TIP3P 물 분자 모델을 사용하여 WT과 MT 수용체 시스템을 solvation하였으며, NaCl 이온으로 전산모사 시스템을 전기적 중성상태로 만들고, 최종적으로 이온 농도를 150 mM로 만들었다.
시스템이 준비된 후 먼저 complex conjugated energy minimization을 1,000단계를 수행하여 solvation으로 인한 bad contact 등을 제거함으로 전산모사를 시작하였다. 이후 60 picosecond (ps) 동안 점진적으로 온도를 4 K에서 298.15 K까지 등적(constant volume) 조건으로 올린 후, 마지막으로 등온 등압(constant temperature and pressure) 조건으로 전환하여 두 시스템 각각 215 nanosecond (ns) 동안 전산모사를 계속 수행하였다. 시스템의 온도는 평균 298.15 K, 압력은 평균 1 bar로 유지되었고, 수소원자와 연결된 모든 공유결합은 고정시켜 Newton 운동방정식의 적분 간격을 2 femtosecond로 하였다. 주기적 경계 조건을(periodic boundary condition) 사용하여 원거리 정전기력은 Particle Mesh Ewald (PME) 방법으로 다루었으며(Toukmaji et al., 2000), 근거리 정전기력과 판데르발스 상호작용은 12 Å cutoff를 사용하였다. 전산모사를 통한 모든 원자들의 좌표는 10 ps 마다 trajectory로 저장 되어 결과 해석에 사용되었으며, 20 ns와 30 ns 이후 각각 WT과 MT 두 시스템의 에너지와 구조가 안정되었고(equilibration stage), 그 이후부터 215 ns까지 trajectory를 WT과 MT 수용체의 비교하는데 사용하였다 (production stage). 전산모사를 위한 시스템의 준비와 전산모사 이후 trajectory의 해석 및 visualization 등은 Visual Molecular Dynamics (VMD) 프로그램을 사용하였고 (Humphrey et al., 1996), 모든 전사모사는 NAMD2.9 (Phillips et al., 2005) 프로그램을 이용하여 수행되었으며 force field는 CHARMM27을 사용하였다(Mackerell et al., 2004).
우리는 먼저 mutation 이후 MT 수용체의 세 loop C 주위의 아미노산 잔기들의 구조에 유연성의 변화가 오는 것을 발견하였다. WT 수용체가 상대적으로 보다 안정된 2차 구조를 (secondary structure, β9와 β10) 보이는 반면, MT 수용체는 loop C 주위의 잔기들은(잔기 185부터 200까지) 2차 구조를 잃고 유연성이 상대적으로 증가하는 것을 관측하였다(Fig. 3). 아미노산 잔기 180에서 200 사이 알파 탄소(Cα)의 root-mean-square fluctuation (RMSF)을 계산하여 WT과 MT 수용체의 loop C 주위의 유연성 변화를 정량적으로 측정하였다(Fig. 4). 특히 세 α subunit에 대한 loop C (Cys192 and Cys193)의 평균 RMSF가 각각 2.24 Å과 3.73 Å으로 약 1.5 Å 정도 MT의 경우 유연성이 더욱 크다는 것을 확인할 수 있었다. 추가적으로 우리는 α subunit의 loop C 주위의 β9와 β10가 WT의 경우 서로 가깝고 안정되게 유지되는 반면, MT의 경우 두 β strands 사이의 거리가 증가하는 것을 확인하였다(Table 1, Fig. 3).

The distances between two selections of Cα atoms averaged over the subunits and over the simulation time. In the selection of principal and complementary subunits, the transmembrane (TM) domain and loop C region (185-200) were not included
두 β strands 사이에는 backbone 수소결합이 형성되어 그 구조를 안정화 시키는데, 앞선 계산에서 mutation 이후 loop C 주위의 RMSF와 두 β strands 사이의 거리가 증가하는 것이 그들 사이에서의 수소결합이 끊어졌기 때문인 것으로 확인되었다(Fig. 3). 세 아미노산 잔기의 mutation 이후 loop C 주위의 β9과 β10 사이의 backbone 수소결합의 개수가 2.00에서 0.76으로 감소하였고(Table 2), 추가적으로 이들 두 β strand의 side chain 사이의 수소결합의 변화 또한 확인하였다. Fig. 3에 나타난 것과 같이 WT에서의 양전하를 띤 아미노산 Arg188과 음전하를 띤 아미노산 Asp199 사이의 수소결합인 salt bridge가 mutation (Arg188Ile)에 의해 α6 subuint의 전하를 띠지도 않고 소수성(hydrophobic) 아미노산 잔기인 isoleucine으로 대체되면서 MT에서는 더 이상 형성되지 못하는 것을 알 수 있었고, MT에서 Lys185와 Asp187 사이에 새로운 salt bridge가 형성된 것 또한 확인하였다(Table 2).

Hydrogen bond calculations of loop C region averaged over three α subunits and over simulation time. Hydrogen bond was defined by 3.4 A (distance between donor and acceptor atoms) and 60 degrees (angle formed by donor, attached hydrogen and acceptor atoms)
>
독소와의 상호작용(interactions between α -conotoxin and binding site)
본 연구의 목적인 α6 subunit의 α-conotoxin에 대한 특이성을 이해하기 위해 mutation 이후 독소와 결합부위(binding site)와의 상호작용의 변화를 살펴보았다. 결합부위는 loop C, α subunit인 principal subunit, 그리고 complementary subunit 세 부분으로 구성되어 있어, 이들과 α-conotoxin과의 거리를 측정하였다. Table 1에서와 같이 독소는 loop C와는 조금 멀어지고, 두 subunit과는 상당히 가까워 진 것을 확인하였다. 이는 α-conotoxin과 이들과의 상호작용이 변한 것으로 해석되어 그들 사이의 number of contact를 측정하여 재확인하였다. Table 3에 나타낸 것과 같이 독소와 loop C 주위와의 상호작용은 줄어든 것에 반해, principal subunit and complementary subunit과는 상호작용이 증가한 것을 알 수 있었다.

Number of contacts with 3 A distance criterion between α-conotoxin BuIA and given selections averaged over the subunits and over the simulation time
우리는 α-conotoxin BuIA의 α6 subunit에 대한 특이성을 분자 수준에서 이해하기 위해 간단한 모델로서 α4 subunit의 세 아미노산 잔기를 α6 subunit의 세 아미노산 잔기로 mutation시킨 후 그 구조 변화를 분자동역학 전산모사를 통해 살펴보았다. Mutation 이후 loop C 주위의 두 β9과 β10 strand의 유연성이 증가하고, 두 β strands 사이의 거리가 증가되었으며, backbone 수소결합이 감소하고, side chain의 salt bridge가 사라진것을 확인하였다. 또한 α-conotoxin은 loop C와는 거리가 증가하면서 상호작용이 감소하였지만, 결합부위의 다른 두 부분을 구성하는 principal subunit과 complementary subunit과의 거리는 감소하고 상호작용은 증가하는 것을 관측하였다. 이 모든 변화의 원인은 세 아미노산 잔기의 mutation (Tyr185Lys, Thr187Asp, Arg188Ile)으로 설명이 되어야 한다.
본 연구 결과에 따라 이러한 변화의 원인이 WT에서의 β9의 Arg188과 β10의 Asp199 사이의 salt bridge가 mutation (Arg188Ile)에 의해 더 이상 형성되지 못하기 때문인 것으로 예측된다(Fig. 3). 서로 반대의 전하를 띄는 두 아미노산 잔기들의 side chain 사이의 수소결합인 salt bridge는 일반 중성 분자들의 side chain 사이의 수소결합 혹은 backbone 원자들 사이의 수소결합보다 훨씬 더 강하여, 두 β strand인 β9과 β10를 사이 거리를 안정되게 유지하지만, mutation 이후 salt bridge를 잃게 되어 거리는 증가하게 된다. 이로 인해 β9과 β10를 사이의 backbone 원자들 사이의 수소결합도 잃게 되어 loop C 주위는 유연성이 커지게 된다. Mutation 이후 새롭게 형성된 Lys185와 Asp187 사이의 salt bridge는 모두 β9에 있어 두 strand의 거리 유지에는 도움이 되지 못한다. Loop C 주위의 극성을 띤 backbone 원자들은 수소결합을 잃은 후 주위 환경에 노출되어 소수성 상호작용(hydrophobic interaction)이 주가 되는 α-conotoxin과의 상호작용이 약해져서 그 거리가 증가되고(Ulens et al., 2006), 상대적으로 principal subunit과 complementary subunit으로 구성된 결합부위(binding site)와의 상호작용은 증가하여 거리가 감소하게 된다. 이로 인해 최종적으로 독소에 대한 특이성이 커지게 되는 것이다.
본 연구를 통해 우리는 실험적으로 밝혀진 α6 subunit의 세 아미노산이(Lys185, Asp187, Ile188) 어떻게 α-conotoxin BuIA에 대한 특이성을 결정하는지 분자모델링과 분자동역학 전산 모사를 통해 연구하였다. 본 연구를 통해 실험만으로는 이해할 수 없었던 세 아미노산이 독소에 대한 특이성을 나타내는 기작을 조사할 수 있었다. 특히 분자 모델링 만으로는 세 아미노산 잔기들이 독소의 결합부위와는 거리적으로 떨어져 있어 직접적 상호작용이 아닌 간접적으로 영향을 주는 것 밖에 예측할 수 없었다. 우리는 분자동역학 전산모사를 통해 α6의 독소에 대한 선택성의 근원이 정전기적 상호작용인 것으로 결론 낼 수 있었다. 최종적으로 α6의 독소 선택성에 대한 기작이 Fig. 5에 묘사되어있다. 반대 전하를 띠고 salt bridge를 형성하는 α4 subunit과 달리 α6 subunit의 loop C 주위 부분은 backbone 수소결합을 잃고 극성 원자들이 노출이 되어 상대적으로 상호작용이 약해지고 두 subunit이 형성하는 결합부위와는 더욱 상호작용이 증가하여 결합력이 커지는 것을 알 수 있었다. β9과 β10 사이의 salt bridge를 파괴하는 또 다른 방법은 Asp199을 mutation하는 것이다. 실험에서는 salt bridge의 다른 짝이 되는 Asp199는 mutation을 수행하지 않았는데 이는 α4와 α6 subunit이 동일한 아미노산을 가지고 있기 때문이었다(Kim and McIntosh, 2012). 여기서 우리는 본 연구를 통해 α4 subunit의 Asp199를 mutation하더라도 α-conotoxin에 대한 특이성을 얻을 수 있을 것이라는 하나의 가설을 제안할 수 있게 된다.