동물의 장관에 서식하고 있는 고유한 장내미생물의 대부분은 숙주에 유익한 영향을 미치거나 무해하다. 일반적으로 장내미생물은 장관으로 침입하는 병원체로부터 숙주를 보호하며, 섭취된 음식으로부터 영양성분과 에너지를 얻고 정상적인 면역 기능을 수행하는데 도움을 주는 것으로 알려져 있다(Sonnenburg et al., 2005; Candela et al., 2008; Fukuda et al., 2011; Olszak et al., 2012; Yatsunenko et al., 2012). 최근 장내미생물이 숙주의 체중 증가 또는 비만에 미치는 영향을 이해하기 위한 연구가 많이 진행되고 있다(Ley et al., 2006; Angelakis et al., 2012). 또한 차세대 염기서열 분석방법을 이용한 장내 미생물 생태계의 특징과 이해를 위한 연구가 매우 활발히 수행되고 있지만(Eckburg et al., 2005), 아직도 많은 부분이 이해되지 않고 있다. 사실 어류에서도 장내미생물의 중요성이 점점 인식되고 있지만, 사람이나 다른 포유동물에 비해서는 그와 관련된 연구가 매우 드문 편이다(Gatesoupe, 2010). 다른 동물과 마찬가지로 장내 마이크로바이옴이 어류에 미치는 영향을 이해하기 위해서는 어떤 종류의 고유한 미생물이 어류의 장관 내에 서식하는지를 이해하는 것이 우선시 되어야 할 것이다. 그럼에도 불구하고 우리나라 최대 양식어종인 넙치의 장내세균의 다양성에 관한 연구는 매우 제한적이다(Sugita et al., 2002; Sugita and Ito, 2006; Kim and Kim, 2013). 이번 연구의 목적은 배양되는 넙치 장내미생물 다양성 이해를 위한 최적의 배양조건을 탐색하고, 그 조건에 따른 미생물 다양성을 비교하며 16S rRNA gene clone library를 제작하여 비배양 의존적 방법을 이용한 미생물의 다양성을 이해하고자 하였다.
이번 연구에서는 배합사료만 급여한 양식 넙치(
Marine agar와 TNY (1.5%의 NaCl과 0.3%의 yeast extract가 첨가된 TSA)에서 분리된 세균들을 25℃에서 24시간 동안 배양한 이후 3,000
>
Polymerase chain reaction (PCR)
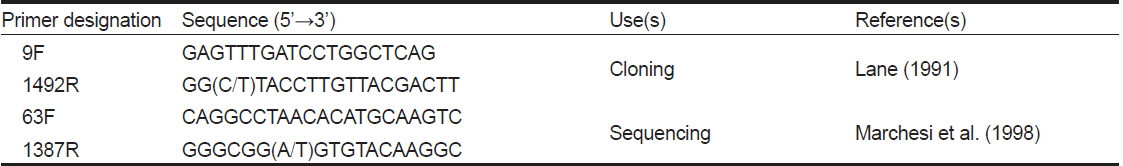
각 분리균주와 장점액으로부터 추출한 DNA를 Table 1에 제시된 프라이머를 이용하여 증폭시켰다. PCR의 조건은 다음과 같이 수행하였다; 94℃에서 3분 동안 initial denaturation을 수행하고, 94℃에서 30초, 56℃에서 30초, 72℃에서 1분의 과정을 30회 반복한 후, 72℃에서 7분간 final extension하였다. 증폭 산물은 ethidium bromide (1 ng/mL)가 첨가된 1% (w/v) agarose gel 상에서 전기영동하여 분석하였다.
[Table 1.] Primers used for PCR and sequencing in this study
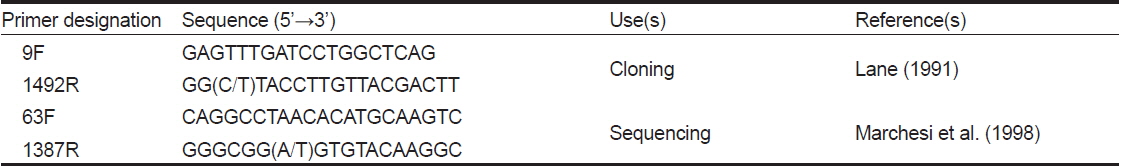
Primers used for PCR and sequencing in this study
장점액으로부터 추출한 DNA를 9F와 1492R primer (Table 1)를 사용하여 PCR로 증폭시켰다. 정제된 PCR product를 pGEM®-T vector (Promega)와
배양된 세균과 insertion을 가진 plasmid DNA의 정제된 PCR product의 sequencing reaction은 이전에 서술되었던 것과 같이 Big Dye Terminator cycle sequencing kit와 ABI Prism 310 Genetic Analyzer (Applied Biosystems, Foster City, CA, USA)를 사용하여 수행하였다(Han et al., 2011). DNA sequence의 quality 확인을 위하여 Bioedit (version 7.0.9.0)과 Genetyx version 7.0.3 packed program (Software Development Co., Tokyo, Japan)을 사용하였다. 또한, 16S ribosomal DNA clone library의 염기서열은 online tool인 DECIPHER(Wright et al., 2012)를 사용하여 chimeric construct의 유무를 확인하였고, 3′과 5′ 말단에 있는 부정확한 염기서열 또한 제거하였다. 1% 이하의 염기서열 상동성을 가진 경우 동일한 phylotype (operational taxonomic units)으로 간주하여 분석을 진행하였고, 각 phylotype과 가장 근접한 표준균주를 결정하기 위해 RDP SEQMATCH program (http://rdp.cme.msu. edu/index.jsp) (Cole et al., 2013)을 이용하였다. 계통수 제작에는 ClustalX (version 1.83)와 MEGA6(Saitou and Nei, 1987; Tamura et al., 2011) 프로그램이 이용되었으며, Kimura-2-parameter distances에 기초하여 neighbor-joining tree를 만들었으며 1,000번의 re-sampling을 통해 bootstrap value를 결정하였다.
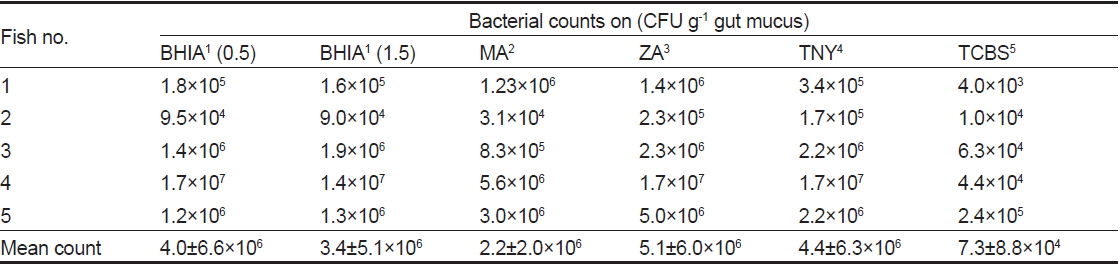
넙치 장점액 분리 후 단계 희석하여 BHIA (0.5), BHIA (1.5), TNY, MA, ZA, TCBS에 배양한 결과를 Table 2에 나타내었다. TCBS를 제외한 모든 배지에서 104-107 CFU/g gut mucus의 세균이 배양되었으며, TCBS에서는 7.3±8.8×104 CFU/g gut mucus으로 나타나 배양되는 세균 중에서 Vibrio 과에 속하는 세균이 많이 존재하고 있음을 확인 할 수 있었다(Table 2). 이 결과는 이전 넙치의 연구 결과(Kim and Kim, 2013)와 매우 유사하였다. Senegales sole
[Table 2.] Microbial colony counts of gut microbes grown on varying culture media
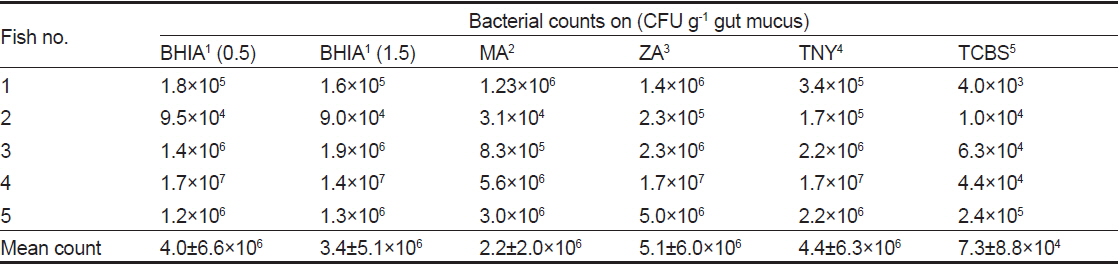
Microbial colony counts of gut microbes grown on varying culture media
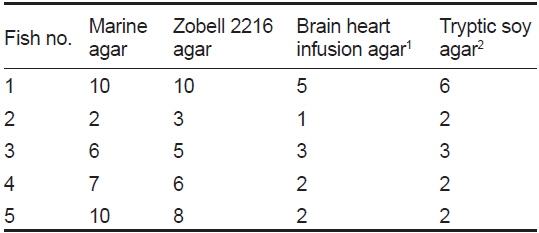
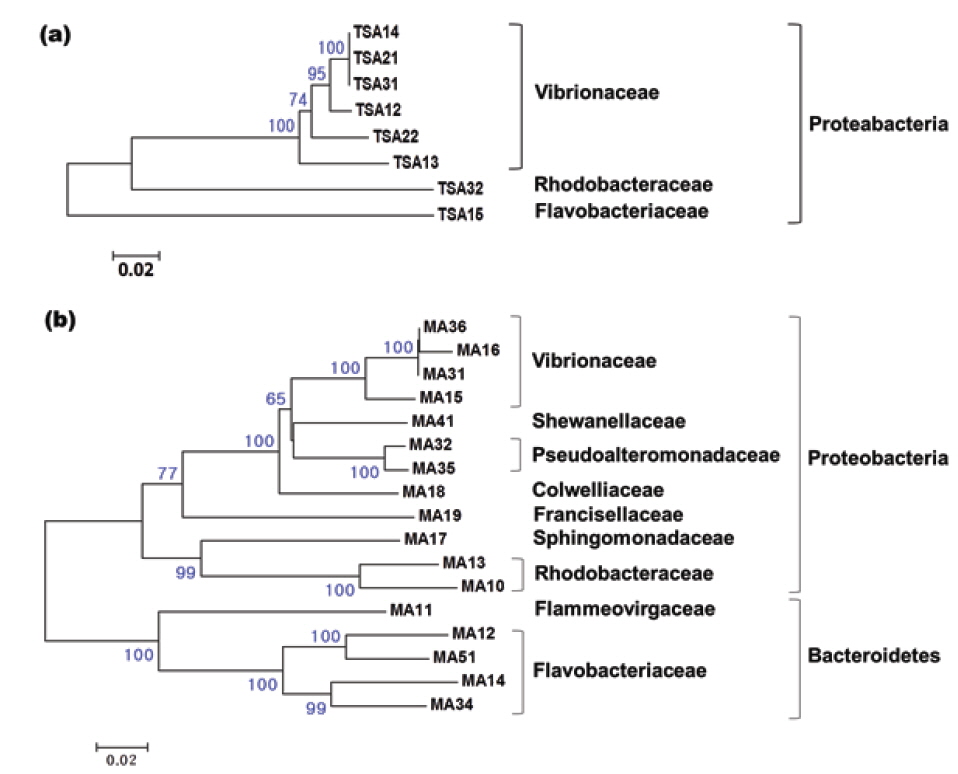
넙치의 장내세균의 다양성에 관한 이전 연구가 존재하지만 매우 드물다(Sugita and Ito, 2006; Kim and Kim, 2013). 이번 연구에서는 6종류의 배지를 이용하여 넙치 장내세균의 다양성 연구에 최적인 배지를 탐색하고자 하였으며, 그 중 4종류의 배지에서 관찰된 결과를 Table 3에 나타내었다. 분별된 세균 집락의 종류는 Marine agar와 Zobell 2216 agar에서 가장 많았으며, 개체에 따라 서로 1 또는 2개 정도의 차이 밖에 나지않아 그 다양성은 유사하였다. BHIA와 TNY배지에서는 MA와 ZA와 달리 다양한 세균 집락이 배양되지 않았다. 유전적으로 99% 이상의 상동성을 가지는 경우 동일한 phylotype으로 간주하였을 때, TNY와 MA에서 각각 8과 17개의 phylotype이 나타났으며 그 결과를 Fig. 1에 계통수로 나타내었다. TNY 배지에서 분리된 8개의 phylotype은 모두 Proteobacteria 그룹에 포함되었으며, 이 중 6개가 Vibrionaceae, 나머지는 Rhodobacteraceae와 Flavobacteriaceae에 각각 속하였다(Fig. 1a). 이에 반해 MA 배지에서는 TNY에서보다 더 다양한 세균이 분리되었다(Fig. 1b). 17개의 분리된 세균 중 Proteobacteria 그룹에 속하는 phylotype은 12개로 우점적이었으며, Vibrionaceae와 Pseudoalteromonadaceae에 각각 4개와 2개의 phylotype이 포함되었고, Shewanellaceae, Colwelliaceae, Francisellaceae, Sphingomonadaceae, Rhodobacteraceae에 각각 하나의 phylotype이 속하였다. 나머지 5개의 phylotype은 TNY에서 배양된 세균에서는 관찰되지 않은 Bacteroidetes 그룹에 속하였으며, 이 중 4개와 1개의 phylotype이 각각 Flavobacteriaceae와 Flammeovirgaceae에 포함되었다. 일반적으로 TNY와 BHIA, nutrient agar는 기본 영양 배지로 세균 배양 시 많이 사용되지만, 배양되는 장내세균의 다양성 연구에는 Marine agar 또는 Zobell 2216 agar가 가장 적합한 것을 확인하였다.
[Table 3.] Differential colony counts appeared on varying media
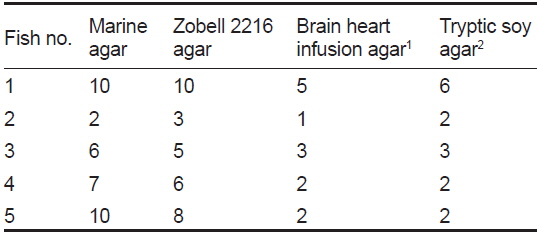
Differential colony counts appeared on varying media
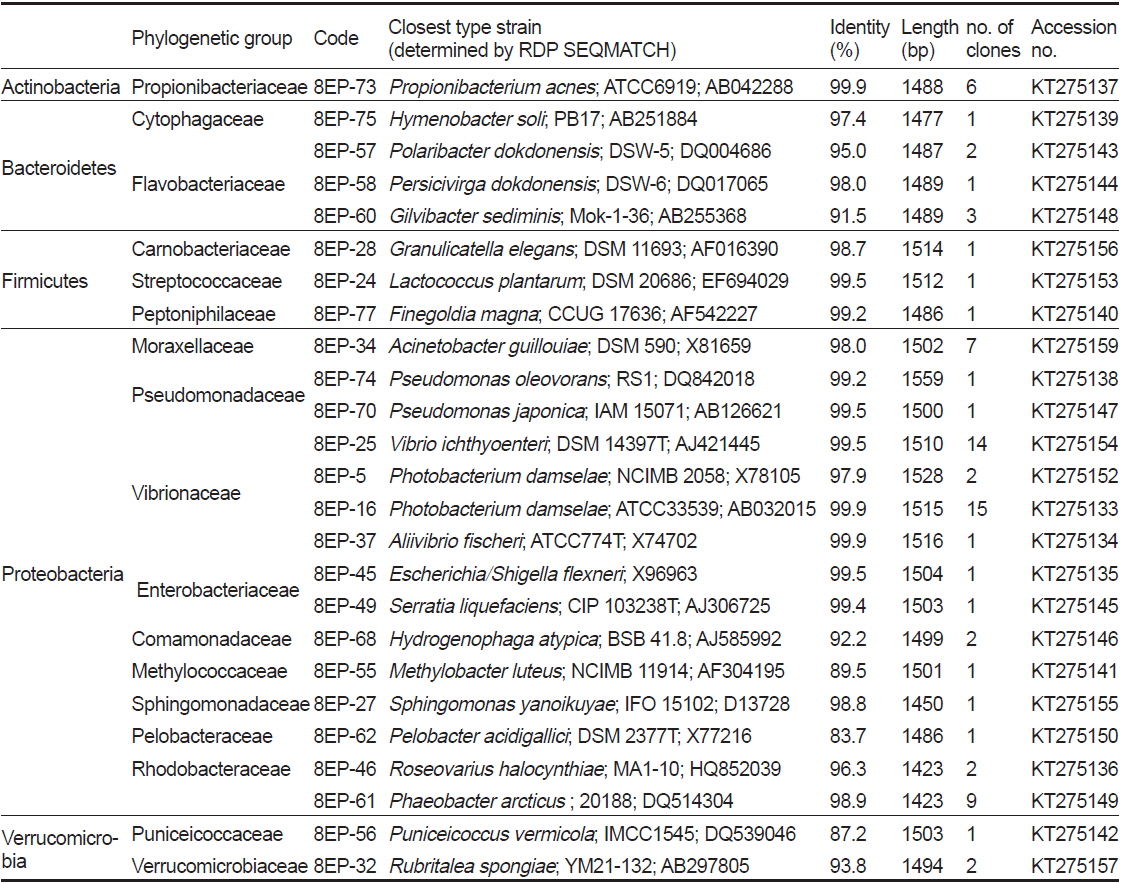
16S rRNA gene clone library의 염기서열 분석으로 확인된 세균 종
이번 연구에서는 총 78개 clone의 염기서열을 분석하여 25개의 phylotype을 얻었으며 가장 근접한 세균 종(기존의 염기서열이 밝혀진 표준균주)의 결과를 Table 4에 나타내었다. 결론적으로 clone library에서 도출된 많은 염기서열은 이전에 어류의 장내세균으로는 알려진 적이 없는 새로운 세균 종의 서열임이 드러났다. 이 25개의 phylotype은 Actinobacteria, Bacteroidetes, Firmicutes, Proteobacteria 및 Verrucomicrobia에 속하였으며, phylotype의 대부분(약 76%)이 Proteobacteria로 가장 많은 비율을 차지하였다(Fig. 3). Proteobacteria 그룹 중에서는 Vibrionaceae가 가장 우점적(약 55%)이었으며, 다음으로 Rhodobacteraceae, Moraxellaceae가 각각 약 19%와 12%의 비율을 차지하였다(Fig. 3). 이외에도 Enterobacteriaceae, Pseudomonadaceae, Moraxellaceae, Methylococcaceae, Comamonadaceae, Sphingomonadaceae에 속하는 세균들이 분리되었다(Fig. 2 and Table 4). 양식산과 자연산 넙치 장관의 세균 다양성을 조사한 연구에서 각각 38개와 47개의 phylotype이 분리되었고 Firmicutes, Proteobacteria, Actinobacteria와 Bacteroidetes 그룹에 속하였다(Kim and Kim, 2013). 이 중 Proteobacteria가 양식산 어류에서 60.5%, 자연산 어류에서는 51.1%로 가장 우점적이었으며, 이번 연구에서처럼 Vibrionaceae가 가장 높은 비율을 차지하였다(Kim and Kim, 2013).
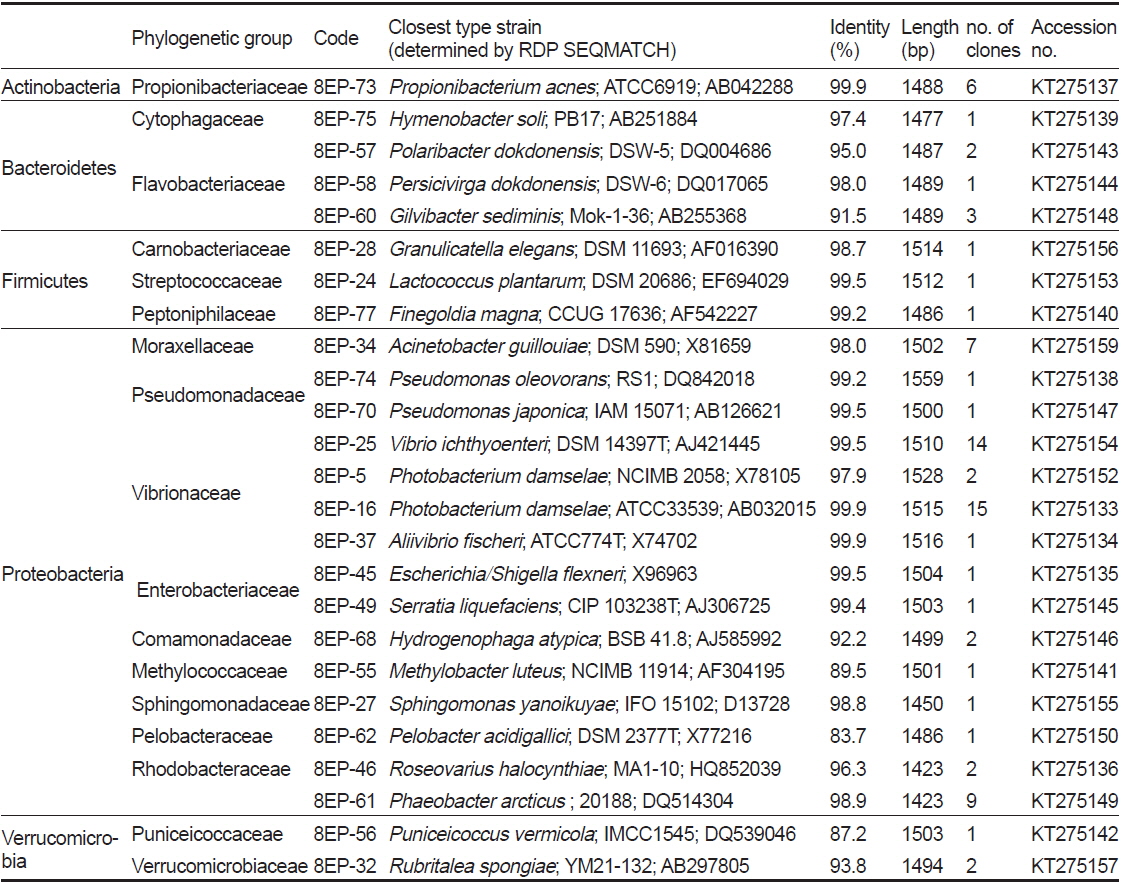
Representative sequences of a clone library isolated from the intestinal mucus of farmed olive flounder Paralichthys olivaceus
이번 연구에서는
Actinobacteria에 속하는 6개의 clone은 모두 Propionibacteriaceae의
이상의 결과를 종합해 본 결과 넙치의 배양되는 세균의 다양 성 연구에는 Marine agar 또는 Zobell 2216 agar가 가장 적합한 것으로 나타났다. 그러나 배양 의존적인 방법은 특별한 배양 환경 및 조건이 요구되어 배양이 까다로운 미생물이거나 절대 혐기성 미생물의 배양이 배제되기 때문에 한계가 있다(Asfie et al., 2003). 이번 연구에서는 5마리의 넙치를 배양되는 세균과 또 다른 5마리의 넙치를 clone library 제작에 사용하여 배양 의존적인 방법과 비배양 의존적인 방법의 결과를 직접적으로 비교하는 것은 의미가 없다. 그렇지만 clone library에서 더 많은 phylotype이 도출되었고 이들 중 많은 염기서열이 이전 연구결과에서 드러나지 않았던 새로운 종임이 밝혀졌으므로 최신의 배양 비의존적 방법은 미생물의 다양성 연구에 매우 유용한 것임을 다시 한 번 확인 할 수 있었다. 최근 들어 차세대 염기서열 분석방법(next-generation sequencing)을 적용한 다양한 연구가 진행되고 있어 이 기술을 적용한 어류의 장내세균 다양성 및 기능성 연구가 필요할 것으로 생각된다.