Genetically modified(GM) trigonal cactus(Hylocereus trigonus Saff.) contained a coat protein gene of cactus virus X (CVX), which conferred resistance to the virus, phosphinothricin acetyltransferase (bar) gene, which conferred herbicide resistance, and a cauliflower mosaic virus 35S promoter (CaMV 35S). This study was conducted to evaluate the possible impact of GM trigonal cactus cultivation on the soil microbial community.
Microorganisms were isolated from the rhizosphere of GM and non-GM trigonal cactus cultivation soils. The total numbers of bacteria, and actinomycete in the rhizosphere soils cultivated GM and non-GM trigonal cactus were similar to each other, and there was no significant difference. Dominant bacterial phyla in the rhizosphere soils cultivated with GM and non-GM trigonal cactus were Proteobacteria, Uncultured archaeon, and Uncultured bacterium. The denaturing gradient gel electrophoresis (DGGE) profiles show a similar patterns, significant difference was not observed in each other. DNA was isolated from soil cultivated GM and non-GM trigonal cactus, we analyzed the persistence of the inserted gene by PCR. Amplification of the inserted genes was not observed in the soil DNA, which was collected after harvest.
This result suggests that the GM trigonal cactus cultivation does not change significantly the microbial community.
유전자변형(Genetically modified) 대두가 상업적으로 재배된 1996년 이후 제초제내성, 해충저항성 등 유전자변형 작물들의 재배면적은 해마다 증가하고 있다. 2012년 전 세계 유전자변형 작물의 재배면적은 1억 7,000만 ha(James, 2012)로, 대한민국 국토면적(1,002만 ha)의 약 17배에 해당된다. 유전자변형 작물은 작물 생산에 필요한 노동력과 농기계 사용 절감에 따른 경제적인 이익뿐만 아니라(Owen, 2000), 기후 변화 등의 농업환경 변화에 지속가능한 식량 생산을 가능케 한다(Brookes and Barfoot, 2006). 그러나 외래 유전자 도입에 의한 독성 및 알레르기 물질 생산 가능성(Konig
접목선인장은 국내 고유의 신품종과 재배기술로 해외시장에서 선호도가 높아 세계시장의 70~80%를 점유하는 고부가가치 작목이다. 접목선인장은 관상가치를 높이고 빠른 생육을 위해 대목인 삼각주(
선인장 바이러스 X(CVX)의 피막 단백질을 암호화하는 유전자가 삽입된 바이러스저항성 형질전환 삼각주와 선인장연구소로부터 분양받은 비형질전환 삼각주 모본을 590(W)×385(D)×150(H)크기의 사각포트에 원예용 상토를 채우고 12주씩 3반복으로 이식하여 재배하였다. 영양번식을 통해 증식하는 선인장의 특성에 맞춰 모체에서 3∼4개의 자구가 생겨날 때까지 일반재배법에 따라 재배하면서 시기별로 포트에 있는 상토를 채취하였다. 상토는 식물체를 뿌리째 뽑아 Kim 등(2008)의 방법에 따라 비근권 토양을 제거하여 군집 분석에 이용하였으며, 화학성 분석은 풍건 후 2 mm의 표준망체로 거른 시료를 이용하였다.
삼각플라스크에 채취한 토양 10 g과 멸균한 0.85% NaCl 90 mL를 넣어 진탕 배양기에서 30분간 200 rpm으로 현탁하였다. 일련의 희석과정을 거친 현탁액을 각각의 배지에 도말하고 28℃에서 배양하였다. 세균은 cycloheximide(0.05 g/L)를 첨가한 R2A agar(NA, Difco, MI) 배지에서 2일간, 진균은 chloramphenicol(0.02%)을 첨가한 R2A agar 배지에서 4일간, 방선균은 Sodium caseinate agar 배지에서 5일간 배양한 후 계수하였다. 미생물 수는 배양된 페트리디쉬에 나타난 균체를 3반복 계수하여 평균값을 생균수(colony forming unit, CFU/g 건토)로 산출하였다.
형질전환 삼각주와 비형질전환 삼각주를 재배한 토양 미생물 군집 변화는 Denaturing gradient gel electrophoresis (DGGE)로 분석하였다. FastDNA Spin Kit(Qbiogen, USA)로 토양 내 DNA를 추출하고, 진정세균의 미생물상 변이 분석을 위해 16S rRNA의 V9 부위를 증폭하는 1070f(5'-ATGGCTGTCGTCAGCT-3')와 여분의 G+C clamp (CGCCCGCCGCGCCCCGCGCCCGGCCCGCCGCCCCCGCCCC)가 부가된 1392r(5'-ACGGGCGGTGTGTAC-3') primer를 이용하여 PCR 반응을 수행하였다. 반응액 조성은 5 μL 10 × PCR buffer, 10 ng 주형 DNA, 25 pmol 양방향 프라이머, 200 μM dNTP, 2.5 U f-Taq DNA polymerase(Solgent, Korea)를 첨가하여 최종 부피는 50 μL로 하였다. PCR 조건은 95℃에서 5분간 변성 후, 95℃ 1 분, 55℃ 1분, 72℃ 1분의 과정을 30번 반복하였으며, 마지막으로 72℃에서 7분간 반응시켰다. PCR 산물은 Dcode Universal Mutation Detection System(Bio-Rad, USA)을 사용하여 변성제인 formamide가 40-70%로 농도 구배된 8% acrylamide gel에서 전기영동하였다. 전개된 DNA를 SYBR Green I(Cambrex BioScience, USA)과 EtBr로 염색하여 UV trans-illuminator에서 관찰하였다.
토양에서 추출한 DNA로부터 세균의 16S rRNA 영역을 증폭하는 27mf(5'-AGAGTTTGATCMTGGCTCAG-3')와 1492r(5'-GGYTACCTTGTTACGACTT-3') primer로 PCR을 수행하고 염기서열을 분석하여 동정하였다. PCR 반응액의 조성과 조건은 미생물 군집 분석 방법과 동일하게 수행하였으며, 1% agarose gel에서 확인된 DNA를 pGEM-T easy vector(Promega, USA)에 삽입하고
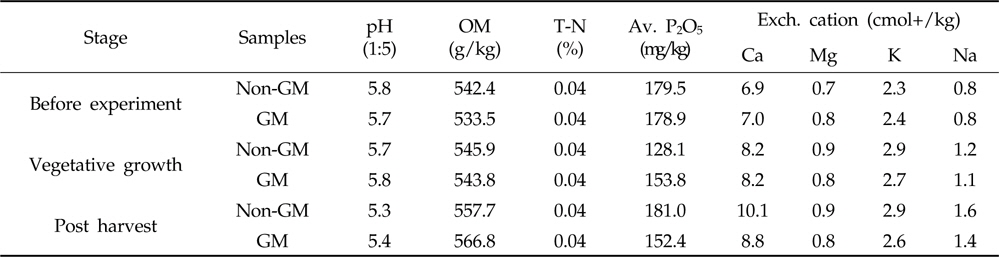
농촌진흥청의 토양 및 식물체 분석법(NIAST, 2000)에 준하여 형질전환 삼각주와 비형질전환 삼각주를 재배한 근권 토양을 음지에서 건조시켜 2 mm체를 통과한 것을 사용하였다. pH와 EC는 토양과 증류수를 1:5로 혼합하여 30분간 진탕한 후 현탁액을 측정하였으며, 유기물 함량은 Walkley와 Black법, 유효인산은 Bray No. 1법으로 각각 분석하였다. 칼륨, 칼슘, 마그네슘, 나트륨 등 치환성양이온은 1N Ammonium acetate(pH=7.0)로 침출한 후 유도결합플라즈마가 장착된 원자발광분광기(ICP-730-ES)로 분석하였다.
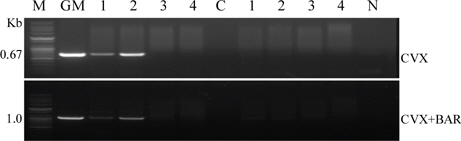
토양에서 추출된 DNA로부터 형질전환 삼각주에 도입된 CVX와 선발표지 유전자(
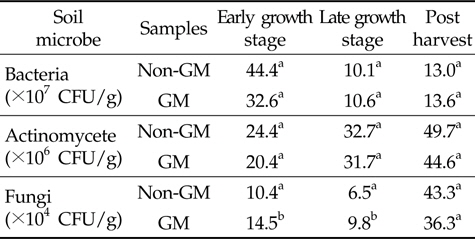
CVX 바이러스저항성 삼각주 재배에 의한 근권 토양의 미생물상 영향을 구명하고자 형질전환 삼각주와 비형질전환 삼각주 재배 토양의 생육시기별 세균, 방선균 및 진균 밀도를 조사하였다(Table 1). 형질전환 삼각주와 비형질전환 삼각주 재배 토양간의 생육시기별 세균, 방선균 밀도는 유의적인 차이가 없었다(P>0.05). 토양 내 진균 밀도는 생육기간 동안 형질전환 삼각주를 재배한 토양에서 비형질전환 삼각주에 비해 다소 높게 유지되었다(P<0.05). 그러나 수확이후의 형질전환 삼각주와 비형질전환 삼각주 토양에서는 유의성이 없어 (P>0.05) 안정적으로 유지되는 것으로 나타났다. 식물 뿌리에서 배출되는 삼출물은 미생물 군집과 활성의 변화를 초래할 수 있고 토양 미생물상의 변화는 유기물의 분해와 순환에 영향을 주어 식물의 생육이나 병원균 생장 등에 직⋅간접으로 영향을 끼칠 수 있다(Miethling
[Table 1.] Number of microbes in GM and non-GM trigonal cactus cultivated soil
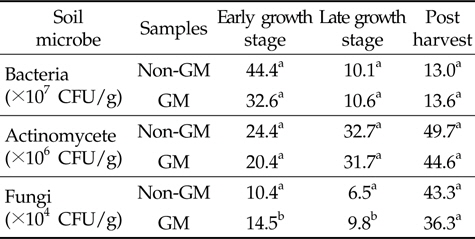
Number of microbes in GM and non-GM trigonal cactus cultivated soil
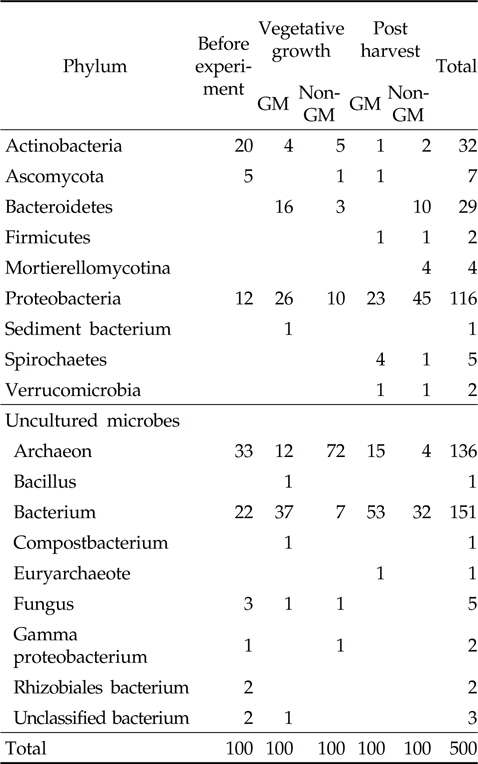
토양 미생물의 우점종 변화는 CVX 바이러스저항성 삼각주 재배 토양에서 DNA를 분리하고 미생물의 16S rRNA 염기서열을 분석하여 확인하였다. 근권 토양에서 동정된 세균들을 phylum 수준에서 비교한 결과(Table 2), 형질전환 삼각주와 비형질전환 삼각주의 근권 토양에는 Proteobacteria, Uncultured archaeon, Uncultured bacterium이 우점하였으나 시료 채취시기에 따라 우점세균은 증감하였다. 형질전환 삼각주 재배 토양의 우점세균은 시료 채취시기에 따라 변동이 크지 않았으나 비형질전환 삼각주 재배 토양은 영양생장기의 비배양 고세균의 편중과 비배양 세균의 감소 등으로 인해 변화가 크게 나타났다. 일반적으로 토양 미생물의 에너지 급원인 잔존물과 뿌리 삼출물 등의 증가로 식물체 생육에 따른 근권 토양 미생물의 변동이 나타나기도 하지만(Wei
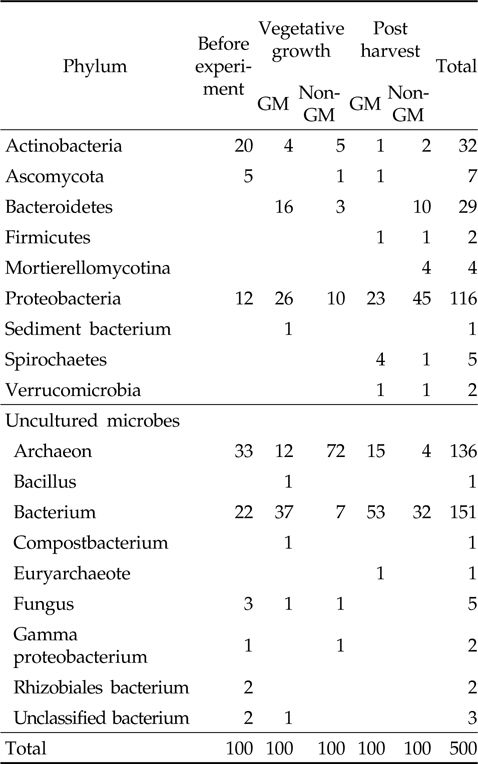
Phylogenetic distribution of isolates obtained from GM and non-GM trigonal cactus cultivated soil
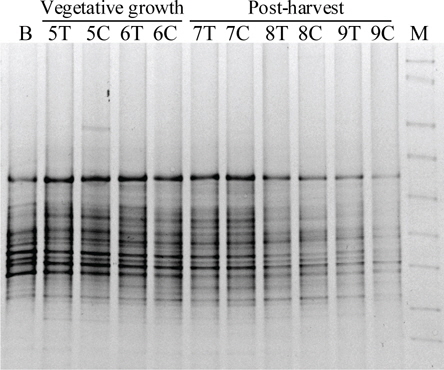
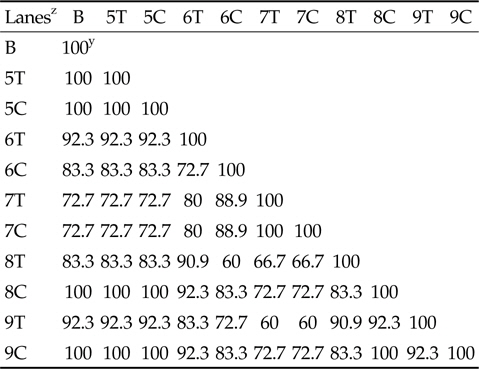
형질전환 삼각주와 비형질전환 삼각주를 재배한 토양으로부터 시기별 DNA를 추출하고 16S rRNA를 특이적으로 증폭하여 DGGE 분석법으로 근권 토양 미생물 군집을 비교하였다(Fig. 1). 형질전환 삼각주와 비형질전환 삼각주의 시기별 토양 DGGE profile은 모든 시기에서 거의 유사하였다. DGGE profile을 기반으로 밴드의 밀도와 수의 양상을 분석한 결과(Table 3), 삼각주 수확 이후 채취한 토양에서는 DNA의 분해 등으로 밴드의 증폭이 거의 일어나지 않았고 또한 삼각주 종류에 따른 차이도 거의 나타나지 않았다. 재배 시기에 따라 삼각주간의 밴드 유사성은 수확시 토양 내 뿌리 잔존물 증가로 7월 형질전환 삼각주 재배 토양의 밴드 유사성은 72.7%로 낮아졌으나 이후 92.3%로 회복되었다. 전체 밴드에 대한 분석 결과, 뿌리 잔존물이 많이 남아 있던 7월 토양과 수확이후 방치된 9월 토양과의 밴드 유사성은 60%로 가장 낮았으며 다른 밴드와의 유사성은 일정하게 유지되어 바이러스저항성 삼각주 재배에 따른 토양 미생물 군집의 변화는 생육기간에 일시적으로 나타나며 이후 수개월내에 회복되는 것으로 추정되었다. 파파야 Ringspot 바이러스저항성 형질전환 파파야를 재배한 토양의 DGGE 결과에서도 시료 채취 지역의 토양 이질성에 의해 생육시기별 밴드 패턴의 차이가 나타났다(Hsieh and Pan, 2006). DGGE 분석은 토양 미생물 군집변이에 대한 재배 품종, 시료 채취 시기 및 지역의 토양 이질성 등에 쉽게 영향을 받을 수 있기 때문에(Kim
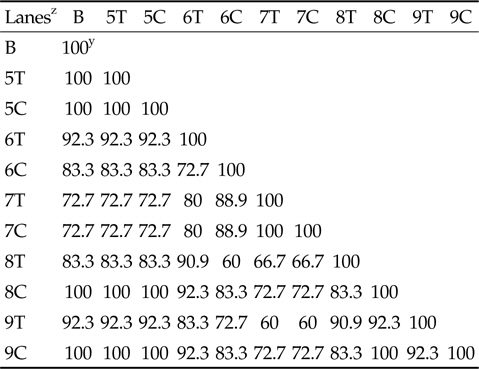
Comparison of similarity between GM and non-GM trigonal cactus based on UPGMA cluster analysis
토양 화학성은 토양 미생물의 변화에 따라 상호 반응하므로 토양 pH, 유효인산, 유기물 함량 등의 화학성 분석을 통해 형질전환 삼각주와 비형질전환 삼각주 재배에 의한 토양 미생물 변화를 비교하였다(Table 4). 토양 pH는 수확 이후 다소 낮아졌으나 생육 적정범위(pH 5.5-6.5)와 품질기준(pH 5.5-7.0)내에 존재하여(Lee
[Table 4.] Chemical characteristics of rhizosphere soil of GM and non-GM trigonal cactus
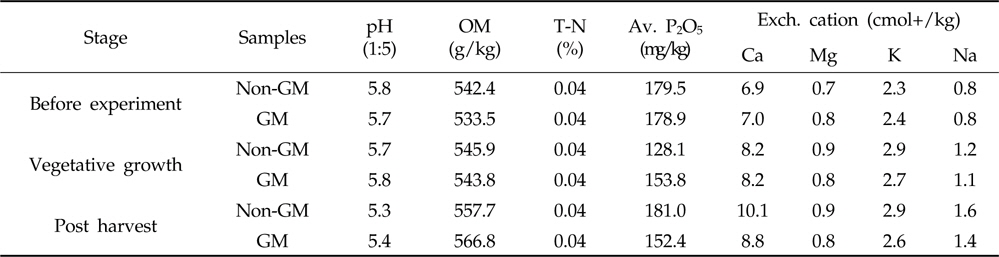
Chemical characteristics of rhizosphere soil of GM and non-GM trigonal cactus
수평적 유전자 이동성(Horizontal gene transfer)은 번식에 의하지 않고 개체로부터 다른 개체로 유전물질이 안정적으로 전달되는 것을 의미한다. 1963년 병원균이 복수의 항생제에 저항성을 나타내는 전달성 유전에서 처음 밝혀진 이 후 세균 진화의 주요한 요인으로 알려져 있다(OECD, 2010). 일반적으로 형질전환체 근권의 토양 미생물로 수평적 유전자 이동이 발생하기 위해서는 장기간 다량의 유전자가 근처에 존재해야만 한다. 도입 유전자의 잔존성은 형질전환 삼각주와 재배 토양으로부터 DNA를 분리하고 도입 유전자를 특이적으로 증폭하여 확인하였다(Fig. 2). 형질전환 삼각주 식물체에서는 도입 유전자와 선발표지로 사용된 제초제저항성 유전자가 증폭되었지만, 비형질전환 삼각주에서는 도입된 유전자가 증폭되지 않았다. 토양에서 추출한 DNA에서는 영양생장기에는 증폭된 밴드가 나타났지만 수확 이후 토양에서는 증폭된 밴드를 검출할 수 없었다. 이는 생육중기의 토양 DNA 추출시 식물체의 뿌리가 혼입된 것으로 추정되었으며, 수확 이후 토양 내 잔존성이 낮아 근권 토양 미생물로의 수평적 유전자 이동 가능성은 없을 것으로 예상되었다. 추후 삼각주 유전체 정보가 밝혀지면 내재유전자의 염기서열을 이용하여 잔존성에 대한 보다 명확한 기준제시와 분석이 가능할 것으로 예상되었다. Widmer 등(1997)은 형질전환 담배와 감자의 항생제 저항성 nptII 유전자가 각각 77일, 137일간 토양 내에 잔존할 수 있다고 보고하였으며 본 연구에서도 토양 내 도입 유전자는 2개월 정도 잔존하였으나 이후 분해되어 PCR 분석으로는 검출할 수 없었다. 식물 DNA의 토양 내 잔존성은 핵산 가수분해 효소에 의한 DNA의 분해(Smalla and Gebhard, 1999; Widmer