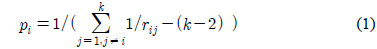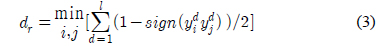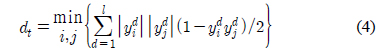One of the important hints for inferring the function of unknown proteins is the knowledge about protein subcellular localization. Recently, there are considerable researches on the prediction of subcellular localization of proteins which simultaneously exist at multiple subcellular localization. In this paper, label power-set classification is improved for the accurate prediction of multiple subcellular localization. The predicted multi-labels from the label power-set classifier are combined with their prediction probability to give the final result. To find the accurate probability estimates of multi-classes, this paper employs pair-wise comparison and error-correcting output codes frameworks. Prediction experiments on protein subcellular localization show significant performance improvement.
단백질은 대부분의 생명현상과 관련되어 있어서, 그 구조와 기능에 대한 연구가 활발하다. 단백질이 존재하는 세포내 위치 예측은 알려지지 않은 단백질의 기능에 대한 힌트를 얻기 위해 수행한다[1-9]. 이는 동물, 식물, 곰팡이와 같은 진핵생물은 세포 내부의 정교한 구획과 세포소기관이 존재하는데, 이러한 세포내 위치마다 서로 다른 생화학적 환경이 생기고, 이러한 환경에서 단백질은 위치 특이적인 기능을 수행하기 때문이다[10].
단백질의 세포내 위치 예측은 주로 하나의 단백질이 하나의 세포내 위치에만 나타나는 것을 대상으로 하였으나, 다중레이블 분류 기술의 발전에 따라서 다중위치에 대한 예측으로 확장되고 있다[1-9]. 다중레이블 분류는 하나의 입력 자료가 여러 분류에 속하는 문제를 처리하기 위해 연구되고 있다[11-13]. 다중레이블 분류가 단백질의 세포내 다중위치 예측에 적용된 예를 살펴보면, 최근접-이웃 분류기의 앙상블을 사용하는 방법[1, 7], 세포내 위치의 모든 쌍들에 대한 분류를 수행하고, 분류결과를 투표를 통하여 최종 결과를 얻는 방법[2], 가우시안 과정 모델과 공분산 행렬로 레이블간의 연관성을 표현하는 방법[3], 각 단일레이블에 관련된 사례들과 관련되지 않은 모든 사례들로 학습하고 분류를 위해서 투표를 하는 방법[4, 6], 다수의 이진 분류기를 체인으로 연결하고,
단백질의 세포내 위치예측 분야에서는 여러 다중레이블 분류 중에서 특정 생물학적 기능을 수행하는 단백질의 세포내 위치간의 관계를 효과적으로 모델링할 수 있는 분류체인 방법[14]과 레이블 멱집합 방법[15]이 성능이 높았다[5, 8, 9]. 본 논문에서는 레이블 멱집합 방법으로 기본 분류기를 구성하고, 각 다중레이블들이 예측될 확률을 구한다. 다중레이블에 속한 각 레이블별로 예측될 확률을 더하여 정해진 문턱치보다 크면 예측결과로 판정한다. 각 다중레이블들이 예측될 확률을 정교하게 구하기 위하여 쌍별 비교를 통한 확률추정 [16-18]과 오류정정 출력코드[19-21]에 대한 확률추정 [22]을 비교하였다. 본 논문에서는 각 다중레이블이 예측되는 확률을 가중치로 사용하여 단일레이블들의 예측 확률을 계산한다. 이렇게 계산된 단일레이블의 예측 확률이 정해진 문턱치보다 크면 예측된 레이블로 판정한다. 따라서 높은 확률로 예측되는 다중레이블들에 여러 차례 속하는 단일레이블의 예측확률은 강화되며, 학습 자료에 나타나지 않은 형태의 다중레이블을 예측할 수 있다.
본 논문의 내용과 관련이 깊은 레이블 멱집합 분류, 쌍별 비교와 오류정정 출력코드를 사용한 다중클래스 확률추정에 대해 알아본다.
다중레이블 분류 방법은 각 사례와 관련된 여러 개의 레이블들을 동시에 예측하므로, 하나의 레이블만을 예측하는 단일레이블 분류에 비하여 분류의 정확도가 높지 않다. 본 논문에서 사용하는 다중레이블 분류 방법인 레이블 멱집합 방법[11-13, 15]은 학습 자료에 나타나는 다중레이블들을 새로운 단일 레이블로 정의하여 다중레이블 분류를 단일 레이블 분류로 변환하는 방법이다. 이 방법은 직접적으로 레이블간의 연관관계를 나타낼 수 있는 장점이 있지만, 새로 정의된 단일레이블의 수가 많으면 분류기의 성능이 저하되고, 충분한 학습 자료가 없는 레이블에 해당하는 모델은 학습이 어렵다. 이를 해결하기 위해 PS(pruned sets) 방법[15]은 많은 사례를 가지는 다중레이블만을 사용하고, 학습에서 제외된 사례들의 다중레이블은 학습 자료로 선택된 다중레이블을 포함하는 경우에 학습 자료에 재도입된다.
EPS(ensemble of PS)[15]는 학습 자료의 일부(63%)를 표본 추출하여 학습되어진 PS 분류기를 사용한다. 이러한 과정을 여러 번 수행하여 예측된 다중레이블들에 포함된 단일레이블들의 개수를 구하고, 이 개수가 문턱치보다 크면 예측된 것으로 판정한다. 이러한 앙상블 방법은 분류기를 구성하는데 사용된 학습 자료에 과도적합(over-fitting)되는 것을 완화시키며, 학습 자료에 포함되지 않는 새로운 형태의 레이블 부분집합을 예측 할 수 있다.
다중클래스에 대한 확률추정을 위하여 쌍별 비교(pair-wise comparison)를 사용할 수 있다. 쌍별 비교는 단일레이블 분류에서
PKPD[16]는 클래스 확률 추정을 위해 식 (1)의 간단한 계산을 사용한다. 과 일반적으로
반복계산으로 클래스 확률을 구하는 HT[17]는
를 최소화한다. 단,
WLW[18]는
2.3. 오류정정 출력코드를 통한 다중클래스 확률 추정
다중클래스 확률추정을 위해서 오류정정 출력코드를 사용할 수 있다[22]. 오류정정 출력코드는 여러 다중클래스 분류의 개념을 포괄하는 일반적 방법이다[19-21]. 이 방법에서는
학습과정에서는 코딩행렬의 각 열
단,
오류정정 출력코드를 사용한 분류 과정에서는, 입력 자료
본 논문에서는 오류정정 출력코드를 사용한 클래스 확률 추정을 위해 GBT(Generalized Bradley-Terry model)[22]를 적용하였다. 이 방법은 코딩행렬의 각 열
Ⅲ. 레이블 멱집합 분류와 다중클래스 확률 추정을 사용한 다중레이블 분류
본 논문에서는 단백질이 존재하는 세포내의 다중 위치를 예측하므로, 효과적인 다중레이블 분류방법이 필요하다. 따라서 관련연구로부터 단백질 세포내 위치 예측에 성능이 높다고 알려진 레이블 멱집합 방법을 변형하여 이용한다. 레이블 멱집합 방법은 다중레이블을 새로운 클래스로 정의하고, 다중클래스 분류를 사용하여 다중레이블을 예측한다. 앙상블 방법인 EPS는 레이블 멱집합 방법을 여러 번 수행하여 예측된 다중레이블들에 포함된 단일레이블들의 개수를 구하고, 이 개수가 문턱치보다 크면 예측된 것으로 판정한다. 본 논문에서는 레이블 멱집합과 마찬가지로 다중레이블을 예측하지만, 분류과정에서 다중레이블의 예측 확률을 추정하고, 추정한 예측 확률을 바탕으로 다중레이블에 포함된 단일레이블을 예측한다. 따라서 EPS처럼 예측된 단일 레이블의 개수만을 고려하지 않고 확률 정보를 이용하여 보다 정교한 예측이 가능하다.
제안한 방법은 학습의 첫 단계에서는 레이블 멱집합 분류기를 학습할 수 있을 정도의 자료 개수를 가진 다중레이블을 선정하여, 이후의 학습과 분류에 사용한다. 즉, 학습 자료에
예측의 첫 단계에서는 II장에서 알아본 PKPD, HT, WLW와 같은 쌍별 비교를 사용한 다중클래스 예측 확률 추정 방법과 오류정정 출력코드의 관점에서 GBT를 사용하여 다중클래스 예측 확률을 추정한다. 즉, 각 평가 자료가 각각
최종적인 분류는 간단한 수식 (6)을 사용하여, 미리 정해진 확률 문턱치 보다 큰 단일레이블로 결정하였다. 즉, 확률 벡터
제안한 방법은 식 (5)에서 보듯이, 높은 예측 확률
이 장에서는 단백질의 세포내 다중위치 예측에 대하여 다중레이블 분류방법들의 성능을 비교한다. 본 논문에서 제안한 방법은 클래스 확률추정 방법으로 쌍별 비교 방법인 PKPD[16], HT[17], WLW[18]를 사용하였고, 또 다른 확률추정 방법으로 GBT[22]를 사용하였는데, 오류정정 출력코드의 코딩행렬의 형태로서 쌍별 비교, 일대전부, Sparse를 사용하였고, Sparse는 개선된 코딩행렬 구성방법[21]도 적용하였다. 오류정정 출력코드에서 Dense 방법은 쌍별 비교, Sparse, 개선된 Sparse보다 성능이 높지 않고[20-21], 실험에 훨씬 많은 시간이 필요하므로 비교에서 제외하였다.
실험에는 14개의 세포내 위치 (centriole, cytoplasm, cytoskeleton, endoplasmic reticulum, endosome, extracell, golgi apparatus, lysosome, microsome, mitochondrion, nucleus, peroxisome, plasma membrane, synapse)로 구성된 인간 단백질 자료[1-5]를 사용하였다. 이 자료에서 2,580개 단백질은 하나의 세포내 위치, 480개는 두 개의 위치, 43개는 3개의 위치, 3개는 4개의 위치에 동시에 존재하며, 25% 이하의 적은 단백질 서열 동일성을 가지므로, 기본적인 서열 유사성만으로는 단백질의 세포내 위치 예측이 어려운 자료이다. 분류실험에는 자료를 균등하게 5개로 나누어, 하나는 평가에 사용하고 나머지 4개는 학습 자료로 사용하는 방법을 5회 반복하는 5겹 교차검증(fivefold cross-validation)을 사용하였다.
분류기의 특징벡터는 각 단백질 서열과 가장 유사한 단백질을 유전자 온톨로지를 가진 단백질 데이터베이스(http://www.ebi.ac.uk/GOA)에서 찾아, 그것의 유전자 온톨로지를 사용하는 방법을 사용하였다[1,2,4-7,9, 23]. 유전자 온톨로지는 분자적 기능, 생물학적 과정, 세포 요소의 관점에서 특징화한 용어로 유전자를 표현한 것으로, 각 단백질의 특징을 표현할 수 있다. 단백질의 세포내 위치에 따라 보다 판별력이 높게 나타내는 유전자 온톨로지를 가중하는 방법[23]을 사용하였고, 가장 유사한 두 개의 서열에서 나타나는 유전자 온톨로지의 빈도를 이용하는 방법[9]을 사용하였다.
분류 방법의 성능 평가는 예측된 다중레이블이 실제 다중레이블과 일부만 일치하는 경우를 고려하는 다중레이블 분류의 평가 척도를 사용한다[11-13]. 다중레이블 평가척도에서 부록의 식 (S1)~(S6)의 사례기반 방법은 각 사례에 대해 실제 레이블과 예측된 레이블간의 차이를 평균하고, 식 (S7)~(S12)의 레이블기반 방법은 각각의 레이블에 대해서 예측성능을 구한다. 식 (S13)은 평가척도들을 합해 간략한 비교가 가능하게 한다[9].
본 논문과 같은 실험 자료를 사용하는 논문[3]에서는 최근접-이웃 분류기들을 조합하는 Hum-mPLOC 2.0[1]은
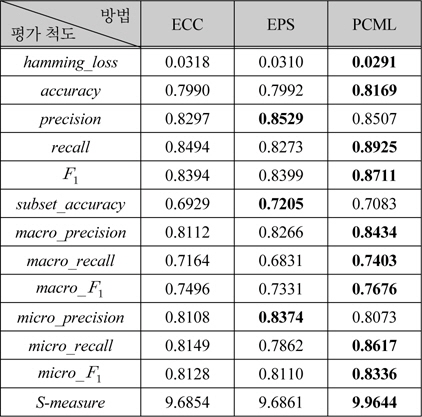
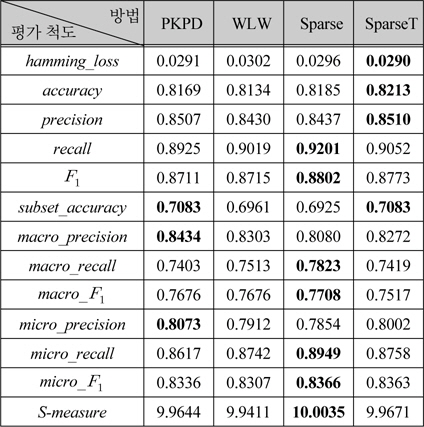
표 1의 ECC, EPS는 Mulan 라이브러리[24]를 사용한 결과[9]로서, 이전 연구 결과[5]와 유사하다. 본 논문에서 제안한 PCML(Probabilistic Combination of Multi- Labels)은 클래스 확률추정으로 PKPD를 사용한 결과 이며, 대부분의 평가 척도에서 ECC나 EPS보다 높은 성능을 보였다. EPS와 ECC의 성능을 비교해보면,
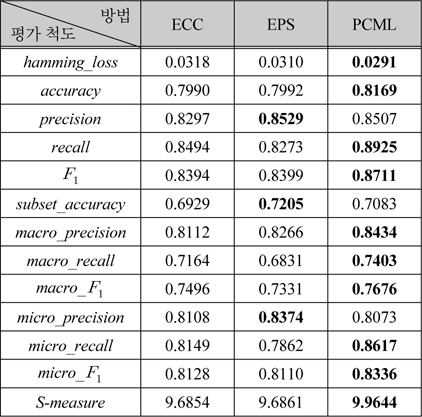
ECC, EPS와 PCML의 성능비교
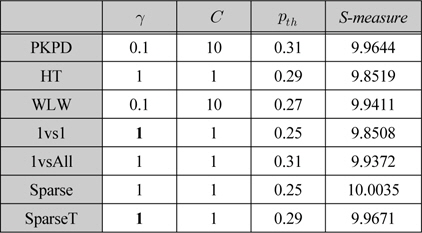
PCML에서 레이블 멱집합 분류기를 구현하기 위하여 LIBSVM[25]을 사용하였고, 가우시안 커널의 𝛾 =0.001, 0.01, 0.1, 1, 비용 파라미터
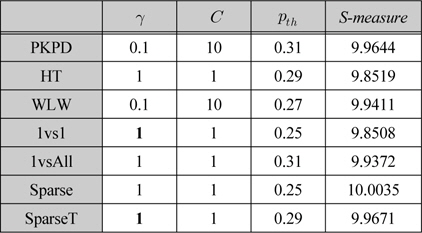
다중클래스 확률 추정 방법들의 성능비교
표 3은 표 2에의 파라미터를 사용한 PKPD, WLW, Sparse, SparseT의 성능을 여러 평가척도로 나타내었다. Sparse가 가장 높은
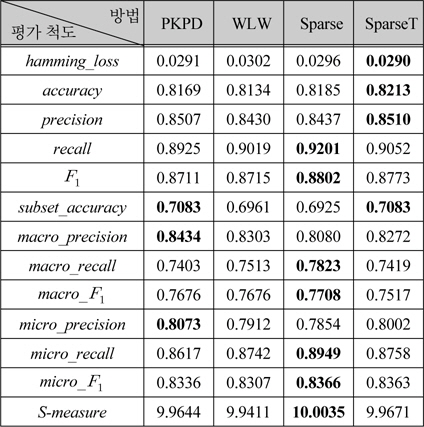
다중클래스 확룰추정 방법들의 성능비교
본 논문에서는 단백질의 다중 세포내 위치 예측에 적합한 다중레이블 분류방법을 제안하였다. 첫 번째로 레이블간의 연관관계를 효과적으로 모델링할 수 있는 다중레이블 분류방법이 다중 세포내 위치를 보다 정확하게 예측할 수 있다는 점을 이용하였다. 본 논문에서는 레이블 멱집합 방법처럼 다중레이블 자체를 하나의 단일레이블로 구성하였다. 두 번째로 단백질 세포내 다중위치 자료는 각 다중레이블들에 대한 학습 자료가 충분하지 않으므로, 앙상블 방법을 이용하는 대신에, 각기 다른 확률로 예측된 여러 다중레이블을 동시에 고려하여, 중복된 단일레이블의 예측확률을 얻었다. 각 다중레이블에 대한 정확한 예측확률을 구하기 위하여 여러가지 다중클래스 확률 추정 방법을 적용하였다.
제안한 PCML방법은 단백질 세포내 위치예측에 효과적인 EPS와 ECC보다 대부분의 평가척도에서 우수 하였고, 제안한 방법에 적용한 다중클래스 확률 추정 방법에서 PKPD와 Sparse, SparseT가 효과적이었다.
본 논문에서는 최종적인 분류 방법으로 간단한 확률 문턱치를 사용하였는데, 이후에는 각 단일레이블의 특성을 고려하여 문턱치를 구성하고, 앙상블 방법을 추가적으로 적용할 예정이다. 또한, 제안한 방법을 동물, 식물, 곰팡이, 바이러스 등의 여러 영역의 세포내 위치 예측에 적용할 계획이다.