BACE2 is a membrane-bound aspartic protease that is highly homologous with BACE1. While BACE1 processes the amyloid precursor protein (APP) at a key step in generating β-amyloid peptide and presumably causes Alzheimer’s disease (AD), BACE2 has not been demonstrated to be involved in APP processing directly, and its physiological functions are unknown. To determine its function and to develop inhibitors from marine sources, we constructed an overexpression vector for producing BACE2. The gene encoding human BACE2 protease was amplified using the polymerase chain reaction and cloned into the pET11a expression vector, resulting in pET11a/BACE2. Recombinant BACE2 protease was overexpressed successfully in
치매(dementia)는 신경세포의 노화에 의한 퇴행성 뇌질환으로 뇌혈전 및 뇌출혈 등에 의한 뇌혈관의 장애로 생기는 뇌혈관성 치매와 알츠하이머병(Alzheimer's disease)에 의한 것으로 분류할 수 있다. 뇌혈관성 치매는 그 원인이 명확히 구명되어 있어 진단 및 치료가 용이하지만, 알츠하이머병은 현재까지 정확한 원인이 밝혀져 있지 않다. 알츠하이머 환자의 뇌를 검사한 결과 병리조직학적으로는 뇌의 전반적인 위축, 뇌실의 확장, 신경섬유의 다발성 병변(neurofibrillary tangle)과 초로성 반점(neuritic plaque) 등의 특징을 보인다. 이 병의 원인 중의 하나인 이상단백질 축적설은 베타-아밀로이드(β-amyloid)라는 40-42개의 아미노산 잔기로 구성된 펩타이드가 과도하게 만들어져 뇌세포에 축적되면, 뇌의 신경세포 기능이 떨어져서 알츠하이머병이 발생한다고 추정하고 있다(Hardy, 1997; Selkoe, 1997). 베타-아밀로이드는 β-secretase와 γ-secretase라는 단백질 가수분해효소에 의해 아밀로이드 전구체 단백질(Amyloid Precursor Protein: APP)의 베타 부위와 감마 부위가 절단되어 생성된다. APP의 감마 부위를 절단하는 활성을 가진 프로테아제는 presenilin-1 (PS1)에 의해 조절되거나 PS1 자신인 것으로 추정되고 있다(Wolfe et al., 1999). 한편, APP의 베타 부위로 β-secretase로 확인된 BACE1 (EC 3.4.23.46)에 의해 절단되며 베타-아밀로이드 생성의 속도조절단계로 확인되었다(Sinha and Lieberburg, 1999). 따라서 BACE1은 알츠하이머병의 치료제 개발에 중요한 표적단백질로 인식되고 있다(Lin et al., 2000).
알츠하이머병의 원인을 명확히 규명하고 치료제 개발을 성공적으로 달성하기 위하여 표적단백질 및 관련 효소의 생리적 및 병리학적 기능의 깊은 이해가 요구된다. BACE1과 동시에 발견된 human aspartic protease인 BACE2 (EC 3.4.23.45)는 down syndrome region이 존재하는 chromosome 21의 21q22.3에 그 유전자가 존재하고, 효소촉매 활성 잔기인 두 개의 aspartic acid를 포함한 특이적인 아미노산 배열이 BACE1과 높은 상동성이 있는 것으로 확인되었다(Saunders et al., 1999; Yan et al., 1999; Lin et al., 2000). 생리적인 기능이 잘 알려져 있는 human aspartic protease로는 pepsin & gastricsin (food digestion), cathepsin D & cathepsin E (intracellular protein catabolism), 및 rennin (blood pressure regulation) 등이 있다(Tang and Wong, 1987). 진핵생물에 존재하는 aspartic protease들은 단백질뿐 만아니라 유전자 단계에서도 상동성을 가진다. 전형적인 aspartic protease의 합성은 일반적으로 세포내의 구획에서 유도되어 single-chain zymogen으로 합성된다. 이렇게 합성된 전구체는 자가분해반응에 의해 pro-segment에 해당되는 N-말단의 약 45개 아미노산 잔기가 절단되어 성숙체 효소로 활성화된다. 그렇지만 pro-renin 및 pro-BACE1을 포함하는 소수의 aspartic protease는 다른 protease에 의해 활성화된다(Jutras and Reudelhuber, 1999; Creemers et al., 2001). 대부분의 aspartic protease의 기질 결합 잔기를 포함하는 촉매활성 부위 부근의 3차원 구조는 유사한 것으로 보고되어있다(Tang and Wong, 1987).
BACE2 유전자는 다운증후군 관련 유전자들이 위치하고 있는 인간 염색체 21에 존재하므로 알츠하이머병 뿐만 아니라 다운증후군에도 관련이 있는 제2의 β–secretase로 작용할 가능성이 있다고 제안하였다(Saunders et al., 1999; Solans et al., 2000). 이러한 BACE2 유전자 위치는 BACE2와 BACE1은 구조적으로 유사할 뿐만 아니라 기능적으로도 유사할 것이라는 여러 연구자들의 예측과 일치한다. 더구나 BACE2 및 BACE1은 뇌의 모든 조직에서 발현되었다(Hussain et al., 2000). 또한 BACE2는 BACE1과 같이
알츠하이머병 환자들의 약물치료법으로는 acetylcholinesterase inhibitor, Aricept, Exelon, Reminyl, NMDA receptor antagonist를 비롯한 다양한 약물이 널리 사용되고 있다. 이러한 알츠하이머병의 치료제들은 이 병을 근본적으로 치료하는 것이 아니라 단지 인지 기능을 개선시키는 약물로 이 질환의 진행 속도를 다소 늦추어 주는 역할만을 한다(Han SH, 2009). 따라서 수 많은 연구자들은 알츠하이머병을 근본적으로 치료할 수 있는 치료제 후보물질로 β-secretase 효소활성을 억제하는 저해제 개발에 관한 연구를 진행하고 있다. 대표적인 억제제로는 peptide-based inhibitor, Isophthalamide-based inhibitor, acyclic acylguamidines, cyclic acylguamidine inhibitor 등의 후보물질들이 개발되고 있으나 알츠하이머성 치매에 효과적인 완전한 치료제는 아직 알려져 있지 않다(Stachel SJ, 2009). 한편, 해양생물 종의 다양성과 진화과정의 독자성 및 서식환경의 특이성이라는 요인에 의하여 기존의 육상생물이 보유하고 생산할 수 없는 다양한 생체분자 및 화학물질을 보유하고 있을 가능성이 높아 새로운 알츠하이머성 치매 억제제 개발에 이 이상의 좋은 소재는 없을 것으로 생각된다.
따라서 본 연구에서는 BACE2의 효소학적 특성 규명 및 해양생물 유래 알츠하이머병 관련 저해활성물질 탐색에 필요한 활성형 BACE2를 생산하는 최적 방법을 고안하고자 하였다. 본 연구에서는 BACE2의 대량발현을 위하여 다양한 발현계를 작성하여 효소 생산에 최적인 발현벡터 및 단백질 발현 방법을 검토하였다. 그 결과 pET11a/BACE2 발현벡터를 이용한 BACE2 단백질의 발현량이 최적인 것으로 확인되었다. 또한 대량 발현된 BACE2의 inclusion body를 refolding한 후, Sephacryl S-300 gel filtration 및 ResourceQ 컬럼 크로마토그래피법을 이용한 2단계의 간단한 정제방법을 확립하였으며, 정제한 효소의 다양한 효소학적 특성을 규명하였다.
본 연구에서 사용한
BACE2의 유전자는 human placenta cDNA library (Clontech)로부터 아래의 oligonucleotide primers를 사용하여 polymerase chain reaction (PCR) 법으로 유전자를 증폭하여 사용하였다. Forward primer (5’-GGATCCGCCGCCCCGGAGCTGGCCCCCGCGC-3’)에는
BACE2의 단백질 발현은 pET11a/BACE2로 형질전환시킨 대장균 BL21 (DE3)의 overnight culture를 ampicillin 50 μg/mL가 포함된 Luria-Bertani broth (4 liter) 배지에서 약 2-3시간 정도 37℃에서 진탕 배양하였다. OD600이 0.6-0.8이 되었을 때 단백질 발현유도제인 IPTG (최종 농도, 1 mM)를 투여하여 37℃에서 3시간 더 진탕 배양한 후, 원심분리(5,000×
단백질 리폴딩이 완료된 효소용액은 ultrafiltration 법으로 농축하여, 원심분리(12,000×g, 30 min, 4℃)하였다. 상층액을 완충액 C (0.4 M Urea를 포함하는 20 mM Tris/HCl, pH 8.0)로 평형화시킨 Sephacry S-300 (2.6×60 cm, prepacked, GE Healthcare)을 사용한 gel filtration 방법으로 refolded 단백질을 정제하였다. 효소 단백질이 함유된 분획을 회수하여 완충액 C로 평형화시킨 Resource-Q (1.6×3 cm, prepacked, GE Healthcare) 컬럼에 주입하고, 충분한 양의 완충액 C로 컬럼 내의 비흡착단백질을 제거하기 위해 세척한 후, 완충액 C의 NaCl 농도가 0-0.5M에 이르도록 농도 기울기법으로 분별 용출하였다. 효소 단백질이 함유된 Resource-Q 컬럼 분획을 회수하여 완충액 C로 투석한 다음, 원심분리(12,000×g, 30 min, 4℃)하였다. 이상의 과정으로 정제한 효소를 4℃에 보관하면서 정제 효소의 활성 및 특성실험에 사용하였다. 정제 효소의 단백질 정량은 소 혈청 알부민을 표준 단백질로 사용하여 Pierce BCA Protein Assay kit (Thermo)으로 측정하였다.
BACE2의 효소 활성 측정은 Kim et al. (2002)의 방법으로 다음과 같이 결정하였다. 본 실험에서 사용한 기질은 화학적으로 합성한 펩타이드(Val-Gly-Ser-Gly-Val-Leu-Leu-Ser-Arg-Lys: NCH-γ peptide)을 사용하여 분석하였다(Schroeter et al., 1998). 일반적인 효소활성은 총 40 μL 반응액 중에 50 mM sodium phosphate (pH 6.5) 완충액, 0.8 mM 기질, 및 적당한 효소농도(6.26 μM)가 되도록 혼합하여 37℃에서 30분간 반응시켜 측정하였다. 효소 반응은 기질의 첨가에 의해 개시되고, 2% trifluoroacetic acid를 40 μL 첨가하여 반응을 종결시켰다. 이 반응 혼합물을 3,000×
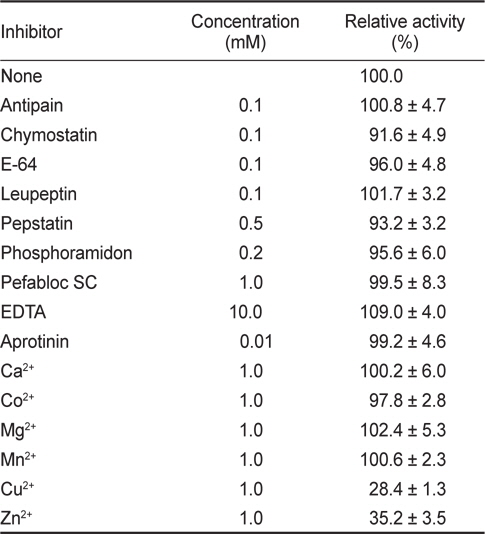
무기금속 이온과 효소 저해제가 BACE2의 활성에 미치는 영향을 조사하였다. 무기금속이온과 저해제의 최종농도는 각각 0.1-1 mM이 되도록 첨가하여 효소활성을 측정하였다. 정제 단백질의 분자량은 MALDI-TOF mass spectrometer (Applied Biosystems Voyager-DE STR mass spectrometer, USA)를 사용하여 측정하였다. N-말단 아미노산 서열 분석은 Porcise® cLC System Model 492 cLC (Applied Biosystem, USA)를 사용하여 Edman 분해법으로 분석하였다.
BACE2는 세포막에 결합되어있는 human aspartic protease로 그 유전자는 다운증후군 관련 유전자가 많이 존재하는 인간 염색체 21번에 내포되어있을 뿐만 아니라 알츠하이머병을 야기시키는 원인으로 추정되고 있는 β-amyloid peptide 생성에 중요한 역할을 담당하는 효소인 BACE1과 높은 상동성을 가지고 있다(Solans et al., 2000). 이와 같이, 인간 질병에 관여할 가능성이 높은 BACE2 protease의 효소학적 특성 및 해양생물 유래 천연저해물질을 탐색하기 위해서는 다량의 활성형 BACE2를 생산할 필요가 있다. 본 연구에서는 효소활성을 가지고 있는 BACE2를 간편한 방법으로 대량으로 생산하기 위하여, 그 유전자를 함유하고 있는 human placenta cDNA library를 사용하여 PCR법으로 BACE2의 유전자를 증폭하고, pET11a 발현 벡터의
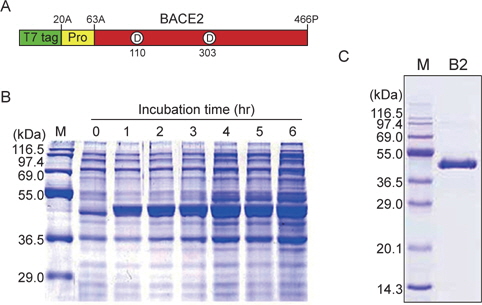
BACE2의 최적 발현조건을 검토하기 위하여 대장균 BL21, BL21(DE3), BL21(DE3)pLysS 및 TB1을 pET11a/BACE2 발현벡터로 각각 형질전환시켰다. 각 형질 전환된 숙주 내의 BACE2 발현은 각각의 대장균을 37℃에서 약 2시간동안 배양하여, 1 mM IPTG (최종 농도)를 첨가하여 단백질 발현을 유도하였다. 이들 대장균을 동일조건 하에서 4시간 동안 배양 후, 각 배양균체의 단백질을 12.5% SDS-PAGE 전기영동을 실시하여, 그 효소 발현량을 비교 검토하였다. 그 결과, BL21(DE3) 대장균에서 recombinant BACE2 (461 아미노산: 49.2 kDa)에 해당되는 새로운 단백질의 발현을 확인할 수 있었다(Fig. 1B). 또한, 단백질 발현에 영향을 미치는 단백질 유도제 첨가 후의 발현 시간을 최적화하기 위하여 발현시간을 다양하게 변화시키면서 BACE2를 발현시킨 후, 각 균체를 12.5% SDS-PAGE 전기영동법으로 단백질 발현량을 비교 검토하였다. Fig. 1B에 나타낸 것과 같이, 전기영동 겔 사진의 lane 2부터 lane 8까지는 BL21(DE3)/pET11a/BACE2 배양액에 단백질 발현 유도제를 첨가한 후, 매 시간 별 반응액의 전기영동 패턴을 나타내고 있다. Blank (lane 2)와 각 배양 시간 별 반응액의 전기영동 패턴을 비교한 결과, 배양시간이 증가함에 따라 50 kDa 부근에 새로운 단백질 밴드가 비례적으로 두꺼워지는 것을 확인할 수 있다. 이 발현 단백질 밴드의 분자량은 재조합 BACE2의 분자량 49.2 kDa과 일치한다. 발현 벡터 pET11a/BACE2에 의해 대량 발현된 BACE2의 전기영동 젤을 densitometer로 분석한 결과, 총 대장균 단백질의 약 10%까지 발현되는 것으로 확인되었다. 따라서, BACE2 단백질의 발현량을 효율적으로 높이기 위해서는 단백질 발현 유도제를 첨가한 후 약 3시간 이상 배양하는 것이 최적인 것으로 확인되었다(Fig. 2B).
BL21(DE3) 대장균을 pET11a/BACE2 플라스미드로 형질전환시켜 ‘재료 및 방법’에서 설명한 것과 같이 재조합 BACE2를 대량 발현하였다. 이 균체를 세척, 세포막 파쇄, 및 재세척 과정을 거쳐 inclusion body를 분리하였다. 이 BACE2 inclusion body를
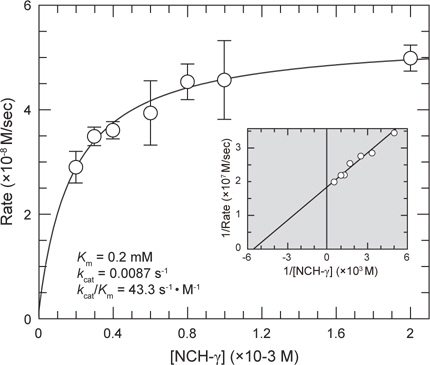
재조합 BACE2를 정제하기 위하여, 위의 상층액을 Sephacryl S-300 gel filtration 및 Resource-Q 컬럼 크로마토그래피 방법을 이용하여 BACE2를 완전 정제하였다. 최종적으로 정제한 효소의 정제도를 확인하기 위하여 전기영동을 실시한 결과, 분자량 50 kDa 부근에서 단일 밴드가 확인되어(Fig. 1C, lane 2), 재조합 BACE2 단백질이 균일하게 정제된 것으로 판단되었다. 이상의 정제과정의 결과, 4 Liter의 배양액으로부터 완전 정제한 BACE2 protease를 약 100 mg 생산할 수 있는 것으로 확인되었다. 정제한 BACE2의 효소활성은 합성 펩타이드 기질, NCH-γ peptide (Val-Gly-Ser-Gly-Val-Leu-Leu-Ser-Arg-Lys)를 사용하여 Leu잔기와 Ser잔기 사이의 펩타이드 결합의 절단속도로 결정하였다. 그 결과 BACE2 protease의 특이적인 효소 활성은 15,120 (pmol/min·mg)으로 확인되었다. 이러한 결과는 human BACE2를 pET11a/BACE2 발현벡터를 사용하여 대장균에서 발현시켜 리폴딩 및 정제한 재조합 BACE2 protease는 충분한 효소활성을 가지고 있는 것으로 판단된다. 본 효소의 반응속도상수(
[Table 1.] Effects of protease inhibitors and metal ions on the activity of BACE2.1
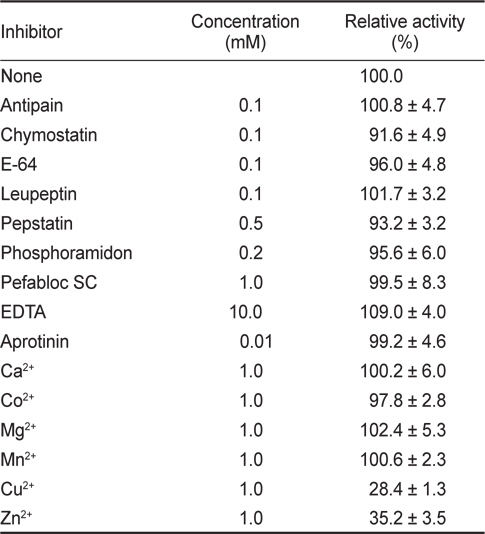
Effects of protease inhibitors and metal ions on the activity of BACE2.1
>
BACE2의 분자량 및 N-말단 아미노산 서열 결정
최종적으로 정제한 재조합 BACE2의 분자량은 MALDI-TOF mass spectrometer를 이용하여 측정한 결과 m/z=49183.51(M+H)+ 이었다(결과 생략). 이론적으로 pET11a/BACE2 벡터에서 발현되는 재조합 BACE2는 461개의 아미노산으로 구성되어있고, 그 분자량은 49,173 kDa인 단백질이 발현되도록 디자인하였다. 본 실험에서 정제한 단백질과 재조합BACE2의 이론적인 분자량을 비교해 보면 그 분자량이 약 0.02%의 오차 범위인 것으로 확인되어 정제한 단백질과 재조합 BACE2의 분자량은 일치하는 것으로 확인 되었다. 한편, 정제한 효소의 N-말단 아미노산 배열을 알아보기 위해 아미노산 서열 분석을 한 결과, Ala-Ser-Met-Thr-Gly의 5개의 아미노산 배열을 확인하였다. 이 배열은 재조합 BACE2의 1번 아미노산부터 5번 아미노산까지의 배열에 해당한다. 위의 정제한 단백질의 분자량 측정 및 N-말단 아미노산 분석 결과를 종합해 보면, 본 실험에서 생산된 단백질은 재조합 BACE2 protease (461개 아미노산; 분자량: 49,173)인 것으로 명확히 확인되었다.
이상의 결과처럼, 우리는 본 연구에서 구축한 pET11a/BACE2 발현 벡터를 이용하여 human BACE2 protease를