We applied a combination of most probable number-polymerase chain reaction (MPN-PCR) methods using a PCR procedure targeting the H-NS (VP1133) gene to detect
장염비브리오(
식품의약품안전처의 식중독 통계시스템 자료에 의하면 2002년부터 2013년까지 12년간 우리나라에서 발생한 장염비브리오에 의한 식중독 사고는 주로 여름철에 집중적으로 발생하며 사고건수 및 환자수는 전체 세균성 식중독 사고의 16.5% 및 8.2%를 차지하고 있다(MFDS, 2014). 사고건수로는 병원성대장균, 살모넬라에 이어 3위에 환자수로는 병원성대장균, 황색포도상구균, 살모넬라에 이어 4위를 차지하고 있기 때문에 수산물의 안전성 확보는 국민보건위생상 매우 중요하다(MFDS, 2014). 해수에서 장염비브리오의 존재는 수온과 밀접하여 수온이 17℃ 이상으로 상승하는 하절기에는 자유 유영의 형태로 해수에서 쉽게 검출되나 수온이 10℃ 이하로 낮아지면 검출빈도는 급격히 떨어지며 동절기에는 저질 또는 동물성 플랑크톤의 키틴질 등에 부착하여 월동한다고 보고되어 있으나 상세한 내용에 대해서는 아직 규명되어 있지 않다(Kaneko and Colwell, 1975; Makino et al., 2003). 장염비브리오는 최적의 조건에서 균의 세대시간이 8-12분 정도로 어느 세균보다도 빨라 단시간(3-4시간)에 식중독을 일으킬 수 있는 균수에 도달할 수 있기 때문에 장염비브리오에 의한 식중독 사고 예방을 위해서는 신속 검출이 매우 중요하다(Makino et al., 2003). 수산물로부터 장염비브리오의 검출방법에는 PCR assay, most probable number(MPN) (Copin et al., 2012), DNA hybridization (Wang et al., 2011), Chromogenic agar (Su et al., 2005)를 이용하는 방법들이 보고되어 있으나 이중에서 PCR assay는 다른 방법에 비해 신속하며 검출한계가 뛰어난 점 등의 장점으로 인하여 많이 이용되고 있다(Kim et al., 1999; Kim et al., 2008; Yu et al., 2010). 지금까지 PCR assay에 사용된 장염비브리오의 표적 유전자로는
H-NS (histone-like nucleoid structuring) 단백질은 저분자 물질로 AT-rich DNA에 결합하여 병원성 유전자를 포함한 다수 유전자의 전사를 양성 또는 음성적으로 조절하는 전사조절 인자로 알려져 있으며 병원성대장균, 살모넬라,
본 논문은 해수 또는 수산물에 존재하는 장염비브리오의 정량을 위하여 most probable number (MPN)와 H-NS 유전자를 표적으로 하는 PCR assay 방법을 병행하여 해수 및 바지락 중의 장염비브리오 농도를 정량하는 조건을 검토하였다.
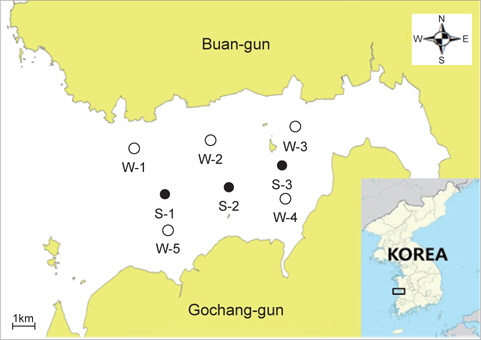
해수 및 바지락 시료의 채취지점은 Fig. 1에 제시하였다. 해수는 2013년 6월부터 11월까지 전라북도 곰소만 해역 5곳의 조사지점에서 매월 1회씩 총 6회에 걸쳐 채취하였으며, 바지락은 3곳의 조사지점에서 2013년 7월과 8월 총 2회 채취하였다. 해수채취는 만조 시 각 조사지점에서 표층용 채수기를 사용하여 멸균된 250 mL 광구병에 직접 채취하였으며, 바지락은 간조 시 각 조사지점에서 채취도구를 사용하여 직접 채취하였다. 채취한 해수 및 바지락 시료는 얼음이 채워진 아이스박스에 넣어 10℃ 이하로 유지하여 실험실로 운반한 후 미생물시험 을 실시하였다. 해수의 수온, pH, 염분 및 용존산소는 수질분석기(YSI556 multiprobe system; Yellow Springs, YSI Life Science, OH, USA)를 사용하여 현장에서 측정하였다.
PCR assay에 의한 장염비브리오의 최소 검출농도를 측정하기 위하여 3% sodium chloride이 첨가된 Luria-Bertani (1% tryptone, 0.5% yeast-extract, 3% NaCl) broth 에서 하룻밤 배양한 장염비브리오 RIMD2210633 균주를 원심 분리(12,000rpm, 2 min)하여 집균하였다. 여기에 1.0 mL의 PBS (phosphate buffered saline, pH 7.2)를 가하여 현탁 후 원심 분리하여 PBS로 재차 현탁 하였다. 균수 측정은 현탁액 10 μL를 counting chamber (Paul Marienfeld Gmbh&Co, KG Lauda-Konigshofen, Germany)에 넣고 광학현미경(Olympus CX31RBSF, Olympus Optical Co., LTD. Tokyo, Japan)하에서 균수를 직접 계측하여 계산하였으며 적절한 농도까지 10진법으로 희석하여 PCR assay에 사용하였다.
해수 및 패류시료 중의 장염비브리오 정량을 위해 Recommended Procedures for the Sea Water and Shellfish (APHA, 1970)에 준하여 시험하였다. 해수는 최종농도 2%의 sodium chloride이 첨가된 alkaline peptone water (Merck, Darmstadt, Germany) 10 mL에 3개 시험관법으로 10, 1, 0.1 및 0.01 mL씩 접종한 후 35℃에서 16시간 정치 배양하였다. 또한 바지락은 무균적으로 탈각 후 육 25 g을 멸균된 Waring Blender cup (Torrington, USA)에 넣고 여기에 9배 량의 멸균인산완충희석액(phosphate buffered saline, pH7.4)을 첨가하여 90초 동안 균질화한 다음 10진법으로 적절한 단계까지 희석하여 해수와 동일하게 3개 시험관법으로 접종한 후 35℃에서 16시간 정치 배양하였다. 각 배양액 1.0 mL를 eppendorf tube에 취해 원심분리(12,000 rpm, 2 min)한 후 배양액은 완전히 제거하여 균체를 회수하였다. 여기에 멸균 증류수 0.2 mL를 가하여 현탁 후 98℃에서 5분간 열처리 후 H-NS 유전자 증폭을 위한 PCR assay용주형 DNA로 사용하였다. H-NS 유전자의 증폭산물이 확인된 시험관은 양성으로 판정하여 최확수(most probable number, MPN)에 적용하여 100 mL 또는 100 g 중에 존재하는 장염비브리오의 균수로 계산하였다.
유전자 증폭에는 EmeraldAmp GT PCR Master Mix (Takara, Japan)를 사용하였다.
>
PCR assay에 의한 장염비브리오의 검출 최소농도의 검토
PCR assay에 의한 장염비브리오의 검출한계에 관한 보고는 다수 존재한다. 염색체 DNA를 주형으로
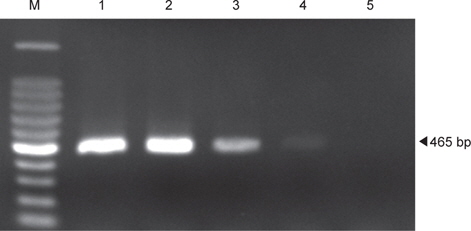
H-NS 유전자를 표적으로 하는 PCR assay에 의한 장염비브리오 균체의 검출 최소농도를 검토하기 위하여 균체 농도를 반응액에 0.8×10⁴CFU에서 0.8×100 CFU 농도가 되도록 조정하여 첨가하였다. Fig. 2에 제시한 바와 같이 H-NS 유전자의 증폭은 0.8×100 CFU 농도에서는 DNA의 증폭산물이 확인되지 않았지만 나머지 0.8×10⁴CFU에서 0.8×10¹ CFU까지 농도에서는 예상 증폭 단편인 465 bp의 DNA 증폭산물이 확인되었다. 그러나 0.8×10¹ CFU 농도의 반응구에서는 다른 농도에 비해 DNA 밴드는 미약하게 나타났다. 또한
[Table 1.] Oligonucleotide primers used in this study

Oligonucleotide primers used in this study
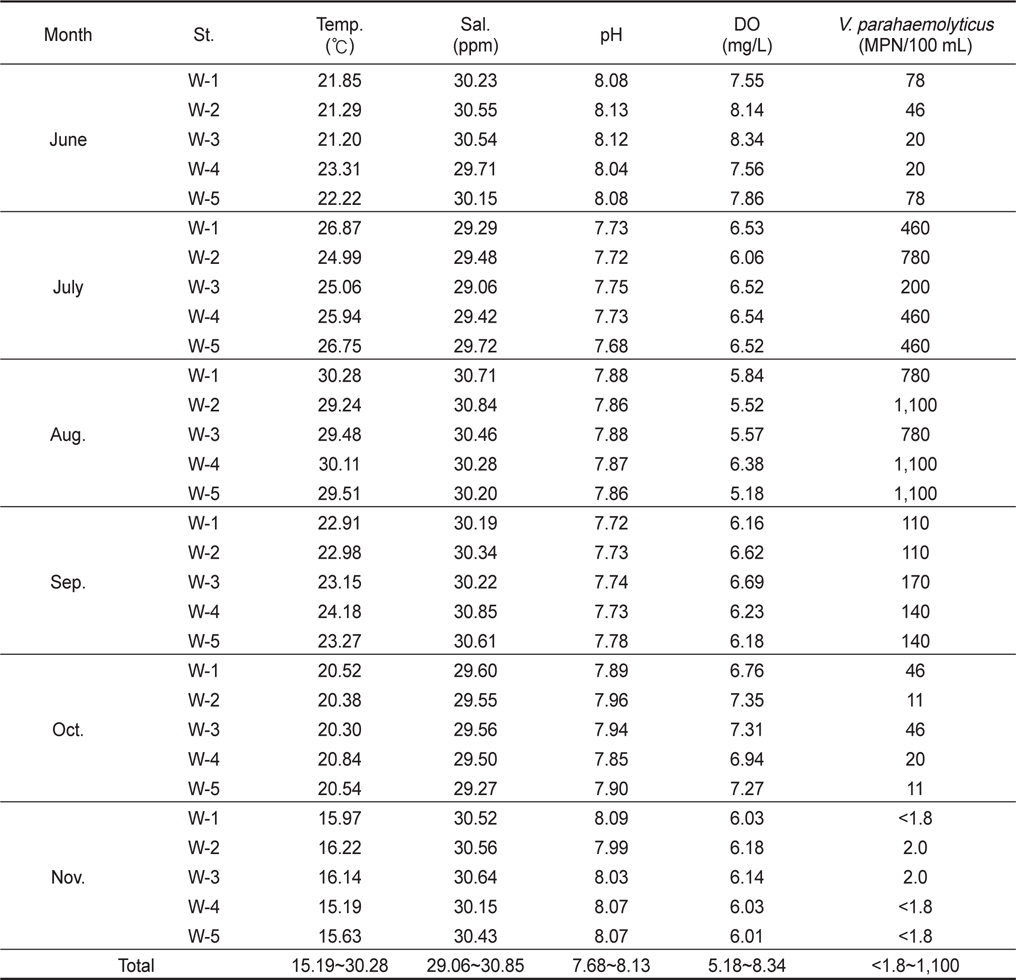
전라북도 부안군 곰소만 표층 해수 중의 장염비브리오 농도를 측정하기 위하여 최확수법과 H-NS 유전자의 primer set를 사용한 PCR assay의 병용에 의해 2013년 6월부터 11월까지 6개월 동안 총 30개의 해수 시료를 대상으로 장염비브리오의 농도를 측정하였다. 각 해수 시료의 온도, 염분, pH 및 용존산소에 대한 측정치는 Table 2에 제시하였다. 수온은 6월에 21-23℃ 였으나 점점 상승하여 8월에는 30℃ 전후로 가장 높게 측정되었으며 이후 점차 하락하여 11월에는 15-16℃ 전후로 측정되었다. 염분과 pH는 월별에 따른 차이는 거의 나타나지 않는 안정한 값을 나타내었으며, 용존산소는 수온이 높은 8월이 다른 월에 비해 가장 낮게 측정되었다. 해수 채취지점이 곰소만으로 한정되어 있기 때문에 정점에 따른 수온, 염분, pH 및 용존산소 등의 수치는 거의 차이가 없는 것으로 파악되었다. 장염비브리오의 농도는 수온이 21-23℃ 전후인 6월의 경우 20-78 MPN/100 mL(평균 48.4 MPN/100 mL), 7월에는 200-780 MPN/100 mL (평균 472 MPN/100 mL), 8월에는 780-1,100 MPN/100 mL (평균 972 MPN/100 mL), 9월에는 110-170 MPN/100 mL (평균 134 MPN/100 mL), 10월에는 11-46 MPN/100 mL (평균27 MPN/100 mL), 11월에는 <1.8-2.0 MPN/100 mL (평균 1.9MPN/100 mL)로 측정되었다. 곰소만 해수 중의 장염비브리오 농도는 수온이 가장 높은 8월에 가장 높게 측정되었으며 수온 저하와 동반하여 장염비브리오의 농도도 낮아지는 것으로 확인되었다. DePaola et al. (1990)은 미국 연안 해수 중의 장염비브리오 농도는 17 CFU/100 mL이며, 다른 요인보다 수온이 장염 비브리오 농도 형성에 가장 밀접하게 작용한다고 보고하고 있다. 또한 Cantet et al. (2013)은 프랑스 지중해 연안 해수 중의 장염비브리오 농도는 1.0에서 >1.1×10²MPN/100 mL로 측정 되었다는 보고도 있는데 이 결과는 곰소만 해수보다 약 10배 정도 낮은 수치이다. Table 2의 결과를 종합해 보면 해수 중의 염분, pH 및 용존산소와 장염비브리오의 검출농도와는 상관관계는 희박하였다. 또한 해수채취 지점에 따른 장염비브리오 농도에는 약간의 차이는 있었지만 대체로 안정적으로 분포하고 있었다. 결론적으로 해수에서 장염비브리오의 검출농도는 해수의 온도와 상관관계가 가장 밀접하여 해수의 온도가 높아질수록 장염비브리오 농도는 높아지고 수온이 낮아지면 장염비브리오의 농도도 함께 낮아지는 것으로 확인되었다. 수온이 16℃ 이하인 11월의 경우에는 장염비브리오의 검출은 거의 확인되지 않았으며 이는 기존의 보고와 거의 동일한 결과이다(Kaneko and Colwell, 1975).
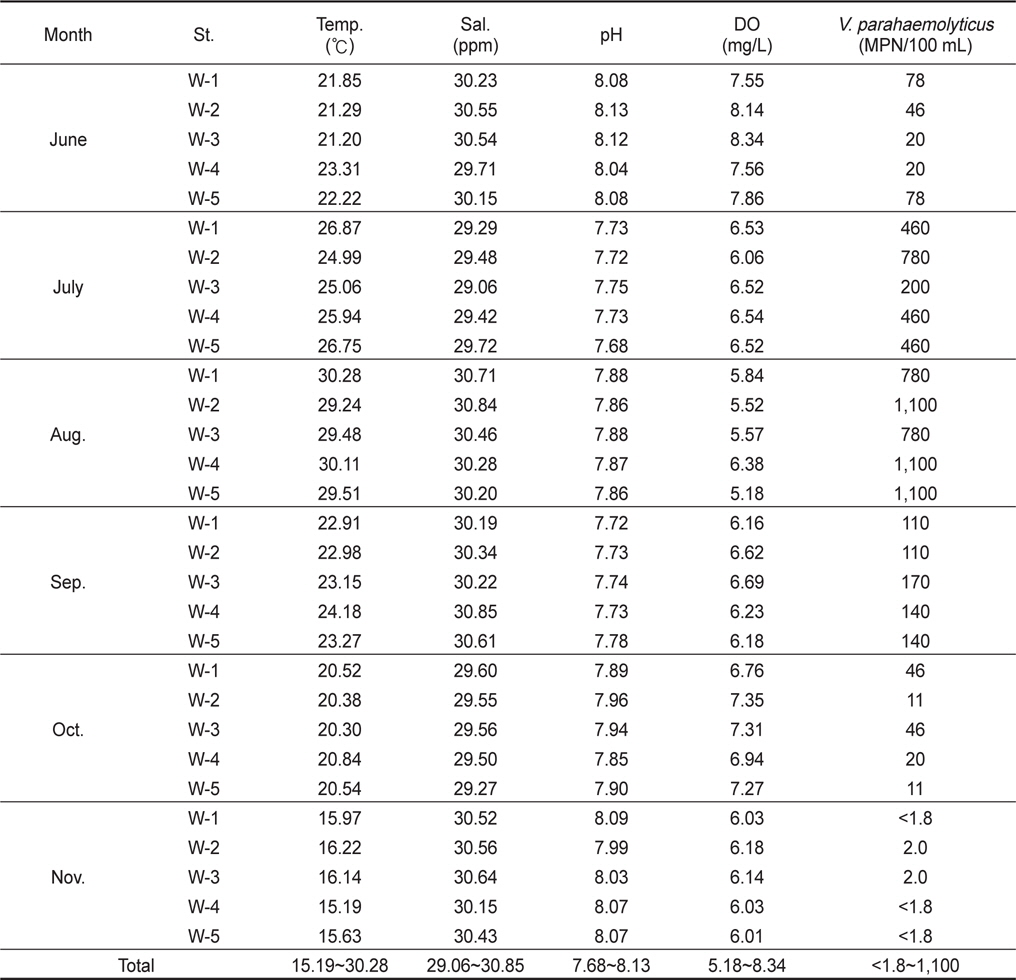
Levels of Vibrio parahaemolytcius in surface seawater in Gomso Bay from June to November 2013
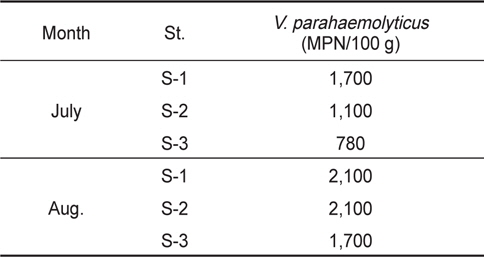
곰소만에서 생산되는 패류종인 바지락 중의 장염비브리오 농도를 측정하기 위하여 2013년 7월과 8월 2개월간 Fig. 1에 제시한 지점에서 바지락을 채취하여 총 6점의 바지락에 대해 장염비브리오의 농도를 MPN-PCR assay를 병행하여 측정하였다. 그 결과 7월의 경우, 장염비브리오는 780-1,700 MPN/100 g (평균 1,193 MPN/100 g) 이였으며, 8월 시료에서는 1,700-2,100MPN/100 g (평균 1,967 MPN/100 g)로 측정되었다. 8월 바지락이 7월 바지락보다 평균 약 1.8배 정도의 높은 농도의 장염비브리오가 검출되었는데 이는 수온상승에 의하여 해수 중의 장염비브리오의 농도가 높았기 때문인 것으로 판단된다. 동일시점의 해수와 바지락에 존재하는 장염비브리오의 농도를 비교해 보면 7월 시료의 경우, 바지락이 해수보다 약 2.5배 정도 장염비브리오의 농도가 높았으며, 8월 시료에서도 바지락이 해수보다 약 2.45배 정도 높은 것으로 확인되었다. 결과적으로 해수보다는 바지락에 약 2.5배 정도 높은 농도로 장염비브리오가 존재 하는데 이는 바지락의 먹이섭취 방식인 여과섭식 때문에 바지락 체내에 장염비브리오의 축적이 용이하였거나 또는 바지락의 생활환경인 저질이 해수보다 영양성분이 많아 장염비브리오 증식에 유리하게 작용하였을 가능성이 제기된다. DePaola et al. (1990)는 동일 장소에서 채취한 굴 중의 장염비브리오 농도가 해수보다 100배 높았다는 보고가 있으나 본 연구의 결과와는 매우 다른 결과이다. 따라서 수온이 상승하는 하절기에는 해수 및 수산물에 높은 농도로 장염비브리오가 존재하고 있다는 점에서 장염비브리오 유래 식중독 사고의 예방을 위하여 지속적인 모니터링과 적절한 예방대책이 요구된다.
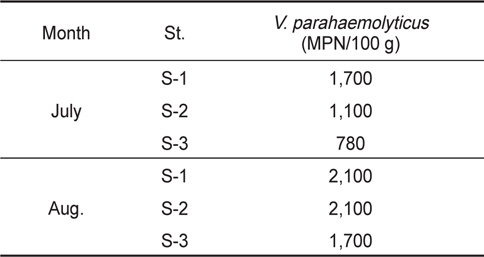
Levels of Vibrio parahaemolytcius in short-necked clam Ruditapes philippinarum in Gomso Bay from July to August 2013