Whole genome transcriptomes from
전사요소(transcription factor; TF)는 특이한 염기서열을 가지고 있는 DNA 결합 단백질로 특정 유전자가 mRNA로 전사되는 과정중 유전자의 프로모터 부위에서 작용을 촉진 또는 방해함으로써 식물체의 발달 또는 여러 환경요인에 대한 반응을 조절하는 작용을 하는 것으로 알려져 있다(Chen et al., 2012; Latchman, 1997). 진핵생물의 경우 전사요소들은 전체 게놈의 약 7%를 차지하는 것으로 알려져 있으며 하나의 전사요소들은 유사 또는 특이한 기능을 가진 여러 개의 유전자 군(gene family)으로 존재하고 있으며, 진화과정에서 여러 생물종 특히 식물종에서 잘 보존되어 있는 것으로 보고되고 있다(Wang et al., 2010).
이제까지 알려진 여러 전사요소들은 각각 특이성 있는 기능을 보이고 있는 것으로 알려졌다. 전사요소 bZip는 생물학적 및 비생물학적 신호작용 또는 발달단계에 관여하는데 특히 ABA (abscisic acid)에 반응하는 유전자들의 전사작용을 촉진하는 것으로 보고 되었다(Jakoby et al., 2002). 또 다른 커다란 식물 전사요소 유전자군에 속하는 Myb은 UV 광, 상처, 혐기성 스트레스, 생물 및 비생물적 스트레스에 반응하는 것으로 보고되고 있다(Cai et al., 2011; Dubos et al., 2010). WRKY 전사요소군 역시 식물체 전사조절에서 매우 독특한 기능을 가지고 있는 것으로 보고되고 있다(Chen et al., 2012). 특히 전사조절 유전자들 중에는 식물이 생육 적온보다 낮은 온도에서의 냉해 또는 빙점에 가까운 온도에서 동해피해를 대비하여 식물의 생리적 및 대사적 조절작용에 관여하는 것들이 다수 알려져 있다(Chen et al., 2012; Tang et al., 2005). 이들은 공통적으로 생육 적온 범위를 벗어나 냉해 또는 동해를 유발할 수 있는 환경조건에서 그 발현량이 증가되어 하부 조절작용에 관여하는 여러 유전자들과 결합하여 그 기능을 향상 또는 억제시킴으로써 식물체의 적응력을 높이는 작용을 한다(Gao et al., 2002; Qin et al., 2004).
난지형 잔디는 27-35°C의 온도 범위에서 원활한 생육을 보이는 것으로 알려졌지만 종 또는 품종간에도 온도에 대한 적응력 차이를 보이고 있고, 특히 St. Augustinegrass는 저온내성이 아주 낮고, seashore paspalum과 bermudagrass가 중간정도, 그리고 상대적으로 한국 들잔디가 높은 것으로 알려졌다(Duncan and Carrow, 2000). 하지만 이들 난지형 잔디에서 저온 반응성과 관련한 유전정보는 다른 식량작물들에 비하여 미약한 편이고 최근의 차세대 염기서열 정보를 활용한다면 실제 유전체 정보가 상대적으로 미흡한 잔디에서 유용한 유전정보를 활용할 수 있을 것으로 기대된다(Duncan and Carrow, 1999). 실제로 최근의 비교유전체 연구를 통해서 유전체 구조가 복잡하거나 유전자 기능이 잘 알려져 있지 않은 잔디에서 기존 모델식물에서 밝혀진 저온내성 유전자 종류, 전사조절 기작 및 유전자 기능들을 활용하는 사례는 많이 보고되고 있다(Takuhara et al, 2011; Thomashow, 2010).
난지형 잔디와 같이 저온 휴면형 식물체는 가을철 온도하강과 함께 잎의 노화가 일어나고 녹기연장형(stay-green type)은 노화의 시작, 노화속도의 지연, 그리고 엽록소 분해정도에 따라서 4가지 유형으로 분류되고 있다(Kusaba et al., 2013). 식물체가 저온에 노출되었을 때 이러한 노화 또는 엽록소의 변화는 많은 유전자의 발현차이와 이들 유전자 조절 전사요소들이 관여하는 것으로 나타났다. 식물 발달단계에 따라 발현하는 전사요소 중에는 식물체의 노화시기를 결정하는데 관여하고 있어 이들 전사조절 유전자의 기능이 상실됨에 따라 식물체 잎의 녹색연장이 가능하다는 것을 실험적으로 증명하기도 하였다(Kusaba et al., 2013).
본 연구의 목적은 억새 전사체 분석을 통하여 얻어진 전사조절 유전자 중에서 저온에 관여하는 유전자를 분리하고, 다른 작물에서의 기능을 근거로 저온에서 발현이 증가하는 유전자를 중심으로 난지형 잔디에서의 저온 반응성을 비교하는 것이다. 이를 위해서 먼저 타겟 전사조절 유전자의 유전정보를 확보하고 다른 작물과의 상동성을 비교하여 특성을 검증할 것이다. 또한 식물 종간에 비교적 잘 보존된 DNA 영역에서 프라이머 디자인을 한 뒤 저온 반응성이 다른 난지형 잔디를 저온에 노출시켜 시간에 따라 저온 반응 유전자의 변화를 조사하여 저온 적응성, 내한성 그리고 녹기지연 특성간의 관련성을 알아보고자 한다.
본 연구에 이용된 식물체는 충남대학교에서 수집 또는 분양받아 보관중인 난지형 잔디 4종 [한국 금잔디 교배종 ‘세밀’(단국대 분양품종,
본 연구에 이용된 전사요소는 잔디와 같이 화본과 작물의 하나인 억새 EST (Expressed Sequence Tag)에서 유래하였다(Kim et al., 2013). 참억새 (
GS-FLX 염기서열 분석장치를 활용한 NGS 분석을 통해 확보한 억새 잎과 지하경의 EST 데이터는 데이터베이스 검색(http://www.phytozome.net/)을 통해 애기장대와 벼에서 밝혀진 유전자를 기초로 억새 EST 염기서열에 대한 annotation을 수행하였다. 억새 전사조절 요소 분석은 Jin et al. (2013)이 개발한 Plant Transcription Factor Database v3.0 (http://planttfdb.cbi.pku.edu.cn/)에 있는 데이터 베이스를 기초로 수행하였다.
억새 전사요소를 분석한 결과 WRKY family가 참억새와 물억새 잎과 줄기에서 발현되는 모든 종류 중 가장 높은 빈도로 출현(9.59%)하였고, 저온 스트레스에서 식물의 방어기작에 관여한다는 사실을 근거로 본 연구에서는 여러 난지형 잔디의 저온 반응의 차이를 알아보기 위하여 WRKY 전사요소를 대상으로 분석하게 되었다.
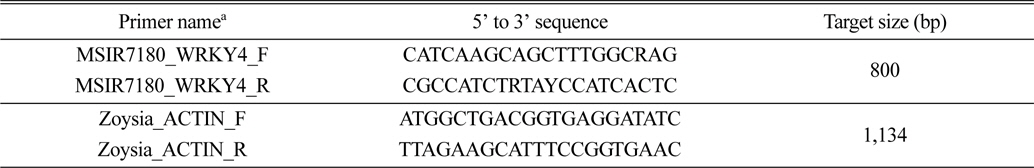
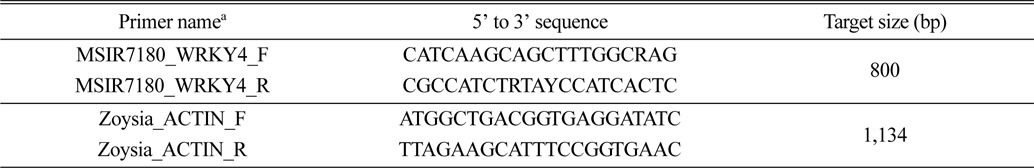
잔디 WRKY 유전자의 염기서열은 억새 transcriptiom 분석을 통해 얻어진 EST 데이터로부터 plant TF DB(http://planttfdb.cbi.pku.edu.cn/)에 있는 데이터를 기초로 약 230여개의 WRKY family와 상동성을 보이는 isotig로부터 확보하였다. 그 중 약 80여개의 isotig가 저온에 반응하는 WRKY family와 높은 상동성을 보이는 것으로 확인되었다. 80여개의 억새 isotig에서 얻어진 염기서열 정보들은 BLASTN을 이용하여 NCBI database에서 검색된 자료들과 비교 분석하였고 ClustalW2 (http://www.ebi.ac.uk/Tools/clustalw2)를 이용하여 여러 식물체에서 공통적으로 나타나는 염기서열을 기초로 degenerate primer를 디자인하였다(Table 3).
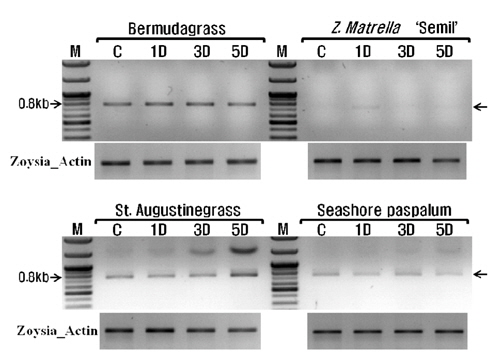
A degenerated primer used for expression analysis of warm-season turfgrass WRKY gene, which was designed from the conserved region of the reported WRKY4 sequences of some plant species including Miscanthus EST. The zoysiagrass Actin gene was used as a control.
NCBI database에서 확보한 여러 식물체의 WRKY family 유사도 분석과 phylogenetic tree는 BioEdit 소프트웨어(V.7.0.9.0, http://www.mbio.ncsu.edu/BioEdit/bioedit)와 Molecular Evolutionary Genetics Analysis (MEGA) 소프트웨어(v.5.2, http://www.megasoftware.net) (Tamura et al., 2011)를 이용하여 neighbor-joining methods으로 작성하였다(Saitou and Nei, 1987).
저온(10oC)에서 잔디 WRKY 전사요소의 발현양상을 알아보기 위하여 4종의 잔디 잎(100 mg)으로부터 Hybrid R(Geneall, Seoul, Korea)를 이용하여 총 RNA를 추출하였다. 추출된 RNA는 흡광도계를 이용하여 정량하여 냉동고(-80°C)에 보관하여 사용하였다. RT (reverse transcription) PCR은 Takara RNA PCR Kit (AMV) Ver. 3.0 (Takara, Shiga, Japan)를 이용하여 제조사 사용방법을 따라 실시하였고, NCBI에 공개되어 있는 한국잔디(Zoysia japonica) beta-actin (GU290545, 1134bp)을 기준으로 참억새와 물억새 잎 또는 지하부 조직으로부터 탐색된 저온반응 WRKY전사요소(MSAL314_WRKY33, MSAL1702_WRKY33, MSIR7180_WRKY4)의 상대적인 발현차이를 조사하였다(Table 3). 발현량을 조사하기 위한 들잔디 Actin 유전자의 프라이머는 Actin_F: 5’-ATG GCT GAC GGT GAG GAT ATC-3’, Actin_R: 5-TTA GAA GCA TTT CCG GTG AAC-3으로 디자인하였다. 잔디 Actin 유전자의 증폭은 우선 94°C에서 5분간 DNA 가닥의 변성, 이어 25회에 걸쳐 변성(94°C에서 30초)-결합(53°C에서 30초)-신장(72°C에서 1분 10초)을 반복한 뒤 마지막으로 72°C에서 7분간 안정화 과정을 거쳐 중합반응을 완료했다. 잔디의 저온반응성을 알아보기 위하여 사용된 MSIR7180_WRKY4 전사요소의 연쇄중합반응은 94에서 5분간 DNA 가닥의 변성, 이어 25회에 걸쳐 변성(94°C에서 30초)-결합(55°C에서 30초)-신장(72°C에서 1분)을 반복한 뒤 마지막으로 72°C에서 7분간 안정화시켜 완료했다.
전사요소는 유전자의 전사작용, 발생의 조절, 세포간 신호전달, 환경에 대한 식물의 반응, 그리고 세포주기의 조절에 필요한 유전자의 발현을 조절하는 중요한 기능을 수행한다(Chen et al., 2012; Latchman, 1997). 따라서 식물의 발달 단계 또는 환경의 변화는 이를 감지하는 신호로 변환되어 하부 관련 유전자의 작용으로 이어지는데 전사조절 요소들은 이 과정에서 전사 개시 전과 후에 타겟 유전자와 결합하여 유전자 발현을 유도(induction) 또는 억제(repression)하게 된다. 본 연구에서는 억새의 잎과 지하경 조직으로부터 확보한 4개의 억새 EST 라이브러리로부터 유전자 전사요소 전체를 탐색하여 그 중 생육 적온보다 낮은 저온에 반응하는 전사조절 요소를 조사하고 난지형 잔디에서 반응차이를 알아보는 것을 목적으로 하였다.
난지형 잔디는 많은 유용한 특성을 가지고 있어 우리나라에서는 매우 중요한 지피 또는 조경소재로 이용되고 있다. 특히 골프장과 같은 대규모 이용지역에서는 이른 봄철 지온 상승에 따른 지상부의 출현 시기와 기온의 하강에 따른 지상부의 휴면돌입 시기가 잔디의 활용빈도에 직접 관련이 있기 때문에 최근 녹색기간이 연장된 새로운 품종에 대한 요구가 높아지고 있다(Chang and Kim, 1986). 휴면의 돌입은 결국 지상부 잎의 노화와 관련이 있고 이에 따라 최근 분자수준에서 잎의 녹색지속 식물형을 Type A에서 Type E까지 5개 그룹으로 분류하여 보고하고 있다(Kusaba et al., 2013).
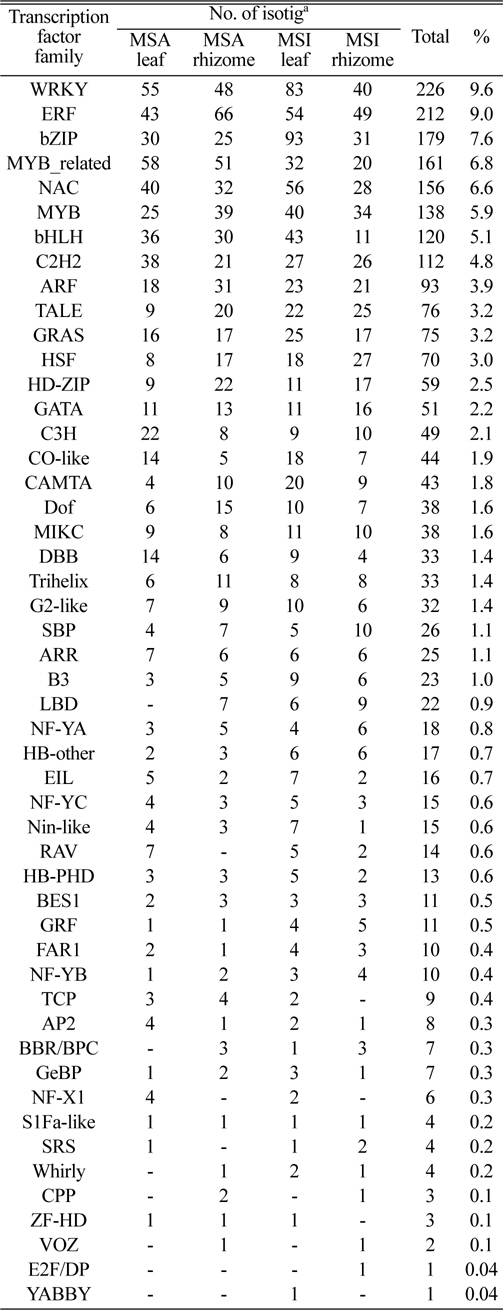
분석결과 탐색된 전사조절 요소의 종류는 총 50 종류로 나타났고, 그 중 WRKY family 전사요소와 관련 있는 isotig가 226개로 가장 많이 발견되었고(9.6%), 다음으로 ERF 전사요소와 관련 있는 isotig가 212개(9.0%), bZIP 전사요소와 관련 있는 isotig가 179개(7.6%), Myb-related 전사요소와 관련 있는 isotig가 161개(6.8%), 그리고 NAC 전사요소와 관련 있는 isotig가 156개(6.6%)순으로 상위 5종류에 포함되었다(Table 1).
[Table 1.] List of the identified transcription factors from two Miscanthus species.
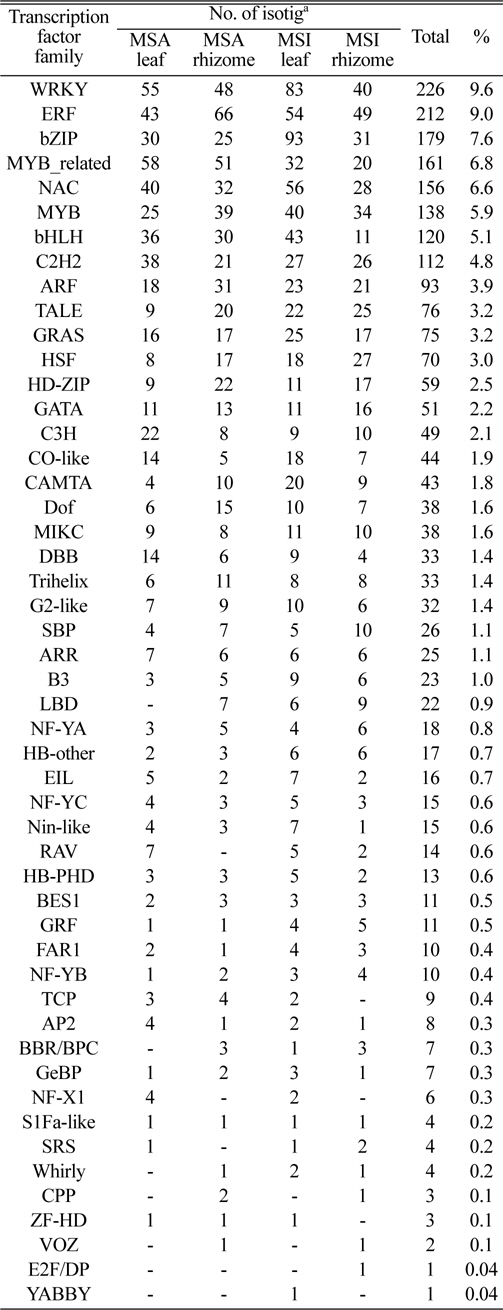
List of the identified transcription factors from two Miscanthus species.
억새 종류 및 조직별 전사요소의 분포를 보면 물억새는 잎에서 MYB_related 유전자가 가장 많았고(10.6%) WRKY(10.1%)와 ERF(7.9%) 유전자가 그 다음 순으로 EST isotig와 관련 되었고, 지하경의 경우 ERF(11.5%), MYB_related(8.9%), 그리고 WRKY(8.3%) 순으로 분포하였다. 참억새 잎의 경우는 bZIP(12.7%) 유전자가 가장 많았고 WRKY(11.4%), NAC(7.7%)순으로 분포하였으며, 지하경에서는 ERF(9.7%), WRKY(7.9%) 그리고 MYB(6.7%)순으로 높게 나타났다. 억새의 조직별로는 CPP와 VOZ 유전자는 지하경에서만 관찰되었으며, NF-X1은 반대로 잎에서만 관찰되었다. 억새의 종류에 따라 E2F/DP는 참억새의 지하경에서, YABBY는 참억새의 잎에서만 관찰되는 것을 알 수 있었고, 물억새 잎과 지하경에서만 관찰되는 유전자는 없는 것으로 나타났다. 이러한 사실로 미루어 볼 때 전사요소는 식물 종 또는 조직 특이적으로 유전자 발현 조절기능이 다르다는 것을 알 수 있었다. 기존의 발표결과에서도 동일한 전사조절 유전자(예, WRKY, CBF) family가 식물 종에 따라 1~197개까지 다양하게 발견된다고 하였고, 잎, 줄기, 종자 또는 꽃 등 발현되는 조직도 달랐으며, 저온 스트레스를 받았을 때 조직 별로 발현차이가 있다는 사실이 보고되고 있다(Rushton et al., 2010; Takuhara et al., 2011).
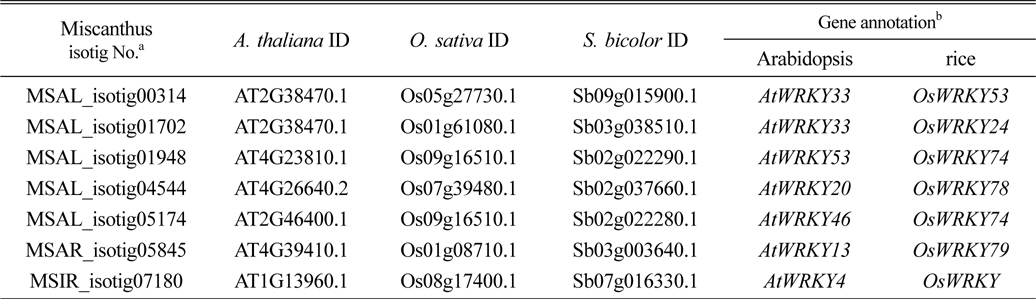
발견된 전사조절 유전자 중에서 제일 많이 발견된 것은 WRKY 전사요소였고, 따라서 본 연구에서는 억새 WRKY family를 애기장대, 벼 그리고 수수에서 기능이 밝혀진 WRKY 유전자들과 비교 검색한 결과 약 80개의 억새 isotig가 저온에 반응하여 발현이 유도 또는 억제되는 것으로 나타났고, 이들은 또한 저온 또는 다른 비생물적 스트레스 환경에서 자란 타 작물의 WRKY 유전자들과 매우 높은 상동성을 보이는 것을 확인할 수 있었다(Rushton et al., 2010). 탐색된 80개의 억새 isotig 중 염기서열 길이가 가장 길고 상동성이 가장 높은 것(e-value 기준)으로 확인된 7개의 억새 isotig를 얻을 수 있었고 애기장대와 벼에서 저온에 대한 반응성을 기초로 상동성이 높은 유전자를 찾을 수 있었다(Table 2). 애기장대의 경우
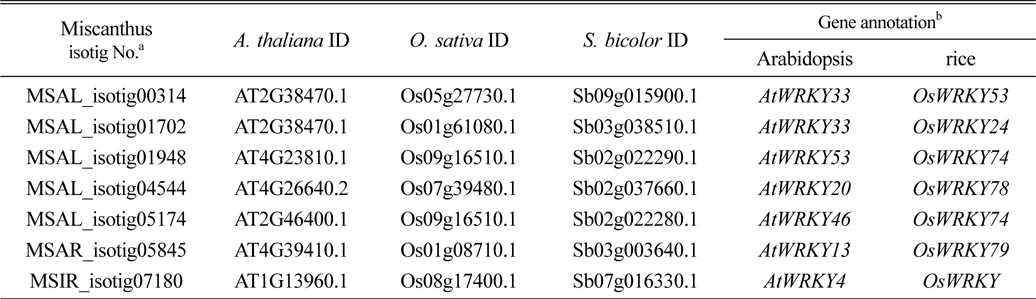
Miscanthus WRKY transcription factors responding to low temperature and their corresponding homologous genes from Arabidopsis, rice and sorghum.
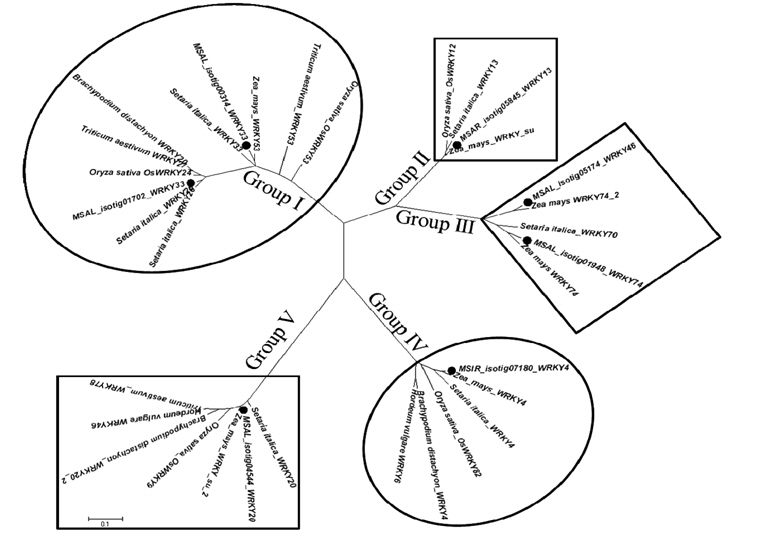
계속해서 이들 7개 isotig들의 염기서열 정보들을 BLASTN을 이용하여 NCBI database에 등록된 다른 유전자들의 염기서열과 비교하였고 여기서 확보한 염기서열들은 ClustalW2(http://www.ebi.ac.uk/Tools/clustalw2)를 이용하여 여러 식물체(
난지형 잔디의 저온 반응성을 비교하기 위하여 무가온 온실에서 자란 4가지 난지형 잔디의
본 연구에 사용한 네가지 난지형 잔디 모두 저온에 대한 내성은 다른 한지형 또는 난지형 잔디에 비하여 상대적으로 낮으나, 본 연구 결과만으로는 저온에 대한 내성을 판단하기에는 무리가 있어 보인다. 하지만 한국잔디류중에서 ‘세밀’과 같은 금잔디가 가을 철 녹색기간이 비교적 오래 유지되는 것은